Les phages au secours des infections multirésistantes ?
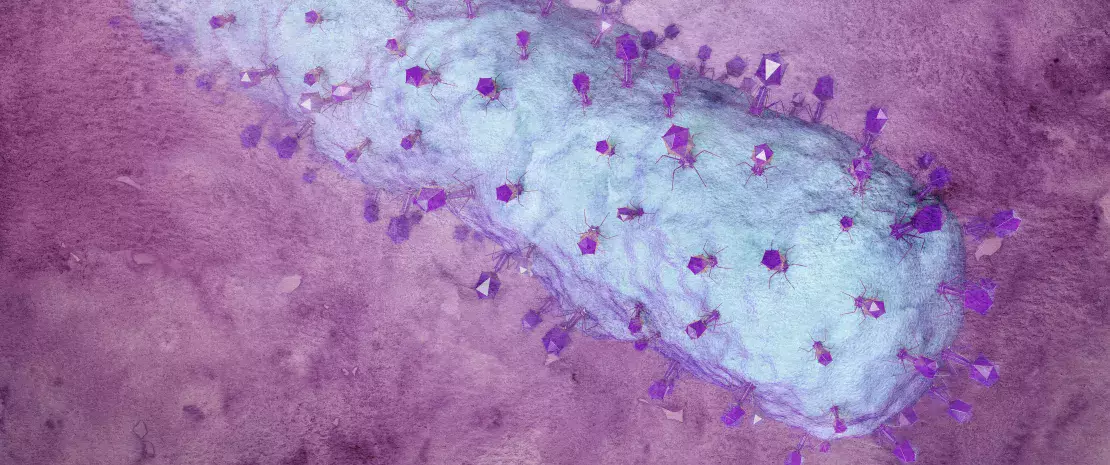
Avec l’émergence de l’antibiorésistance, les bactériophages font leur grand retour. Ces virus du microbiote intestinal pourraient à terme traiter, entre autres de façon plus ciblée les infections multirésistantes. Un article de Nature Reviews Gastroenterology & Hepatology fait le point sur l’histoire, l’avenir et les défis de la phagothérapie.
en_sources_title
en_sources_text_start en_sources_text_end
A propos de cet article
Les bactériophages ou « phages » sont les entités biologiques les plus abondantes et variées sur terre. Prédateurs naturels des bactéries, ils sont ubiquitaires dans le sol, les océans… et le microbiote intestinal humain, où ils constituent le type de virus dominant. Des dysbioses bactériennes intestinales associées à des pathologies gastrointestinales comme la maladie de Crohn ou le syndrome de l’intestin irritable s’accompagnent de modifications dans la composition de ces virus.
Fer de lance de l'arsenal thérapeutique moderne, les antibiotiques ont sauvé des millions de vie. En revanche, leur utilisation excessive et parfois injustifiée peut conduire à l'apparition de différentes formes de résistance chez les micro-organismes. Chaque année, l'Organisation Mondiale de la Santé (OMS) organise la Semaine mondiale de sensibilisation à la résistance aux antimicrobiens (WAAW) afin de sensibiliser la population sur ce problème de santé publique. Lisez la page qui y est consacrée.
Résistance aux antibiotiques : le microbiote au premier plan
Cent ans plus tard, un retour sous le feu des projecteurs
Dès les années 20, des expérimentations menées sur les phages pour évaluer leur potentiel de traitement ont donné quelques résultats prometteurs chez des patients atteints de shigellose, de dysenterie et de choléra. Cette (sidenote: Summers WC. The strange history of phage therapy. Bacteriophage. 2012 Apr 1;2(2):130-133. ) a été balayée par l’arrivée des antibiotiques dans les années 40. Si quelques essais, hélas mal documentés, se sont poursuivis dans des pays de l’Est, les phages ont été relégués au second plan. Mais les récentes inquiétudes concernant les infections multirésistantes, tout comme l’accumulation des connaissances sur l’impact des antibiotiques sur l’équilibre du microbiote intestinal, ont motivé la reprise des études sur la phagothérapie. Chaque espèce de phage ayant généralement pour cible une seule espèce bactérienne, ces virus pourraient apporter une solution « de précision » là où les antibiotiques à spectre large posent question. Cependant, même s’ils suscitent de grands espoirs, les phages ne font pas encore l’objet d’un traitement autorisé par les autorités de santé (sauf pour de très rares exceptions).
Qu'est-ce que la Semaine mondiale de sensibilisation à la résistance aux antimicrobiens ?
Depuis 2015, l'OMS organise chaque année la Semaine mondiale de sensibilisation à la résistance aux antimicrobiens (WAAW) dont l'objectif est de sensibiliser sur le phénomène mondial de la résistance aux antimicrobiens. Cette campagne, qui se tiendra du 18 au 24 novembre, encourage le grand public, les professionnels de santé et les décideurs à faire un bon usage des antimicrobiens afin d'éviter l'apparition de résistance.
Antibiorésistance, dysbioses, ciblage thérapeutique… : de multiples potentiels
(sidenote: Schooley RT, Biswas B, Gill JJ, et al. Development and Use of Personalized Bacteriophage-Based Therapeutic Cocktails To Treat a Patient with a Disseminated Resistant Acinetobacter baumannii Infection. Antimicrob Agents Chemother. 2017 Sep 22;61(10):e00954-17. ) a attiré l’attention : un patient diabétique de 68 ans, infecté par une bactérie multi résistante (Acinetobacter baumannii) et souffrant d’une pancréatite a recouvré la santé en 5 mois grâce à la phagothérapie, après plusieurs échecs de traitements par antibiotiques. D’autres succès ont été rapportés dans des infections par (sidenote: Jennes S, Merabishvili M, Soentjens P, et al. Use of bacteriophages in the treatment of colistin-only-sensitive Pseudomonas aeruginosa septicaemia in a patient with acute kidney injury-a case report. Crit Care. 2017 Jun 4;21(1):129. ) et (sidenote: Dedrick RM, Guerrero-Bustamante CA, Garlena RA, et al. Engineered bacteriophages for treatment of a patient with a disseminated drug-resistant Mycobacterium abscessus. Nat Med. 2019 May;25(5):730-733. ) , avec l’espoir d’alternatives prometteuses pour le traitement des infections bactériennes multirésistantes. La phagothérapie comme modulateur du microbiote intestinal intéresse également les scientifiques. Une étude sur la souris a montré qu’un traitement par phages ciblant Enterococcus faecalis, bactérie associée à un mauvais pronostic dans l’hépatite alcoolique, pouvait améliorer la maladie.
D’autres utilisations potentielles des phages sont désormais évoquées, notamment pour la médecine de précision. Ces phages permettent le transport d’anticancéreux ou d’antibiotiques puissants vers une zone précise du corps, permettant ainsi d’augmenter les doses et de diminuer la toxicité du traitement sur les tissus voisins.
A la recherche de réponses aux défis d’utilisation clinique
La recherche doit désormais répondre aux nombreuses questions que pose la pratique clinique. La phagothérapie est-elle toujours sûre ? Peut-elle remplacer un traitement antibiotique ? Quels sont le bon mode d’administration et le bon dosage ? Quel est leur effet à long terme sur le microbiote et la santé en général ? Selon les auteurs, des essais cliniques randomisés en double-aveugle et contrôlés contre placebo sont nécessaires pour donner une place légitime à la phagothérapie, une pratique séculaire qui pourrait répondre à de nombreux enjeux de la médecine d’aujourd’hui.
Nous vous présentons le Professeur Sørensen, lauréat de la bourse internationale 2022 de la Biocodex Microbiota Foundation.
Son équipe a été la première à lancer une étude ambitieuse sur le résistome de 700 enfants, qui permettra de faire un pas de géant dans la compréhension de l'évolution et de la dissémination de la résistance aux antibiotiques dans l'intestin humain au cours de la petite enfance.