Vers un indice d’espèces clés associé à la santé pour le microbiote intestinal humain
ARTICLE COMMENTE - Rubrique adulte
Par le Pr. Harry Sokol
Gastro-entérologie et nutrition,Hôpital Saint-Antoine, Paris, France
Commentaire de l’article de Goel et al., Cell Reports 2025 1
Section grand public
Retrouvez ici votre espace dédié
en_sources_title
en_sources_text_start en_sources_text_end
Chapitres

A propos de cet article
Un indice robuste des taxons du microbiome intestinal, intégrant leur association avec la santé de l’hôte et la résilience du microbiome, serait précieux pour le développement et l’optimisation de thérapeutiques fondées sur le microbiome. Les auteurs présentent ici un classement unique de 201 taxons, l’indice Health-Associated Core Keystone (HACK), élaboré à partir de leur prévalence/association communautaire chez des sujets non malades, leur stabilité temporelle et leur association avec la santé de l’hôte. Cet indice a été construit à partir de 127 cohortes de découverte et de 14 jeux de données de validation (regroupant au total 45 424 microbiomes intestinaux provenant de sujets de plus de 18 ans, couvrant 42 pays, 28 catégories de maladies et 10 021 échantillons longitudinaux). Les auteurs montrent que cet indice est reproductible, quels que soient les stratégies d’analyse du microbiome et les modes de vie des cohortes. Certains consortiums de taxons à indice HACK élevé répondent positivement aux interventions basées sur le régime méditerranéen, sont associés à une meilleure réponse aux inhibiteurs de points de contrôle immunitaire et à des profils fonctionnels spécifiques au niveau génomique. La disponibilité des indices HACK offre ainsi une base rationnelle pour comparer les microbiomes et faciliter la sélection et la conception de thérapies basées sur le microbiome.
Que sait-on déjà à ce sujet ?
Les thérapeutiques basées sur le microbiome intestinal (probiotiques, biothérapeutiques vivants, pré-/synbiotiques, transplantation fécale) visent à rétablir un microbiote sain mais affichent des succès variables selon les populations. Pour optimiser ces approches, il faudrait disposer d’une définition consensuelle d’un microbiome « sain », tâche ardue en raison de la grande variabilité interindividuelle. Des méta-analyses révèlent toutefois des taxons systématiquement appauvris ou enrichis dans de multiples maladies, suggérant que l’on peut positionner les microbes le long d’un continuum de lien avec la santé de l’hôte 2, 3. Les espèces occupant le haut de ce classement présenteraient le plus grand potentiel : i) comme agents thérapeutiques directs ou cibles à enrichir ; ii) comme marqueurs d’efficacité clinique. Les auteurs proposent donc de créer un indice de priorité intégrant trois critères : association positive avec la santé, contribution à la stabilité du microbiote et forte « interaction » communautaire. Exploitable sur de vastes jeux de données publiques, cet indice servirait à sélectionner et à évaluer rationnellement les futures stratégies thérapeutiques microbiennes.
Quels sont les principaux résultats apportés par cette étude ?
À partir d’une cohorte de découverte comprenant 39 926 microbiomes intestinaux provenant de 127 cohortes (données transversales et longitudinales couvrant 42 pays et 28 pathologies différentes), les auteurs ont élaboré un classement de 201 taxons prévalents du microbiote intestinal (détectés dans ≥ 5 % des échantillon dans ≥ 50 % des cohortes étudiées), l’« indice HACK » (Health-Associated
Core Keystone Index), en leur attribuant un score reposant sur trois propriétés quantifiables : i) prévalence/association communautaire chez des sujets non malades ; ii) stabilité temporelle ; et iii) association négative avec la maladie.
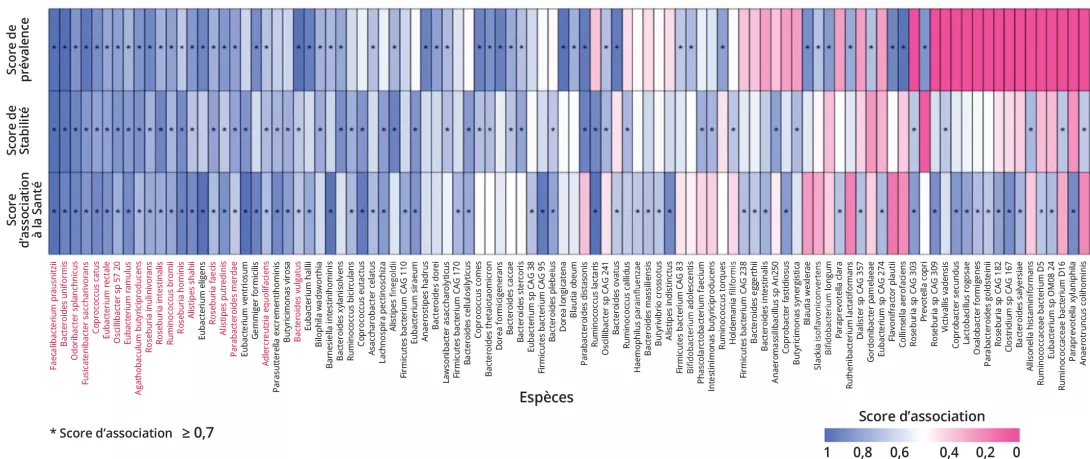
L’indice HACK a été calculé comme le produit de deux scores : i) la moyenne des scores d’association d’un taxon pour chacune des trois propriétés ; et ii) un score de récompense qui évalue la similarité (ou la répartition équilibrée) de ces trois scores entre eux. L’analyse des taxons les mieux classés selon cet ordre a révélé 17 taxons dont l’indice HACK est ≥ 75 % (figure 1). Ces taxons affichaient tous des scores ≥ 70 % pour les trois propriétés. Ils comprennent Faecalibacterium prausnitzii, un marqueur bien connu de la santé du microbiome4, suivi de Bacteroides uniformis. Figurent également dans cette liste plusieurs espèces des genres Roseburia, Alistipes et Eubacterium, ainsi que Coprococcus catus.
Les auteurs ont ensuite démontré la reproductibilité des scores individuels et de l’indice HACK dans son ensemble en recalculant les scores d’association individuellement dans chaque cohorte, en utilisant différentes méthodes de séquençage (Shotgun ou 16S) et différents types de populations (urbaines industrialisées vs autres), puis dans un jeu de validation supplémentaire composé de 14 cohortes supplémentaires totalisant 5 498 microbiomes.
Au-delà de leur lien plus étroit avec la santé et la stabilité du microbiote, certains taxons présentant un indice HACK élevé sont associés à une réponse favorable à diverses interventions thérapeutiques liées au microbiote, telles que le régime méditerranéen ou l’immunothérapie anticancéreuse.
En analysant les annotations fonctionnelles à l’échelle du génome obtenues à partir de 32 005 génomes couvrant 122 des 201 taxons, les auteurs ont identifié 150 familles de fonctions spécifiquement enrichies et conservées dans les génomes des taxons présentant un indice HACK élevé. Cela représente un vaste éventail de fonctions : production de butyrate/propionate aux propriétés antiinflammatoires, synthèse de nombreuses vitamines, biosynthèse d’acides aminés neuro-actifs comme le tryptophane, ainsi que de leurs dérivés anti-inflammatoires bénéfiques tels que les indoles, ou encore les chondroïtine-sulfates. Ces fonctions pourront être explorées pour comprendre les mécanismes sous-jacents.
Quelles sont les conséquences en pratique ?
Les indices HACK ont été calculés à partir d’une cohorte mondiale de 45 000 microbiomes intestinaux couvrant les six grands continents, dans l’une des études les plus exhaustives à ce jour. Ces indices représentent une avancée vers la priorisation rationnelle des espèces microbiennes intestinales comme potentielles thérapeutiques fondées sur le microbiome. En outre, les fonctionnalités associées aux indices HACK pourraient aider à identifier les voies et capacités métaboliques liées à la santé générale et à la stabilité du microbiome.
- À partir de 45 454 microbiomes issus de 141 cohortes (42 pays, 28 groupes de maladies), cette étude a classé 201 taxons selon leurs modes d’association avec trois traits essentiels de la santé de l’hôte et du microbiome : i) prévalence chez des sujets non malades ; ii) stabilité temporelle ; et iii) association négative avec la maladie
- Parmi les 17 bacteries avec les scores les plus élevés, on rerouve Faecalibacterium prausnitzii et Bacteroides uniformis en première et deuxièmes positions
- Le classement était reproductible quels que soient le type de séquençage et le mode de vie des cohortes
- Les taxons les mieux classés sont liés à des réponses positives à différentes interventions thérapeutiques liées au microbiote
Conclusion
En partant d’une très grande base de données, cette étude permet d’identifier un groupe de 17 taxons particulièrement prévalents, stables au cours du temps et associés à la santé. En plus d’avancer vers la définition des composants clés du microbiote humain en termes de taxonomie et de fonctions, ce travail offre une base rationnelle pour la définition de nouvelles thérapies basées sur le microbiote intestinal ou le ciblant.
- Goel A, Shete O, Goswami S, et al. Toward a health-associated core keystone index for the human gut microbiome. Cell Rep 2025 ; 44 : 115378.
- Shanahan F, Ghosh TS, O’Toole PW. The Healthy Microbiome—What Is the Definition of a Healthy Gut Microbiome? Gastroenterology 2021 ; 160 : 483-94.
- Pasolli E, Asnicar F, Manara S, et al. Extensive Unexplored Human Microbiome Diversity Revealed by Over 150,000 Genomes from Metagenomes Spanning Age, Geography, and Lifestyle. Cell 2019 ; 176 : 649-62.
- Martín R, Rios-Covian D, Huillet E, et al. Faecalibacterium: a bacterial genus with promising human health applications. FEMS Microbiol Rev 2023 ; 47.

