Médicaments et microbes : un dialogue bidirectionnel aux implications thérapeutiques
Par le Pr. Emmanuel Montassier
Urgences, soins critiques, anesthésies, réanimation, médecine interne, maladies infectieuses, Université de Nantes, France
Section grand public
Retrouvez ici votre espace dédié
en_sources_title
en_sources_text_start en_sources_text_end
Chapitres

A propos de cet article
Les interactions interactions bidirectionnelles entre les médicaments oraux et le microbiome intestinal sont de plus en plus considérées comme déterminantes dans l’efficacité, la sécurité d’emploi des médicaments et la tolérance à ceux-ci. Si les antibiotiques sont connus pour perturber les communautés microbiennes, environ 24 % des médicaments non antibiotiques inhibent aussi au moins une espèce commensale. De plus, 10 % à 15 % des médicaments oraux sont transformés par les microbes intestinaux in vivo, ce qui affecte leur efficacité et leur toxicité. Les médicaments communs, comme les inhibiteurs de la pompe à protons (IPP), les anti-inflammatoires non stéroïdiens (AINS), la metformine et les statines, peuvent altérer la composition et la fonction du microbiote, ce qui a une influence sur le métabolisme et le système immunitaire de l’hôte. Malgré ces statistiques, le microbiome est souvent ignoré lors de la prescription et de la mise au point de médicaments. Cette étude résume les principales connaissances cliniques et mécanistes, met en évidence les interactions notables entre les médicaments et le microbiote et explore de nouvelles stratégies visant à améliorer les résultats. L’intégration de la pharmacomicrobiomique dans les soins cliniques pourrait réduire les effets indésirables et venir appuyer la médecine de précision.
Le microbiote intestinal joue le rôle d’organe métabolique et participe à la digestion, à l’immunité et à l’homéostasie 1. Ses interactions
avec les médicaments sont toutefois bidirectionnelles : les médicaments peuvent perturber l’équilibre microbien, tandis que les microbes peuvent altérer l’activité médicamenteuse. Le microbiome est donc un facteur important mais souvent ignoré dans le risque de réactions indésirables aux médicaments 2, 3. Les enzymes microbiennes intestinales peuvent augmenter la toxicité des médicaments, ce qui accroît l’exposition des tissus et les effets nocifs. De plus en plus de données probantes montrent que la variabilité microbienne est un facteur clé des différences individuelles en matière de réponses aux médicaments et de réactions indésirables 2, 4. L’intégration de la pharmacomicrobiomique aux évaluations des risques (ainsi que les données génétiques et cliniques) pourrait permettre de prédire la vulnérabilité aux effets nocifs des médicaments et orienter les stratégies de prévention personnalisées.
Perturbation du microbiote induite par les médicaments : antibiotiques et autres
On sait que les antibiotiques perturbent le microbiote intestinal en réduisant sa diversité, en altérant sa composition et en favorisant le développement de souches résistantes (tableau 1) 5, 6. Van Zyl et al. ont montré que les antibiotiques, en particulier les quinolones et les β-lactamines, perturbent systématiquement les communautés microbiennes dans l’ensemble du corps et que les schémas thérapeutiques combinés provoquent une dysbiose prolongée et augmentent la charge pathogène 5. De même, Maier et al. ont montré que les différentes classes d’antibiotiques ont des effets distincts sur les bactéries intestinales : les macrolides et les tétracyclines entraînent des pertes marquées chez les bactéries anaérobies ; quant aux médicaments tels que l’amoxicilline ou la ceftriaxone, ils favorisent le développement des Proteobacteria. En dépit des variations individuelles, une tendance globale se dégage : la disparation des bactéries anaérobies indispensables (ex. : Firmicutes) et la prolifération de microbes non essentiels et potentiellement pathogènes 6.
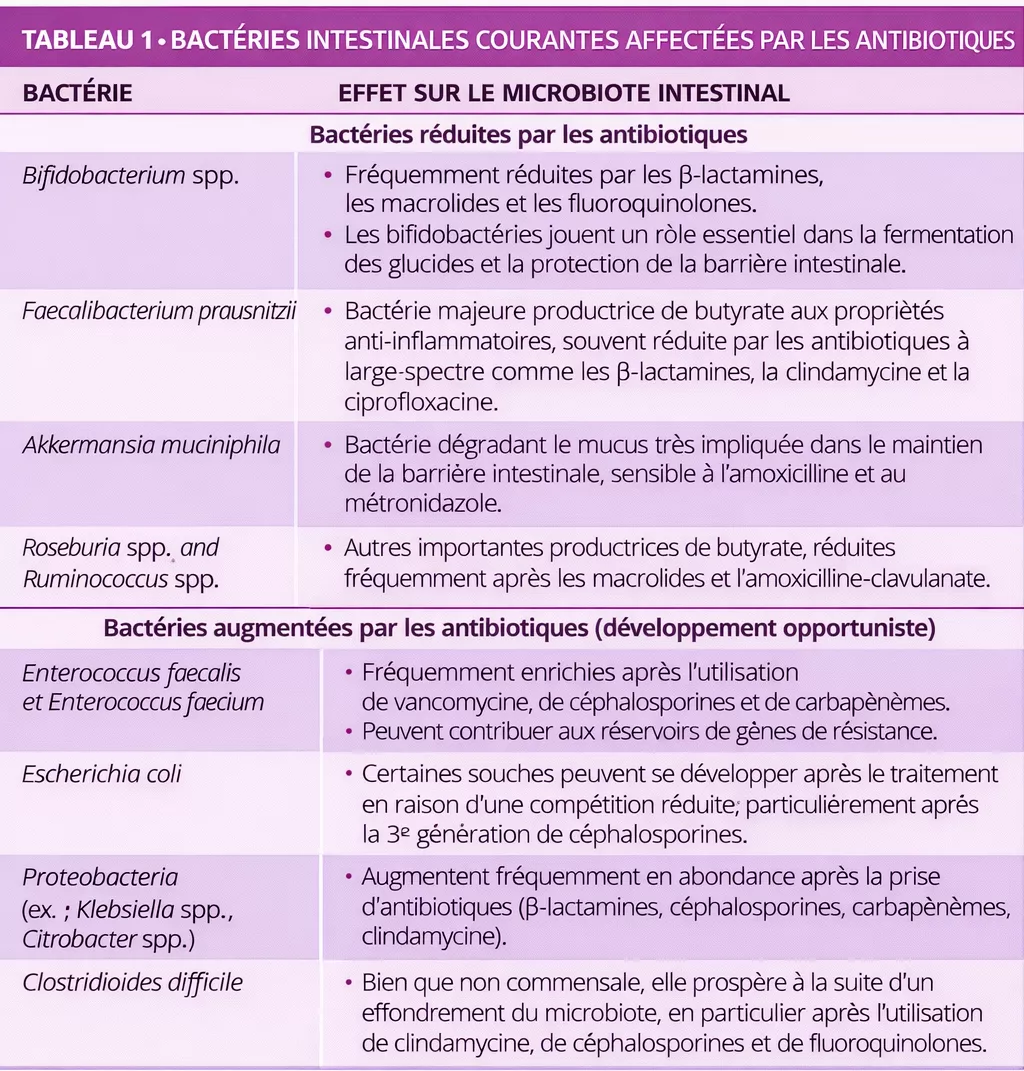
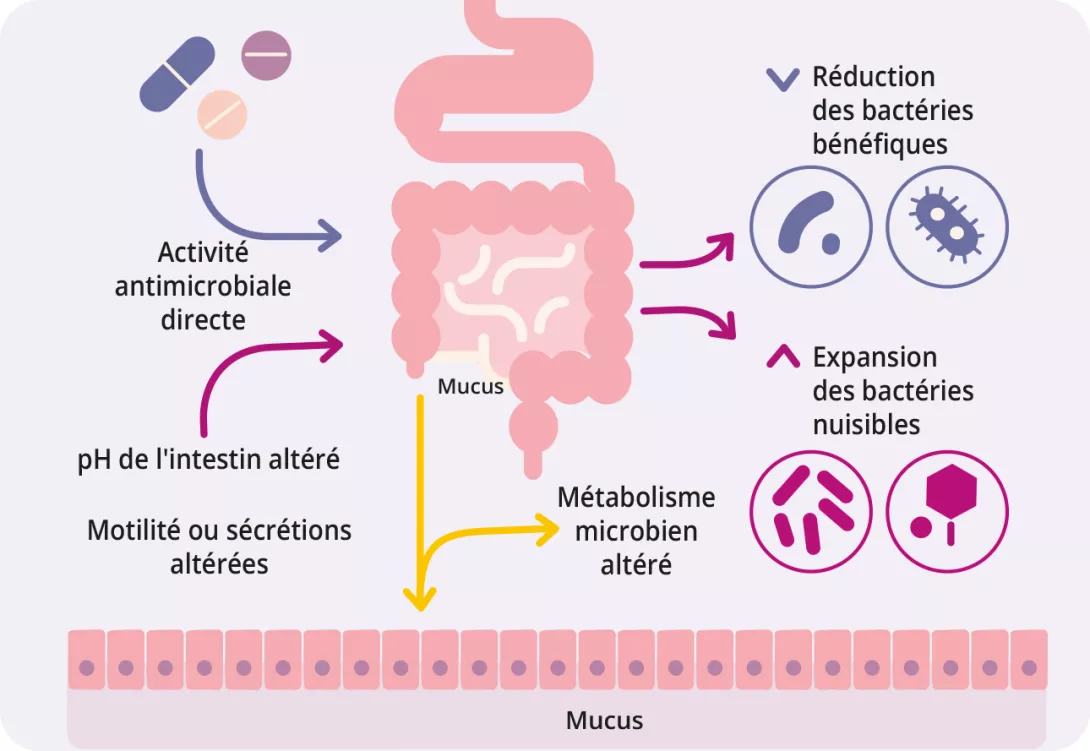
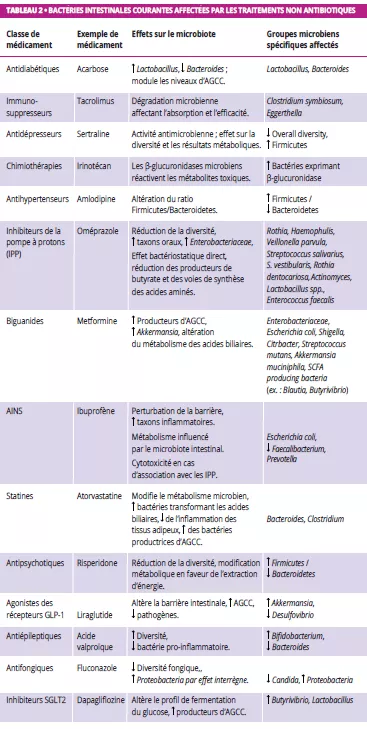
En dehors des antibiotiques, de nombreux médicaments non antibiotiques altèrent également le microbiote intestinal, tel que les IPP, la metformine, les AINS, les antipsychotiques et les statines (figure 1, tableau 2) 7, 8. Les médicaments influencent le microbiote intestinal par divers mécanismes : l’action antimicrobienne directe, l’altération du pH, la modulation des acides biliaires, la modification de la motilité intestinale et la sécrétion de mucus 9.
Le microbiote intestinal modifie le métabolisme
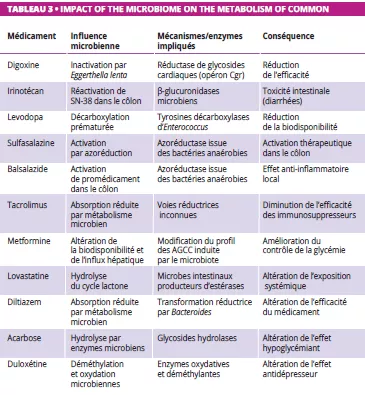
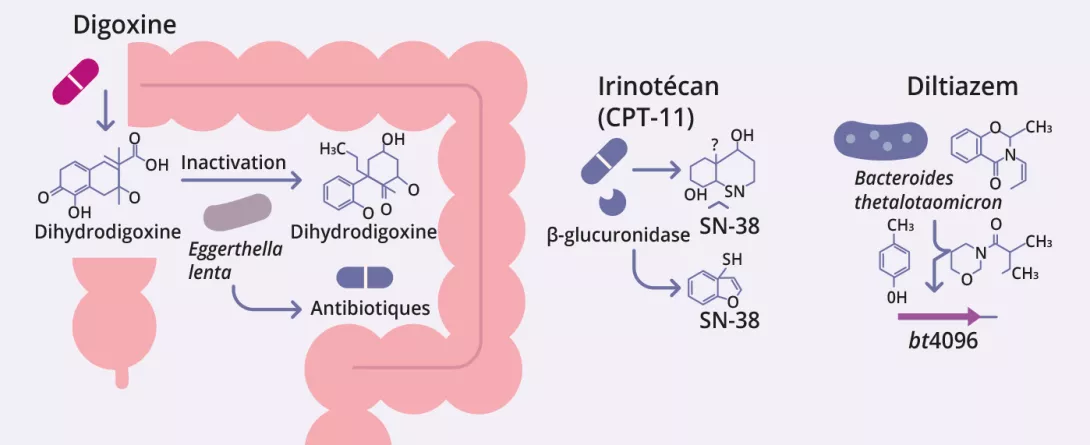
Le microbiote intestinal peut biotransformer les médicaments , modifiant ainsi leur activité, leur efficacité et leur toxicité (figure 2, tableau 3) 12-14. Zimmermann et al. ont schématisé le métabolisme microbien en analysant 271 médicaments oraux par rapport à 76 souches bactériennes intestinales ; ils ont alors constaté que 176 des médicaments étaient métabolisés par au moins une souche. Bacteroides dorei et B. uniformis ont notamment métabolisé près de 100 médicaments. Plus de 40 enzymes microbiennes ont été identifiées comme médiatrices d’un vaste ensemble de réactions, notamment la réduction, l’hydrolyse, la décarboxylation, la désalkylation et la déméthylation 12.
Javdan et al. ont mis au point une plateforme personnalisée (MDM-Screen) pour évaluer le métabolisme microbien des médicaments,
à l’aide du microbiote ex vivo de donneurs individuels. En analysant 575 médicaments, ils ont observé que 13 % étaient métabolisés par les microbes intestinaux, y compris de nombreuses interactions jusqu’alors inconnues. Ces transformations, telles que l’hydrolyse, la réduction et la désacétylation, peuvent activer, inactiver ou augmenter la toxicité du médicament. L’étude a également révélé une variabilité interindividuelle importante et identifié des gènes microbiens clés (ex. : uridine phosphorylase, β-glucuronidase), associés à des voies métaboliques spécifiques 15.
L’efficacité de certains médicaments pourrait dépendre davantage de la composition du microbiote que de la génétique de l’hôte.
Conséquences cliniques : avancée vers une médecine personnalisée
Les interactions entre le microbiote et les médicaments ont des implications cliniques non négligeables. En effet, les différences individuelles au niveau du microbiote peuvent expliquer la variabilité de la réponse aux médicaments et des effets secondaires. Il faut noter que la composition du microbiote n’est pas le seul facteur influençant les résultats du traitement ; c’est également le cas de sa stabilité fonctionnelle.
Dans les cas de mélanome avancé, les patients répondant bien au traitement anti-PD-1 ont présenté des fonctions microbiennes stables et des cellules T CD8+ réactives aux peptides bactériens de Lachnospiraceae, qui imitent les antigènes tumoraux. On peut en déduire que la fonctionnalité microbienne est un marqueur prognostique potentiel et un complément thérapeutique dans l’immunothérapie du cancer 16.
Ces résultats soulignent la nécessité d’intégrer la génomique humaine et microbienne aux évaluations pharmacologiques. Au cours du développement des médicaments, la simulation des interactions entre le microbiote et les médicaments in silico est devenue essentielle.
Dodd et Cane ont proposé un cadre détaillé combinant les systèmes in vitro (ex. : banque de souches, communautés dérivées de selles), les outils génétiques (tests de gain/perte de fonction) et la métagénomique pour identifier les gènes microbiens impliqués dans le métabolisme des médicaments. Les modèles murins gnotobiotiques permettent également de distinguer les effets microbiens de ceux de l’hôte sur la pharmacocinétique.
À mesure que ce domaine progresse, les prescriptions tenant compte du microbiote deviennent un moyen de personnaliser les traitements et de réduire les effets indésirables. À l’avenir, la pharmacomicrobiomique pourrait orienter le choix et la posologie des médicaments en fonction des biomarqueurs microbiens, ce qui permettrait de mettre en place une médecine véritablement personnalisée 17.
La personnalisation des traitements pourrait un jour nécessiter un profil microbiotique complet.
Préserver et restaurer le microbiote : une frontière thérapeutique
La protection du microbiote intestinal pendant un traitement médicamenteux est une stratégie prometteuse pour réduire les effets indésirables et préserver l’efficacité. Bien que les probiotiques et les prébiotiques présentent certains bénéfices face à la dysbiose médicamenteuse, leur efficacité varie. Les probiotiques ciblés, adaptés aux effets spécifiques des médicaments, et la transplantation de microbiote fécale (TMF) offrent des options plus fiables, notamment en cas d’infection à C. difficile récidivante.
Les outils de précision comme les inhibiteurs d’enzymes microbiens (ex. : les inhibiteurs de β-glucuronidase pour la toxicité de l’irinotécan), les probiotiques issus de la bioingénirie, les médicaments épargnant le microbiote et les interventions basées sur l’alimentation sont à l’étude. Les études cliniques explorent les symbiotiques adaptés aux schémas thérapeutiques pour améliorer les résultats tout en perturbant le moins possible le microbiote. Les postbiotiques, comme le butyrate, sont également évalués pour leurs effets anti-inflammatoires et de soutien de la barrière intestinale.
L’intégration de stratégies ciblant le microbiote à la pharmacologie nécessitera des outils avancés (multiomique, apprentissage automatique et modélisation systémique du microbiome) pour prédire et gérer efficacement les interactions entre le microbiote et les médicaments.
La manipulation du microbiote intestinal pourrait améliorer le taux de réussite des traitements et réduire les complications
Conclusion
Les interactions entre le microbiote et les médicaments sont un nouvel aspect important mais souvent négligé de la médecine, avec des implications majeures sur les résultats de traitement. L’intégration de ces connaissances à la pratique clinique est essentielle pour mettre au point des traitements plus sûrs, précis et respectueux du microbiote. À mesure que les données probantes s’accumulent, de nouvelles opportunités apparaissent pour moduler le microbiome afin d’accroître l’efficacité
des médicaments, de réduire leur toxicité et d’assurer la réponse aux médicaments.
Les approches innovantes, telles que les produits biothérapeutiques vivants, les microbes issus du génie génétique et les métabolites dérivés du microbiote (les « pharmabiotiques »), transforment la pharmacothérapie. Malgré un intérêt réglementaire croissant, les protocoles cliniques standardisés sont encore en développement. Dans un avenir proche, l’ingénierie du microbiome pourrait devenir une composante courante des soins médicaux personnalisés et systémiques.
- Valdes AM, Walter J, Segal E, Spector TD. Role of the gut microbiota in nutrition and health. BMJ 2018; 361: k2179.
- Zhao Q, Chen Y, Huang W, Zhou H, Zhang W. Drug-microbiota interactions: an emerging priority for precision medicine. Signal Transduct Target Ther 2023; 8: 386.
- Wallace BD, Wang H, Lane KT, et al. Alleviating cancer drug toxicity by inhibiting a bacterial enzyme. Science 2010; 330: 831-5.
- Bolte LA, Björk JR, Gacesa R, Weersma RK. Pharmacomicrobiomics: The Role of the Gut Microbiome in Immunomodulation and Cancer Therapy. Gastroenterology 2025 Online publication ahead of print.
- Nel Van Zyl K, Matukane SR, Hamman BL, Whitelaw AC, Newton-Foot M. Effect of antibiotics on the human microbiome: a systematic review. Int J Antimicrob Agents 2022; 59: 106502.
- Maier L, Goemans CV, Wirbel J, et al. Unravelling the collateral damage of antibiotics on gut bacteria. Nature 2021; 599: 120-4.
- Vich Vila A, Collij V, Sanna S, et al. Impact of commonly used drugs on the composition and metabolic function of the gut microbiota. Nat Commun 2020; 11: 362.
- Macke L, Schulz C, Koletzko L, Malfertheiner P. Systematic review: the effects of proton pump inhibitors on the microbiome of the digestive tract-evidence from next-generation sequencing studies. Aliment Pharmacol Ther 2020; 51: 505-26.
- Le Bastard Q, Berthelot L, Soulillou JP, Montassier E. Impact of non-antibiotic drugs on the human intestinal microbiome. Expert Rev Mol Diagn 2021; 21: 911-24.
- Maier L, Pruteanu M, Kuhn M, et al. Extensive impact of non-antibiotic drugs on human gut bacteria. Nature 2018; 555: 623-8.
- Weersma RK, Zhernakova A, Fu J. Interaction between drugs and the gut microbiome. Gut 2020; 69: 1510-9.
- Zimmermann M, Zimmermann-Kogadeeva M, Wegmann R, Goodman AL. Mapping human microbiome drug metabolism by gut bacteria and their genes. Nature 2019; 570: 462-7. •
- Haiser HJ, Gootenberg DB, Chatman K, Sirasani G, Balskus EP, Turnbaugh PJ. Predicting and manipulating cardiac drug inactivation by the human gut bacterium Eggerthella lenta. Science 2013; 341: 295-8.
- Takasuna K, Hagiwara T, Hirohashi M, et al. Involvement of beta-glucuronidase in intestinal microflora in the intestinal toxicity of the antitumor camptothecin derivative irinotecan hydrochloride (CPT-11) in rats. Cancer Res 1996; 56: 3752-7.
- Javdan B, Lopez JG, Chankhamjon P, et al. Personalized mapping of drug metabolism by the human gut microbiome. Cell 2020; 181: 1661-79.e22.
- Macandog ADG, Catozzi C, Capone M, et al. Longitudinal analysis of the gut microbiota during anti-PD-1 therapy reveals stable microbial features of response in melanoma patients. Cell Host Microbe 2024; 32: 2004-18.e9.
- Dodd D, Cann I. Tutorial: Microbiome studies in drug metabolism. Clin Transl Sci 2022; 15: 2812-37.

