Résilience du microbiote intestinal d'adultes en bonne santé suite à l'exposition à un antibiotique
Article commenté - Rubrique adulte
Par le Pr. Harry Sokol
Gastro-entérologie et nutrition, Hôpital Saint-Antoine, Paris, France

Commentaire de l’article original de Palleja et al. (Nature Microbiology 2018)
Pour minimiser l’impact des antibiotiques, les micro-organismes intestinaux ont et échangent des gènes de résistance aux antibiotiques appelés leur résistome. En utilisant une méthode de séquençage métagénomique par shotgun, les auteurs ont analysé l’éradication partielle du microbiote intestinal et sa résilience chez 12 hommes en bonne santé sur une période de 6 mois à la suite d’une intervention de 4 jours avec un cocktail de 3 antibiotiques de dernier recours : méropénème, gentamicine et vancomycine. Les changements initiaux comprenaient la prolifération d’entérobactéries et d’autres pathobiontes, comme Enterococcus faecalis et Fusobacterium nucleatum, et la disparition des bactéries du genre Bifidobacterium et des producteurs de butyrate. Le microbiote intestinal des sujets étudié est revenu à un état proche de l’état initial en 1,5 mois, bien que 9 espèces, qui étaient présentes chez tous les sujets avant le traitement, soient restées indétectables chez la plupart des sujets après 180 jours. Les espèces porteuses de gènes de résistance aux β-lactamines ont été sélectionnées positivement pendant et après l’intervention. Le portage de gènes de résistance aux glycopeptides ou aux aminoglycosides augmentaient les chances de colonisation de novo.
Les changements de composition du microbiote induits par une intervention antibiotique in vivo étaient concordants avec les résultats obtenus par des tests in vitro. Malgré une empreinte légère mais durable à la suite de l’exposition aux antibiotiques, le microbiote intestinal de jeunes adultes en bonne santé est résilient à une intervention antibiotique à large spectre à court terme, et leur portage de gène de résistance aux antibiotiques module leur processus de récupération. [1]
Que sait-on déjà à ce sujet ?
Le microbiote intestinal humain forme un écosystème complexe et équilibré. Les perturbations de cet écosystème peuvent jouer un rôle dans la survenue d’infections, d’obésité, de diabète et pathologies inflammatoires et neurologiques. Il est estimé qu’une augmentation de l’espérance de vie de 2 à 10 ans est attribuable aux antibiotiques. Cependant, l’exposition précoce aux antibiotiques a également été associée à des effets métaboliques, inflammatoires et neurologiques délétères, à la fois dans les modèles animaux et chez l’homme. Lorsqu’elles sont exposées à des antibiotiques, les communautés microbiennes réagissent non seulement en modifiant leur composition, mais aussi en évoluant, optimisant et diffusant les gènes de résistance aux antibiotiques (GRA), formant collectivement le résistome [2]. Le microbiote intestinal humain est considéré comme un réservoir pour les GRA, ses membres échangent ces gènes, propageant ainsi la résistance [3]. Le développement et la diffusion de la résistance microbienne aux antibiotiques est un grave problème de santé publique. Seules quelques études ont caractérisé les effets d’antibiotiques particuliers sur les écosystèmes intestinaux vis-à- vis des résistomes associés. Plusieurs études précédentes ont montré que l’administration d’antibiotiques induisait une diminution de la diversité et une augmentation du portage de GRA [4, 5]. Cependant, les effets d’une combinaison d’antibiotiques sur le microbiote et le rôle des GRA dans la persistance microbienne intestinale n’ont pas encore été étudiés. Dans la présente étude, 12 hommes en bonne santé (âgés de 18 à 40 ans) ont reçu un cocktail d’antibiotiques de dernier recours (vancomycine, gentamicine et méropénème), et les auteurs ont étudié les effets sur le microbiote intestinal en utilisant une approche par séquençage en shotgun sur des échantillons fécaux prélevés avant et à quatre moments différents sur une période de six mois après le traitement.
Fer de lance de l'arsenal thérapeutique moderne, les antibiotiques ont sauvé des millions de vie. En revanche, leur utilisation excessive et parfois injustifiée peut conduire à l'apparition de différentes formes de résistance chez les micro-organismes. Chaque année, l'Organisation Mondiale de la Santé (OMS) organise la Semaine mondiale de sensibilisation à la résistance aux antimicrobiens (WAAW) afin de sensibiliser la population sur ce problème de santé publique. Lisez la page qui y est consacrée.
Résistance aux antibiotiques : le microbiote au premier plan
Qu'est-ce que la Semaine mondiale de sensibilisation à la résistance aux antimicrobiens ?
Depuis 2015, l'OMS organise chaque année la Semaine mondiale de sensibilisation à la résistance aux antimicrobiens (WAAW) dont l'objectif est de sensibiliser sur le phénomène mondial de la résistance aux antimicrobiens. Cette campagne, qui se tiendra du 18 au 24 novembre, encourage le grand public, les professionnels de santé et les décideurs à faire un bon usage des antimicrobiens afin d'éviter l'apparition de résistance.
Quels sont les principaux résultats apportés par cette étude ?
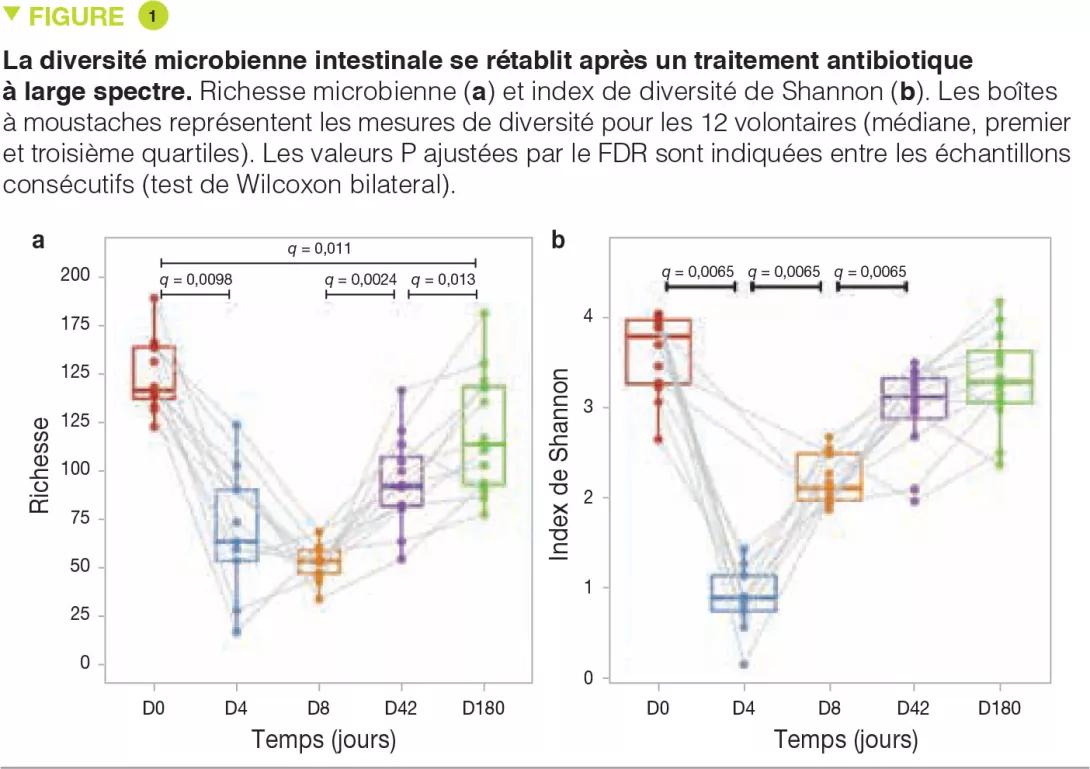
À J4, immédiatement après l’intervention, la richesse et la diversité du microbiote étaient considérablement réduites par rapport à J0. Cependant, malgré le très large spectre du traitement antibiotique utilisé, de nombreuses espèces étaient toujours détectables à J4 (Figure 1a). Dès J8, la diversité (mesurée par l’index de Shannon) avait considérablement augmenté, suggérant que les micro-organismes survivants avaient commencé à se régénérer (Figure 1b). La diversité était quasiment complètement revenue à l’état initial à 6 mois, mais ce n’était pas le cas pour la richesse, suggérant que certaines souches avaient été éliminées de manière définitive (ou au moins prolongée).
Parmi les changements précoces observés, il y avait un enrichissement en commensaux habituellement sous-dominants comme Escherichia coli, Veillonella spp., Klebsiella spp., Enterococcus faecalis et Fusobacterium nucleatum et une diminution majeure en bactéries productrices de butyrate comme Faecalibacterium prausnitzii, Roseburia hominis, Anaerostipes hadrus, Coprococcus spp. et Eubacterium spp. Ces altérations en composition n’étaient plus significatives dès J42.
Les auteurs ont ensuite investigué le rôle de GRA dans l’évolution du microbiote des sujets étudiés. Ils ont notamment observé que les espèces métagénomiques porteuses de β-lactamases avaient des chances de survie significativement plus élevées (OR = 1,64 [1,24-2,17]) à J8. D’autre part, les espèces métagénomiques non détectées à J0 avaient plus de chances de colonisation de novo à un point ultérieur si elles portaient des GRA contre l’une des trois classes utilisées.
Points clés
-
Le microbiote intestinal de jeunes adultes en bonne santé est résilient à quatre jours de traitement antibiotique à large spectre avec une récupération sur approximativement 6 mois de la plupart des communautés bactériennes
-
La récupération individuelle des espèces est modulée par le portage de GRA
-
L’effet de traitements antibiotiques prolongés ou itératifs reste à étudier, particulièrement en population pédiatrique
Quelles conséquences en pratique ?
Ces résultats démontrent que le microbiote intestinal de jeunes adultes en bonne santé est résilient à quatre jours de traitement antibiotique à large spectre avec une récupération de la plupart des communautés bactériennes en approximativement 6 mois. La récupération individuelle des espèces est modulée par le portage de GRA. D’autres études sont nécessaires pour évaluer l’effet de perturbations itératives et/ou sur des périodes plus prolongées et si ces résultats sont aussi valides chez l’enfant ayant un microbiote et un système immunitaire immatures. Il est possible que l’utilisation itérative d’antibiotiques sur des périodes prolongées sélectionne des bactéries porteuses de GRA au détriment d’autre bactéries commensales, avec des effets prolongés ou permanents sur le microbiote. Dans ce cas, des interventions correctrices avec apport exogène de micro-organismes pourraient être envisagées. Les effets des antibiotiques sur le microbiote intestinal sont donc importants et leur utilisation doit être raisonnée.
Conclusion
Les antibiotiques à large spectre impactent négativement le microbiote intestinal de manière immédiate, significative et durable pour certaines espèces. Chez les adultes jeunes en bonne santé, le microbiote intestinal est résilient mais la récupération quasi-complète prend environ 6 mois. Modulée par le portage de GRA, la capacité de régénération des espèces est plus favorable à la diversité qu’à la richesse.