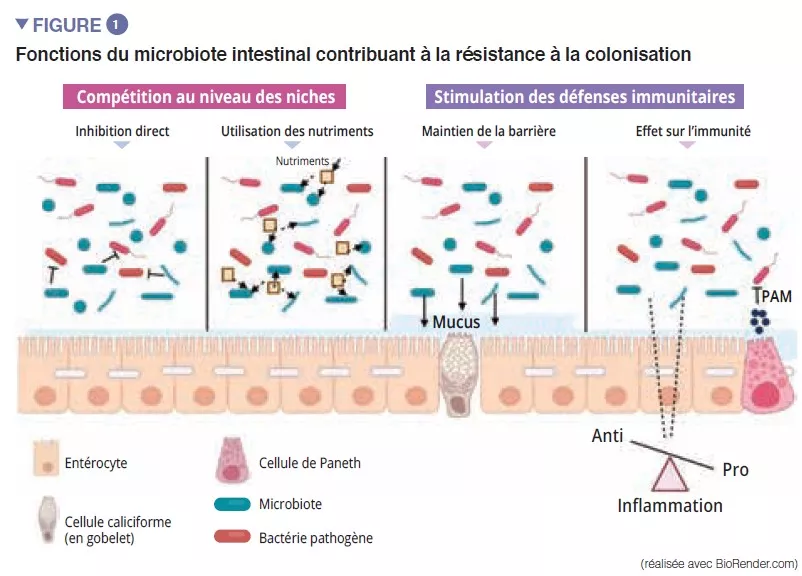
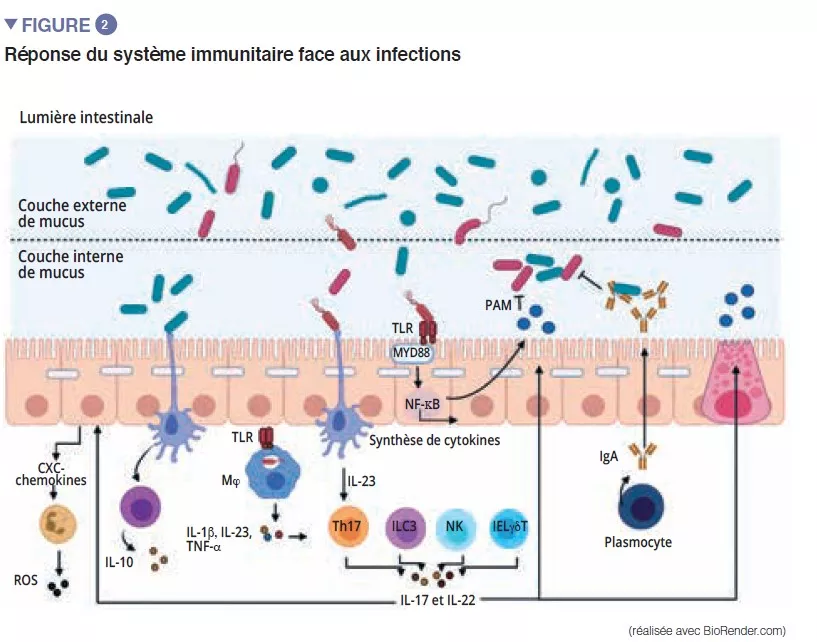
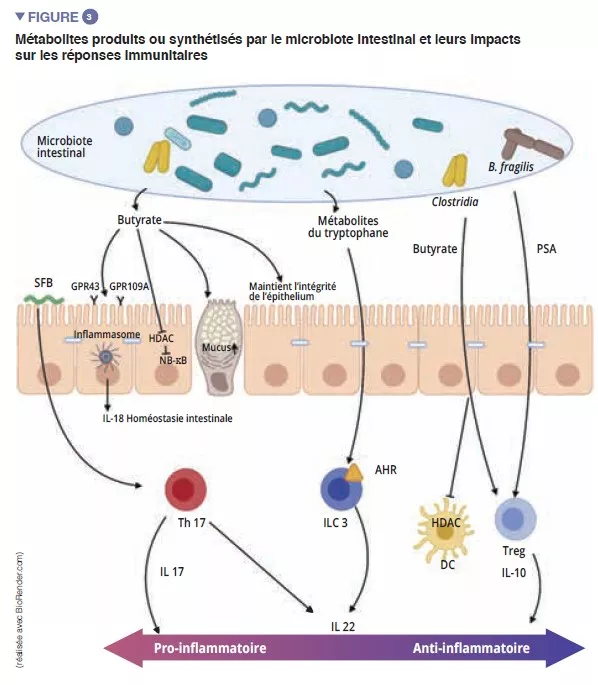
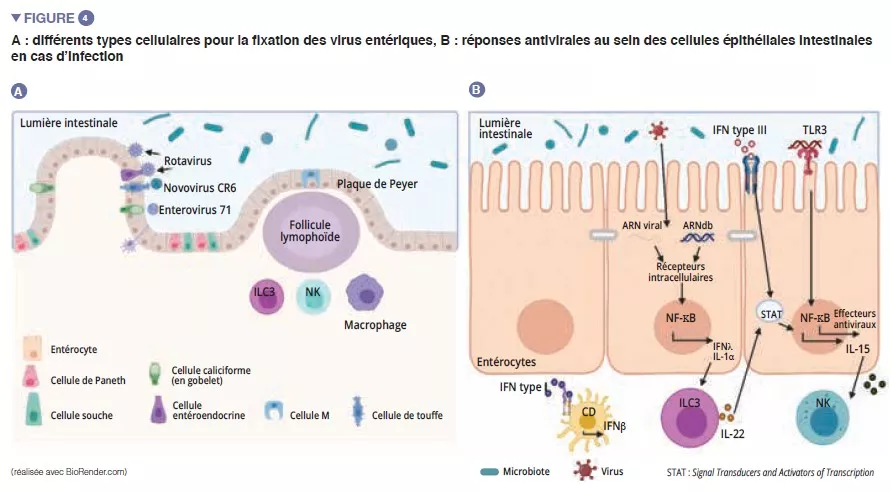
Le microbiote duodénal d'enfants ayant une dénutrition chronique avec retard de croissance secondaire à une entéropathie
Article commenté - Rubrique enfants
Par le Pr. Emmanuel Mas
Gastro-entérologie et nutrition, Hôpital des Enfants, Toulouse, France
Commentaire de l’article original de Chen et al. (N Engl J Med 2020)
L’entéropathie environnementale (EE) est un trouble énigmatique de l’intestin grêle qui jouerait un rôle dans la sous-nutrition infantile, un problème de santé mondial majeur. La définition de l’incidence de ce trouble a été entravée par l a difficulté de prélever directement des échantillons de la muqueuse de l’intestin grêle et du microbiote. Cette étude se concentre sur 110 jeunes enfants présentant un retard de croissance linéaire et vivant dans un bidonville du Bangladesh, et n’ayant pas bénéficié d’une intervention nutritionnelle. Les auteurs ont pratiqué une endoscopie chez 80 enfants dont la biopsie avait confirmé la présence d’une EE et qui disposaient d’échantillons de plasma et de duodénum. Parmi les souches bactériennes obtenues chez les enfants, les niveaux absolus d’un groupe de 14 taxa communs (non typiquement classés comme entéropathogènes) ont été négativement corrélés avec la croissance linéaire et positivement corrélés avec les protéines duodénales impliquées dans les réponses immuno-inflammatoires. La représentation de ces 14 taxa duodénaux dans le microbiote fécal était significativement différente de celle des échantillons provenant d’enfants en bonne santé. L’entéropathie de l’intestin grêle s’est développée chez des souris gnotobiotiques qui avaient été colonisées à partir d’une culture de souches bactériennes duodénales provenant d’enfants atteints de DEE. Ces résultats confirment l’existence d’une relation de cause à effet entre le retard de croissance et les composants du microbiote de l’intestin grêle et l’entéropathie, et justifient la mise au point de thérapies ciblant ces contributions microbiennes à l’EE. [1]
Que sait-on déjà à ce sujet ?
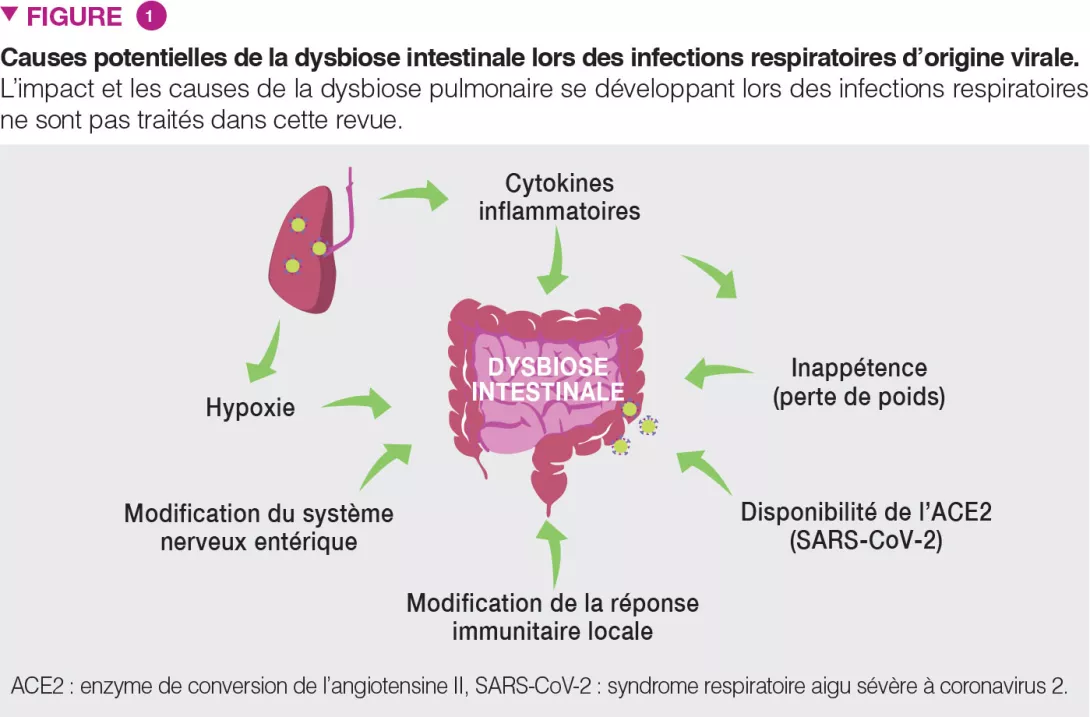
La proportion de dénutrition chronique avec ralentissement statural est de 25 % chez les nourrissons qui ont eu préalablement plus de 5 épisodes diarrhéiques. Ces infections intestinales répétées entraînent une EE, entité caractérisée par une atrophie villositaire associant une diminution de la surface intestinale et des capacités d’absorption, une altération de la barrière intestinale et une inflammation de la muqueuse. Des données plus récentes suggèrent qu’une dysbiose du microbiote du tractus digestif haut serait présente dans l’EE.
Quels sont les principaux résultats apportés par cette étude ?
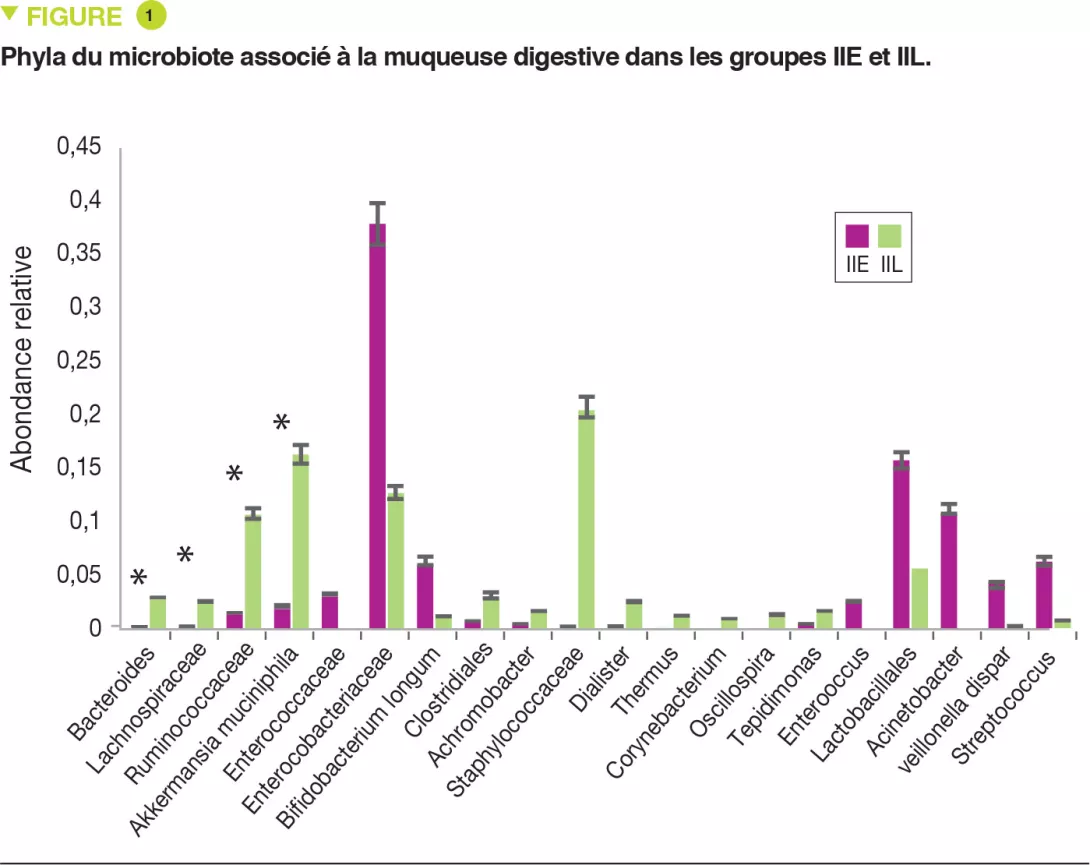
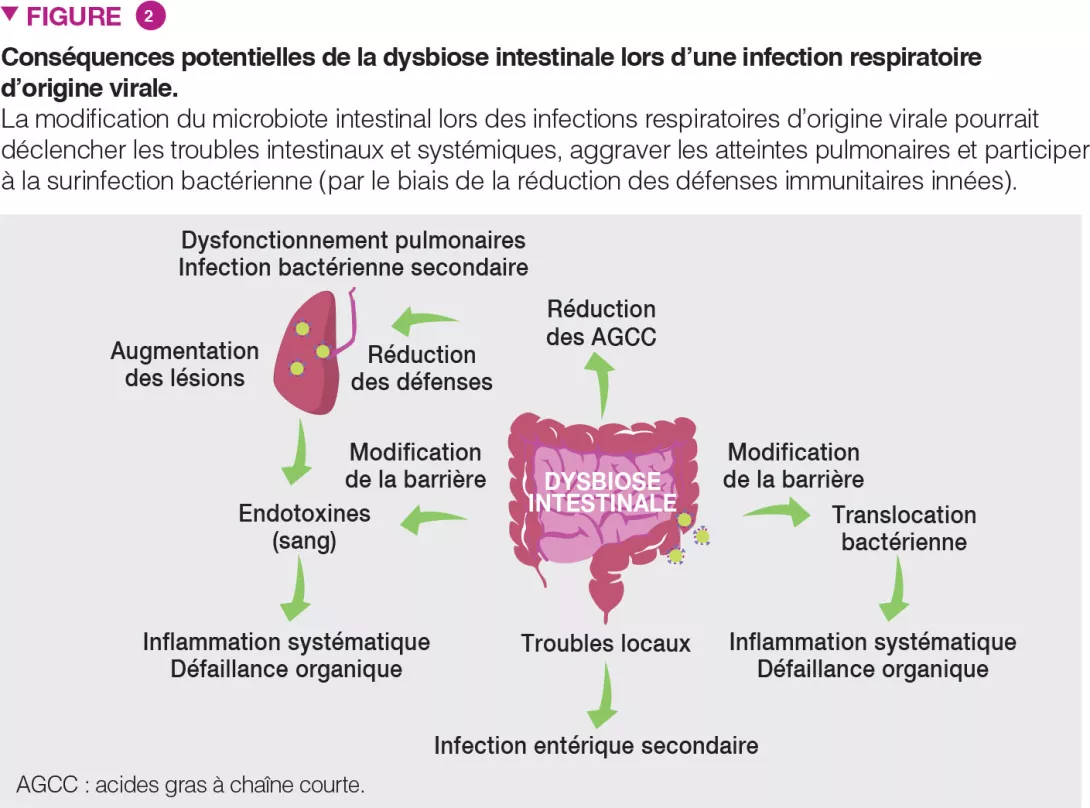
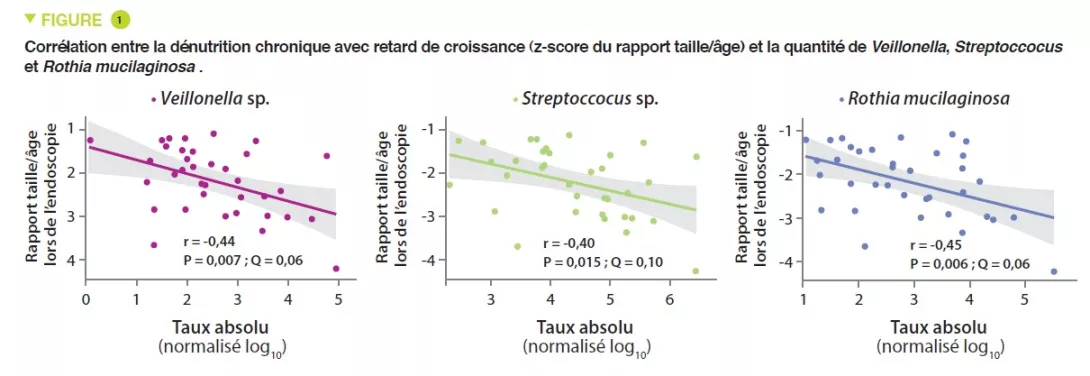
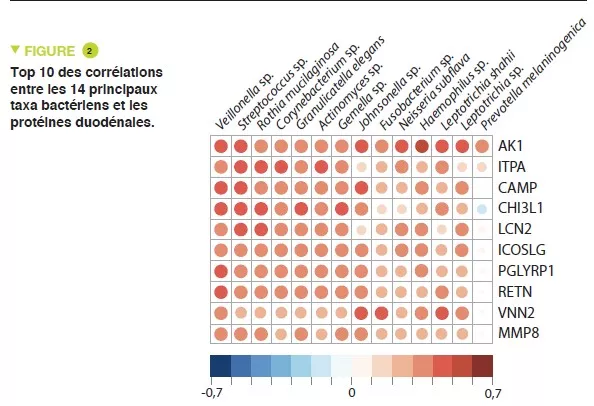
Cette étude a inclus 110 nourrissons âgés en moyenne de 18 mois, originaires de Dhaka, Bangladesh, qui avaient une dénutrition chronique avec retard de croissance, définie par intervention nutritionnelle. Les biopsies duodénales confirmaient une EE chez 80 d’entre eux. Le microbiote d’aspirations duodénales a pu être analysé pour 36 de ces nourrissons ; un groupe de 14 taxa bactériens était présent chez plus de 80 % de ceux-ci et corrélé négativement au rapport taille/âge (r = – 0,049, p = 0,003) (Figure 1). L’étude protéomique des biopsies duodénales a montré une corrélation positive entre ces 14 taxa et 10 protéines dont 2 peptides antimicrobiens, un marqueur de l’inflammation intestinale (LCN2), et négative avec 10 protéines produites par des entérocytes (Figure 2).
Chez les 80 nourrissons ayant une EE prouvée, l’étude protéomique plasmatique a montré une corrélation positive forte avec REG3A et LCN2.
La comparaison du microbiote fécal de nourrissons ayant une EE et de 27 témoins a mis en évidence une augmentation significative des espèces du genre Veillonella, bactéries qui étaient corrélées le plus fortement aux protéines duodénales impliquées dans l’inflammation digestive.
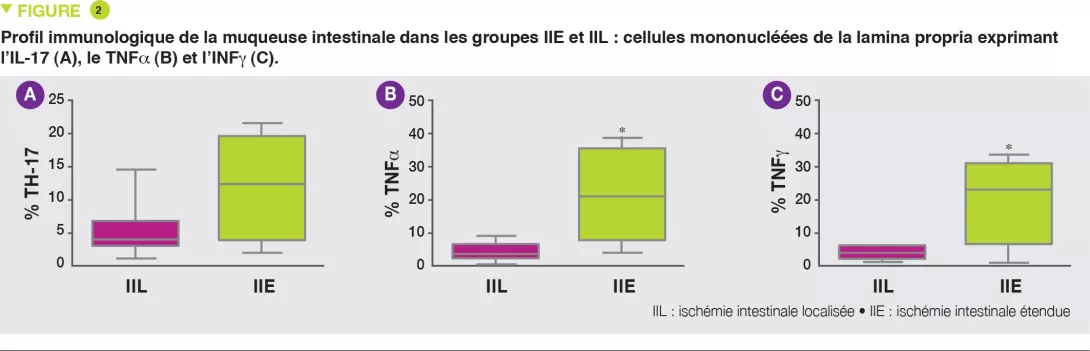
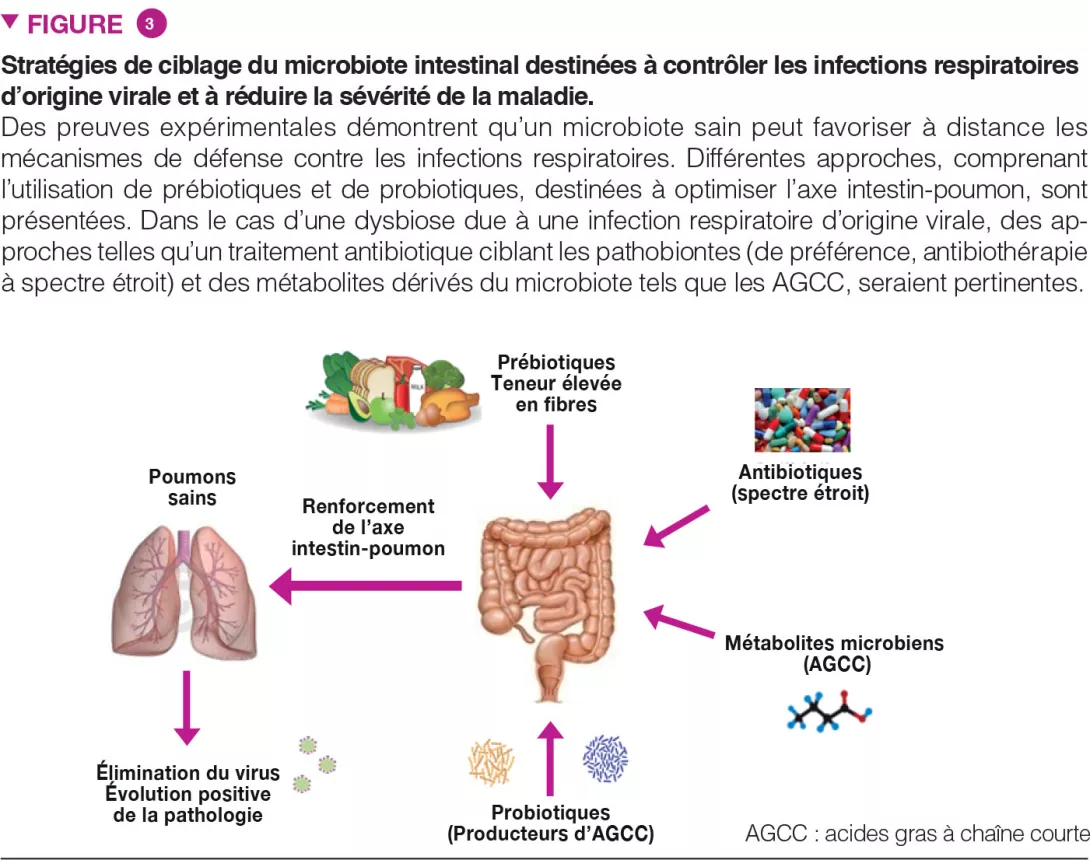
Après avoir cultivé 39 souches bactériennes à partir d’aspirations duodénales de nourrissons ayant une entéropathie environnementale, dont 11 des 14 taxa, celles-ci ont été administrées par gavage oral à des souris nourries avec une alimentation similaire à celle d’un nourrisson de 18 mois de Dhaka. Vingt-trois de ces bactéries étaient retrouvées à une abondance relative > 0,1 % à au moins un niveau du tractus digestif. Les souris témoins recevaient un gavage oral du microbiote caecal de souris conventionnelles. À la différence des souris témoins, il existait chez les souris recevant les bactéries « entéropathie environnementale » un infiltrat inflammatoire de la lamina propria de l’intestin grêle à cellules mononucléées, ainsi que des anomalies épithéliales et des anomalies architecturales avec allongement des cryptes. Ces anomalies avaient une localisation par plaques dans l’intestin grêle mais elles ne touchaient pas le côlon.
Au niveau fonctionnel, les résultats montraient chez ces souris une augmentation des ARNm de peptides anti-microbiens (Reg3b et Reg3g), d’une métalloprotéinase (MMP8) et une diminution des ARNm codant pour des protéines des jonctions serrées. Ces altérations de la réponse immunitaire innée et de la barrière épithéliale pourraient expliquer la translocation systémique de bactéries dans la rate (Escherichia coli et Enteroccocus hirae).
Points clés
-
L’entéropathie environnementale est favorisée par une perturbation du microbiote intestinal au niveau du duodénum
-
Cette dysbiose duodénale est corrélée à la dénutrition chronique
-
La pathologie est transmissible à la souris, ce qui pourra aider dans la connaissance des mécanismes physiopathologiques impliqués (inflammation intestinale, anomalies de la barrière épithéliale et altérations immunitaires de la reconnaissance bactérienne)
Conclusion
Les résultats de cette étude suggèrent une relation de causalité entre des bactéries du duodénum, l’entéropathie environnementale et une dénutrition chronique avec retard de croissance statural. Il serait donc intéressant pour ces enfants de développer des traitements ciblant cette dysbiose.