Microbiota vaginal #11
Por el Pr. Markku Voutilainen
Facultad de medicina de la Universidad de Turku; gastroenterología, Hospital Universitario de Turku, Finlandia


Por el Pr. Markku Voutilainen
Facultad de medicina de la Universidad de Turku; gastroenterología, Hospital Universitario de Turku, Finlandia

El fracaso repetido de implantación (FRI) se define como un fracaso para conseguir un embarazo clínico después de haber implantado al menos cuatro embriones de buena calidad a una mujer de menos de 40 años. Factores embrionarios o uterinos o enfermedades multisistémicas de la madre pueden causar FRI, pero en algunas mujeres la etiología no es reconocible. Los autores se centraron en la microbiota vaginal y el metaboloma de mujeres con FRI. Observaron que las pacientes con FRI sufrían disbiosis vaginal, ya que presentaban bacterias más diversas y abundantes, con un aumento de bacterias anaeróbicas y aeróbicas, que podían estar relacionadas, respectivamente, con la vaginosis bacteriana y la vaginitis aeróbica o con infecciones urinarias. Por el contrario, su microbiota vaginal presentaba una reducción del género Lactobacillus (LB): había un descenso de L. iners mientras que L. crispatus era la especie más abundante en el grupo FRI. Una mayor diversidad bacteriana vaginal, la disminución de LB y los cambios metabólicos asociados podrían servir como biomarcadores capaces de predecir el riesgo de FRI.
Esta revisión resume la función que desempeña la microbiota vaginal en las infecciones urinarias (IU) ante las crecientes evidencias que indican que la vagina podría servir como un reservorio de uropatógenos y aumentar la susceptibilidad a UI. Escherichia coli es la causa más común de UI y puede colonizar la vagina, donde puede crecer si la colonización vaginal de Lactobacillus (LB) es reducida. Se suelen detectar algunas bacterias vaginales en la orina, pero sus características como uropatógenos se infravaloran, ya que son difíciles de detectar en la práctica clínica habitual. Por ejemplo, la vaginosis bacteriana (VB) se caracteriza por anaerobios gramnegativos, especies que pertenecen a los tipos Actinobacteria y Firmicutes mientras que LB es reducido y las pacientes con VB presentan un mayor riesgo de IU. En la VB se detecta Gardnerella vaginalis, que puede causar una IU aguda o recurrente. Los Streptococcus del grupo B pueden causar tanto vaginitis aeróbica como IU. Por último, algunas bacterias vaginales pueden acceder a las vías urinarias y transitarlas brevemente, causar una inmunomodulación o lesiones, y desequilibrar las interacciones huésped-patógeno, lo que influye en la evolución de la uropatogénesis.
De vuelta del congreso
Por el Dr Dragos Ciocan
Servicio de Hepatogastroenterología y nutrición, Hospital Antoine-Béclère, Clamart, Francia

A pesar de la pandemia de la COVID-19, este año hemos podido asistir a una edición en línea de las Jornadas Francófonas de Hepatogastroenterología (eJFHOD), con la ventaja añadida de poder disfrutar de las presentaciones grabadas del 3 al 20 de julio de 2020. Estas jornadas congregaron a más de 7924 asistentes, y se visualizaron 172 937 páginas. Como cada año, durante el congreso se presentaron estudios originales sobre la microbiota intestinal (MI).
El cáncer colorrectal esporádico (CCR), tercer cáncer más frecuente en el hombre, se desarrolla por interacciones entre el huésped y el entorno que podrían implicar a la MI [1]. El profesor Sobhani presentó los resultados de un estudio que examinó la relación entre los mecanismos epigenéticos promovidos por bacterias de la MI en la aparición de los CCR [2]. En ratones a los que se habían trasplantado heces de pacientes con CCR aparecieron lesiones cólicas precancerígenas, asociadas a un aumento de genes hipermetilados. Los donantes con CCR presentaban anomalías de metilación de ciertos genes promotores asociados a una disbiosis intestinal. Un estudio piloto (n = 266) llevado a cabo en humanos utilizó firmas microbianas y epigenéticas identificadas para perfeccionar un análisis de sangre que permita diagnosticar el CCR. Se identificó un índice de metilación acumulado (CMI por sus siglas en inglés, que mide el nivel de hipermetilación de 3 genes) como un factor predictivo de la aparición de CCR. Estos resultados fueron confirmados en una cohorte prospectiva de 1000 pacientes. La disbiosis intestinal de los pacientes con CMI positivo se caracterizaba por un aumento de especies bacterianas que favorecen la metilación. Estos trabajos indican que es posible que la disbiosis intestinal asociada al CCR promueva la carcinogénesis del colon mediante una alteración de la metilación de ciertos genes. El índice acumulado de hipermetilación (CMI) y/o las bacterias que favorecen la metilación podrían ser, por tanto, marcadores para el diagnóstico, o utilizarse en la evaluación de los efectos de tratamientos moduladores de la MI en el CCR.
En un estudio coordinado por el profesor Seksik, los autores analizaron la función de la MAM (molécula antinflamatoria de la microbiota, producida por Faecalibacterium prausnitzii y reducida en pacientes con enfermedad de Crohn, EC [3]) como biomarcador de disbiosis intestinal y como ayuda para el diagnóstico de la EC. Los autores han demostrado que la pérdida de MAM está asociada al diagnóstico de la EC. Este estudio preliminar realizado con un número reducido de pacientes (24 pacientes durante un brote, 24 en remisión y 12 testigos sanos) abre la vía al diagnóstico de la EC a partir de la MI, pero estos resultados preliminares tendrán que ser confirmados con cohortes independientes.
Se sabe que las bacterias detectan señales ambientales y responden a ellas (aptitud conocida como «Quorum Sensing »). De entre las moléculas de este sistema, la 3-oxo-C12:2 se reduce en los pacientes con enfermedades inflamatorias crónicas intestinales (EICI), disminución que parece estar correlacionada con la disbiosis intestinal observada [4]. En un estudio presentado por el doctor Aguanno, los autores analizaron el impacto de esta molécula en las células epiteliales del intestino, y demostraron que esta no modifica la permeabilidad paracelular, pero atenúa las alteraciones de las uniones estrechas inducidas por las citocinas proinflamatorias. En un segundo estudio, Coquant et al. demuestran que la 3-oxo-C12:2 ejerce un efecto antinflamatorio en las células inmunitarias, en parte mediado por el receptor T2R138. Por tanto, es posible que esta molécula tenga efectos protectores en la barrera intestinal, module la respuesta inflamatoria y, por consiguiente, represente una nueva pista terapéutica en las EICI.
Artículo comentado - niños
Por el Pr Emmanuel Mas
Gastroenterología y nutrición, Hospital de Niños, Toulouse, Francia
La microbiota de las primeras semanas de vida es decisiva en la salud, al actuar como una barrera contra la invasión de los patógenos y al conservar la homeostasis inmunitaria intestinal. En recién nacidos afectados con isquemia intestinal se ha observado una alteración del ecosistema de la microbiota fecal (MF). El objetivo de este estudio era describir, en estos pacientes, la MF, la microbiota de las mucosas (MM) y la inmunidad de las mucosas.
Catorce recién nacidos recibieron una resección intestinal a causa de una isquemia intestinal. Según la gravedad de las lesiones, se identificaron dos grupos: isquemia intestinal extensa (IIE) e isquemia intestinal localizada (IIL). Este estudio demostró la variación de la MF y de la MM en los grupos IIE e IIL.
La microbiota intestinal de los recién nacidos se caracteriza por una menor diversidad bacteriana y una proporción más importante de bacterias patógenas. Además, su sistema inmunitario intestinal es inmaduro. Estos dos factores alteran la barrera epitelial intestinal y favorecen la producción de mediadores proinflamatorios.
La enterocolitis úlceronecrosante (ECUN) es una afección isquémica e inflamatoria digestiva que afecta a los recién nacidos prematuros. La fisiopatología de la ECUN aún no se conoce totalmente, pero se produce una disbiosis intestinal, además de un proceso inflamatorio. La utilización de antibióticos y de antiácidos favorece la disbiosis y también aumenta el riesgo de aparición de ECUN.
Existen otras afecciones isquémicas e inflamatorias que pueden afectar a los recién nacidos, como el vólvulo intestinal y las perforaciones digestivas localizadas.
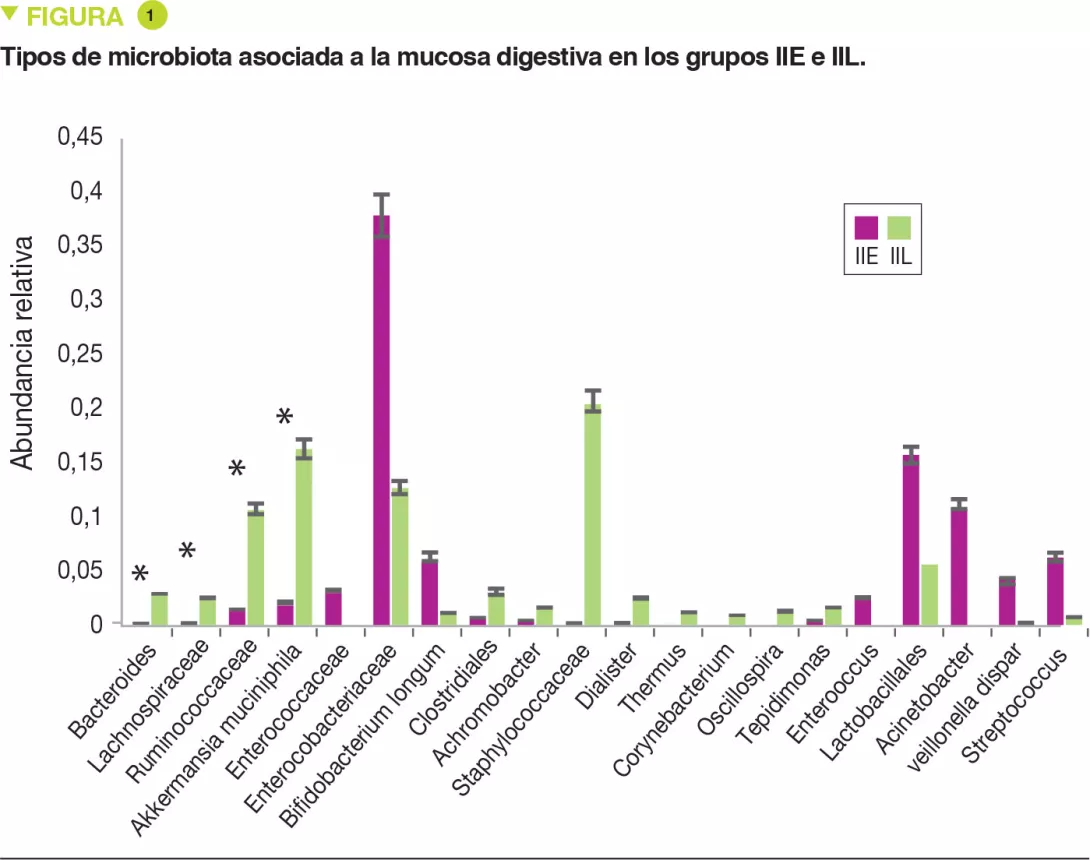
Este estudio piloto monocéntrico caracterizó la MF y la MM así como la células mononucleares de la lámina propia y las citocinas proinflamatorias de dos grupos de recién nacidos a término o prematuros. Siete bebés presentaban una isquemia intestinal extensa (IIE) (5 ECUN, 1 vólvulo intestinal y 1 isquemia cólica total) y otros siete una isquemia intestinal localizada (IIL) (4 perforaciones aisladas y 3 atresias intestinales). Se utilizó como referencia la MF de 9 recién nacidos a término. La MM de los recién nacidos con una IIE, comparada con los que tenían una IIL, contenía: más proteobacterias (p = 0,049) y menos bacteroidetes (p = 0,007) y verrucomicrobios (p = 0,01) (Figura 1) ; menos Bacteroides, Lachnospiracee, Ruminococcaceae y Akkermansia muciniphila (p < 0,05).
La MF presentaba una menor diversidad (índice de Shannon) en los pacientes con IIE que en los que tenían IIL (p = 0,01). La abundancia relativa fue similar a la MM entre IIE e IIL para las proteobacterias y firmicutes (p < 0,05). Asimismo, se encontró una distribución bacteriana con más Enterobacteriaceae en los pacientes con IIE y más Ruminococcaceae, Bacteroides, Lachnospiracee y Staphylococcaceae en los pacientes con IIL.
El grupo IIE presentaba un aumento de los linfocitos B, T y NK (Natural Killer), en concreto linfocitos T CD3+, TH17 (Figura 2A) y un descenso de linfocitos T reguladores (Tregs), con un aumento de las células que expresaban el FNTa (Figura 2B) y el IFNa (Figura 2C).
La enterocolitis úlceronecrosante es una patología digestiva grave que afecta a los recién nacidos prematuros.
Es posible que las anomalías de la microbiota fecal y de la microbiota asociada a la mucosa digestiva intervengan en el proceso inflamatorio e isquémico de la ECUN.
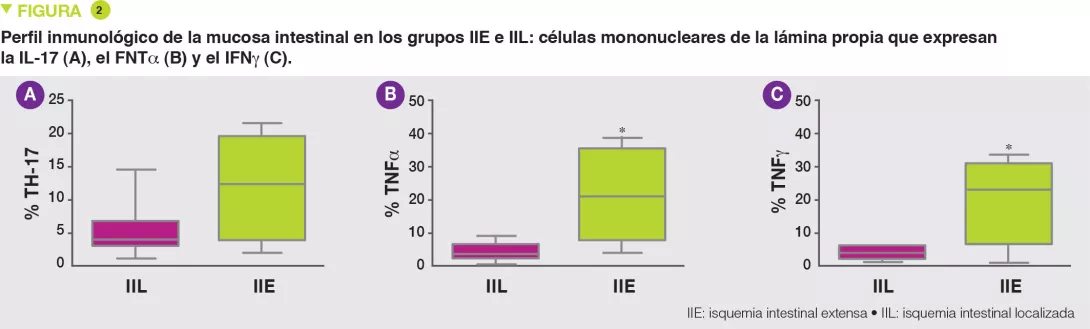
Este estudio piloto confirma que una diversidad bacteriana reducida y la predominancia de Enterobacteriaceae son factores de riesgo de ECUN, al igual que la reducción de las tasas de Akkermansia muciniphila. Es posible que una corrección de esta disbiosis permita modificar el desequilibrio TH17/Tregs y reducir la producción de mediadores de la inflamación (FNTa e IFNa).
La microbiota fecal y la microbiota asociada a la mucosa digestiva tienen características específicas en los bebés prematuros con lesiones isquémicas. Se necesitan estudios complementarios para determinar la función de estas bacterias en el proceso inflamatorio e isquémico de la ECUN.
Artículo comentado - Adulto
Por el Pr Harry Sokol
Gastroenterología y nutrición, Hospital Saint-Antoine, París, Francia

El microbioma humano posee una gran capacidad metabólica, pero nuestra comprensión de los mecanismos que vinculan las bacterias intestinales con el metabolismo humano sigue siendo limitada. En el artículo, los autores se centran en la conversión por parte de la microbiota intestinal del colesterol en coprostanol, un esterol que se absorbe mal, para desarrollar un marco para la identificación de las enzimas funcionales y de las bacterias. Mediante la integración de datos aparejados de metagenómica y de metabolómica provenientes de cohortes existentes con conocimientos y experimentos bioquímicos, los autores proponen predecir y validar la existencia de un grupo de deshidrogenasas bacterianas del colesterol que contribuye a la formación del coprostanol. Estas enzimas se codifican por los genes ismA en un grupo de bacterias no cultiva das, comunes en cohortes humanas geográficamente diversas. Los individuos que albergan las bacterias intestinales que contribuyen a la formación del coprostanol tienen índices de colesterol fecal significativamente más bajos y un co lesterol total sérico inferior con efectos comparables a los que se atribuyen a las variaciones de los genes de la homeostasis lipídica. Por ello, es posible que el metabolismo del colesterol por parte de estas bacterias desempeñe una función importante en la reducción de las concentraciones intestinales y séricas de colesterol, con un impacto directo en la salud humana
On El colesterol es una molécula biológica clave que funciona como un componente estructural de todas las membranas celulares animales y es un precursor de las hormonas esteroides, de la vitamina D y de los ácidos biliares. Dos fuentes principales de colesterol influyen en su concentración en el suero: el colesterol endógeno sintetizado en el hígado y el colesterol exógeno derivado de componentes alimenticios de origen animal (Figura 1). El colesterol sintetizado en los hepatocitos se transporta hacia la vesícula biliar y después se secreta en el intestino delgado con otras sales biliares. En el intestino, el colesterol biliar (~ 1-2 g/día) se mezcla con el colesterol alimenticio (~ 0,2-0,4 g/día en la alimentación americana media) y, finalmente, ambas son transportadas en los enterocitos para ser acondicionadas en partículas de lipoproteínas y secretadas en el plasma. La hipercolesterolemia es un factor de riesgo de las enfermedades cardiovasculares (ECV), causantes de un cuarto de todos los fallecimientos en los países industrializados.
La reducción del transporte del colesterol en el intestino es una estrategia clínicamente aprobada para reducir el índice de colesterol sérico. Una serie de bacterias intestinales metabolizan y modifican las moléculas alimenticias derivadas del huésped en el intestino delgado. Como ambas fuentes de colesterol atraviesan este entorno, es posible que la microbiota intestinal influya en los índices de colesterol sérico. De hecho, la transferencia de microbiota de donantes humanos con índices de colesterol sérico elevados es capaz de transmitir este fenotipo hipercolesterolémico a ratones [2, 3]. Otros estudios indican que la administración de especies bacterianas particulares puede tener efectos hipocolesterolemiantes [4]. Sin embargo, en la actualidad se desconocen los mecanismos precisos en que se fundamentan estas observaciones. Es posible que la microbiota intestinal ejerza efectos hipocolesterolemiantes metabolizando el colesterol intestinal en coprostanol (Figura 1), lo que reduciría la cantidad de colesterol absorbida por el intestino.
Desde principios de 1900, se sabe que esta transformación, que depende de la microbiota, se produce en los humanos. Se ha informado de la existencia de varias bacterias intestinales que generan coprostanol con características físicas y bioquímicas similares, a partir de varias fuentes, en concreto ratas, babuinos y humanos. Sin embargo, la mayoría de las cepas no está disponible actualmente y nunca se ha secuenciado. Los primeros trabajos han mostrado que la formación de coprostanol por este grupo de bacterias intestinales pasa por una vía de reducción indirecta que implica la oxidación inicial del colesterol (1) en colestenona (2), seguida de una reducción del doble enlace D4,5 para formar la coprostanona (3), y de la rerreducción posterior de la cetona para generar coprostanol (4) (Figura 1). Las enzimas bacterianas responsables de este metabolismo aún no se han identificado. Más recientemente, otros informes han implicado a otras bacterias intestinales filogenéticamente diversas en la formación del coprostanol [5]. A pesar de que los esfuerzos que han intentado elucidar cómo afecta el metabolismo microbiano intestinal del colesterol a los índices de colesterol sérico se remontan a más de 100 años, los elementos mecanísticos siguen siendo desconocidos a causa de una comprensión limitada de las bacterias intestinales, genes y enzimas responsables de la formación del coprostanol.
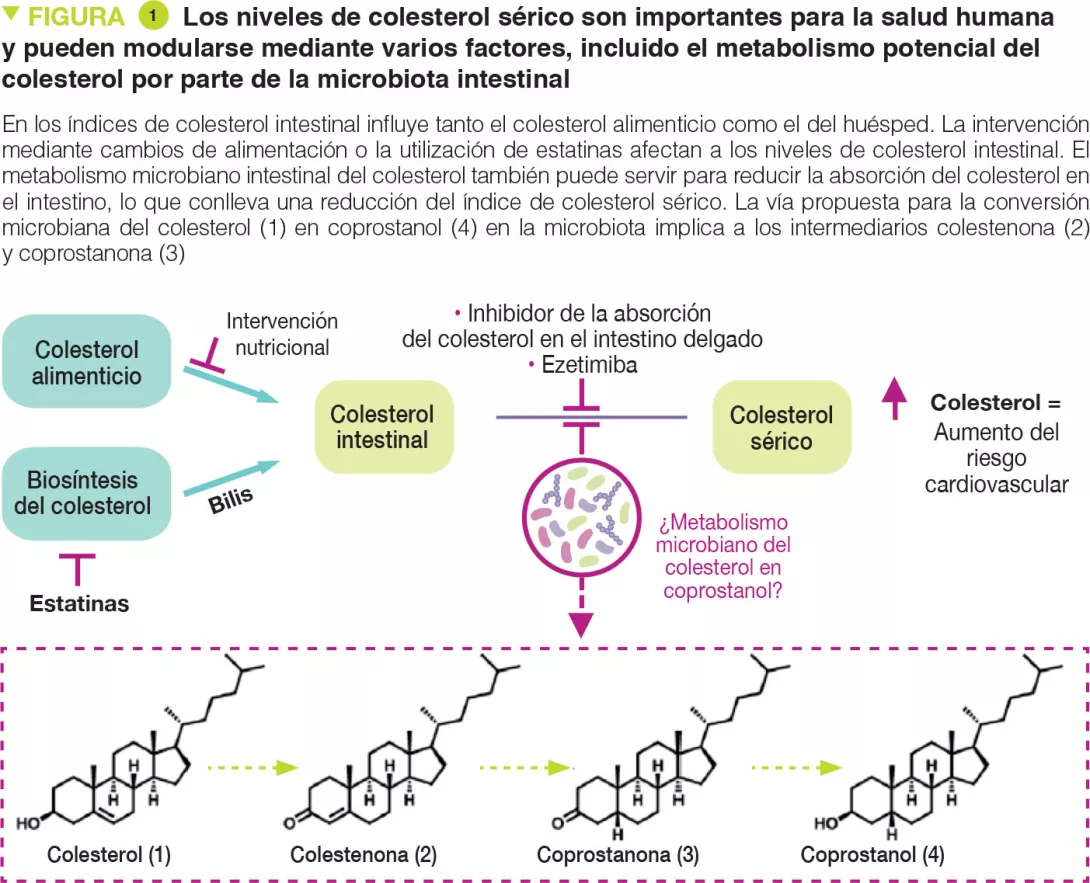
Ciertas bacterias de la microbiota intestinal humana poseen enzimas, de la familia ismA, capaces de degradar el colesterol.
La presencia de especies ismA + en la microbiota intestinal se asocia a una disminución del colesterol fecal y sérico en el ser humano.
El efecto de las especies ismA + en el colesterol sérico es comparable al de la genética humana.
Los autores han utilizado una estrategia multidisciplinar para descubrir las enzimas bacterianas intestinales. Esta estrategia, basada en correlaciones entre datos de metagenómica y de metabolómica provenientes de cohortes humanas existentes, ha permitido identificar y caracterizar una extensa familia de enzimas deshidrogenasa del colesterol a partir de un grupo de bacterias intestinales no cultivadas que intervienen en el metabolismo del colesterol en coprostanol. En primer lugar, la enzima responsable de la primera etapa de degradación del colesterol, denominada ismA, se identificó en Eubacterium coprostanoligenes, una bacteria que ya se conocía por esta función. El análisis de los datos de secuenciación de cohortes humanas permitió identificar, a continuación, enzimas homólogas en un grupo de bacterias anaerobias no cultivadas.
La presencia de estos genes ismA en el microbioma estaba asociada a la presencia de coprostanol en las heces y a una reducción de los índices de colesterol fecal. Por último, para demostrar el potencial de estas bacterias de influir en la salud humana, los autores han demostrado que la presencia de genes ismA en los metagenomas humanos está asociada a una disminución de las concentraciones de colesterol total en el plasma, comparable a los efectos observados a partir de variantes de genes humanos implicados en la homeostasia lipídica.
En conjunto, estos resultados confirman la función del metabolismo bacteriano intestinal en la modulación de los niveles de colesterol del anfitrión en el intestino, pero, lo que es más importante, también a nivel sistémico. Estos trabajos abren la vía a la utilización de la microbiota intestinal como biomarcador predictivo del riesgo de hipercolesterolemia y sientan las bases de intervenciones terapéuticas dirigidas a la microbiota.
Este estudio pone de relieve la función de la microbiota intestinal en la degradación del colesterol con efectos en el nivel sérico de colesterol. Es posible que en un futuro próximo la microbiota intestinal sea un objetivo de las terapias hipocolesterolemiantes.
Artículo comentado - Adulto
Por el Prof. Harry Sokol
Gastroenterología y nutrición, Hospital Saint-Antoine, París, Francia

TEl microbioma intestinal está implicado en gran cantidad de tr astornos gastrointestinales crónicos en humanos. Sin embargo, la falta de correlación entre estudios hechos en animales y en humanos y la ausencia de una perspectiva multiómica integrada de los cambios fisiológicos específicos de l a enfermedad dificultan la determinación de su función. Los autores han integrado datos multiómicos longitudinales del microbioma intestinal, del metaboloma, del epigenoma del hospedador y del transcriptoma, en el contexto de la fisiología de pacientes con síndrome del intestino irritable (SII). Asimismo, han identificado variaciones específicas de subtipos de SII en la composición y l as funciones microbianas, y relacionadas con la sintomatología. Un subconjunto de cambios identificados en metabolitos microbianos corresponde a mecanismos fisiológicos del huésped pertinentes para el SII. Los autores, tras recopilar varios niveles de datos, han identificado el metabolismo de las purinas como una nueva v ía del metabolismo hospedador-microbiota en el SII, con una aplicación terapéutica potencial. Este estudio hace hincapié en el interés que presentan el mues treo longitudinal y la integración de datos multiómicos complementarios para identificar los mecanismos funcionales que pueden ser dianas ter apéuticas en una estrategia de tratamiento global de las enfermedades intestinales crónicas.
IEl síndrome del intestino irritable (SII) es un trastorno observado en pacientes de todo el mundo que se caracteriza por un dolor o una incomodidad abdominal recurrente. El SII, que se manifiesta principalmente en mujeres, está asociado a cambios en la forma o la frecuencia de las heces; son esas formas las que definen los subtipos del SII: con predominio de estreñimiento (SII-E), con predominio de diarrea (SII-D) o mixto (SII-M).
La patogénesis del SII conlleva modificaciones en la motilidad gastrointestinal, en la secreción intestinal, en la hipersensibilidad visceral y en la permeabilidad intestinal, y todas ellas pueden ser modificadas por el microbioma intestinal [2]. Además, los síntomas del SII se ven afectados por el régimen alimenticio, la genética del huésped y el entorno, factores conocidos por modular el microbioma intestinal humano [2]. Las pruebas experimentales que corroboran la función del microbioma intestinal en el SII provienen de experimentos de trasplantes de pacientes a ratones gnotobióticos, en los que se reproducen ciertos síntomas asociados al SII-E y al SII-D (tiempo de tránsito, sensación de dolor, permeabilidad intestinal...). Sin embargo, a falta de modelos robustos de animales con SII, se necesitan estudios en humanos para determinar las interacciones entre el microbioma intestinal y las vías patológicas específicas del ser humano. Los estudios en humanos sobre el SII suelen verse limitados por un muestreo transversal y una falta de estratificación por subgrupos de pacientes, lo que se traduce en una falta de concordancia entre los resultados obtenidos en una cantidad importante de estudios sobre el microbioma [4].
La influencia bien descrita del tránsito gastrointestinal en el microbioma intestinal aumenta todavía más la variabilidad de los estudios. Por otro lado, el SII, al igual que otros trastornos gastrointestinales crónicos, se caracteriza por periodos de remisión y reaparición de los síntomas, pero las muestras transversales no tienen en cuenta la variabilidad de la enfermedad a lo largo del tiempo. Por último, las diferencias inherentes en la fisiología del hospedador entre estudios en seres humanos y en animales han sido un obstáculo para el avance de nuestra comprensión de las funciones del mecanismo del microbioma intestinal en el SII. Los autores han llevado a cabo un estudio longitudinal en subgrupos de pacientes con SII, en el que han integrado mediciones multiómicas, incluido el metagenoma microbiano, el transcriptoma del huésped y el metiloma, con una evaluación de las funciones del anfitrión. Esto ha permitido identificar mecanismos específicos del subtipo de SII, inducidos por un metabolismo microbiano alterado, que correspondía a cambios simultáneos en la fisiología del huésped.
Las funciones de la microbiota intestinal están alteradas en caso de SII con diferencias entre el SII-E y el SII-D
Puede que el aumento de la producción de triptamina y la disminución de la transformación de los ácidos biliares estén implicadas en el SII-D
Es posible que un consumo excesivo de hipoxantina por parte de la microbiota y las células del hospedador influyan en el SII alterando el nivel de energía de las células epiteliales intestinales
En el estudio que nos ocupa, los autores han realizado un estudio prospectivo observacional longitudinal con análisis multiómico del microbioma intestinal y de las células del hospedador. Se compararon personas sanas con pacientes con SII-E y SII-D. En total, 77 pacientes proporcionaron al menos una muestra de heces (se obtuvo un total de 474 muestras de heces), y 42 participantes se sometieron a una sigmoidoscopia para obtener biopsias del colon. Para identificar los factores microbianos que determinan los síntomas específicos de los subtipos de SII, se realizaron una secuenciación metagenómica y un análisis metabolómico de las muestras de heces. El análisis metabolómico y las mediciones de citocinas se realizaron en las muestras de suero. Por último, se realizó una secuenciación del gen 16S y un análisis del metaboloma, del transcriptoma y del metiloma en las biopsias del colon.
Los autores hallaron diferencias en la composición y la diversidad de la microbiota intestinal entre las personas sanas y los pacientes SII-E o SII-D.
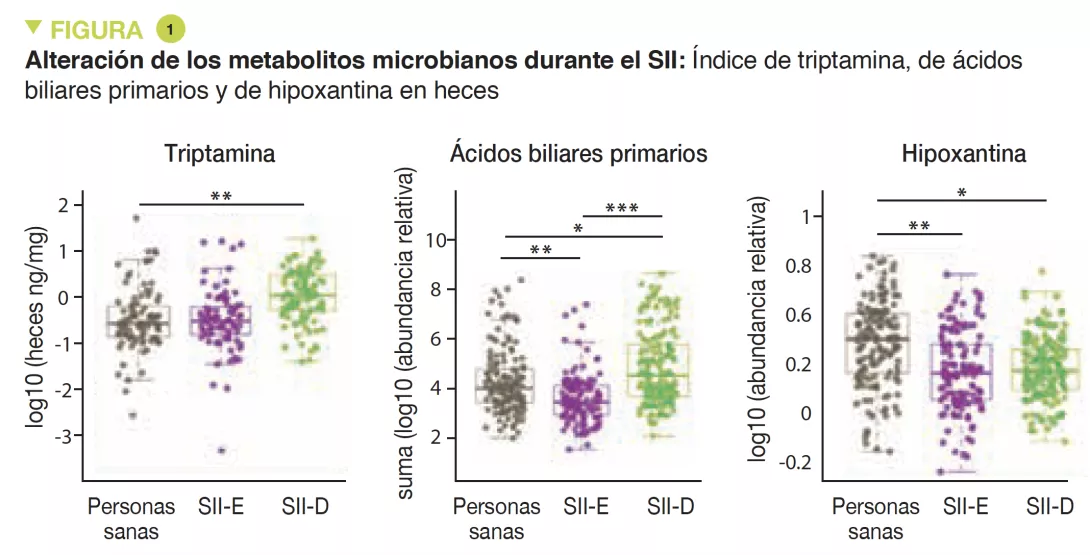
El análisis metabolómico de las heces reveló un aumento de la triptamina, un metabolito del triptófano producido por ciertas bacterias intestinales, en los pacientes con SII-D (Figura 1). Dado que la triptamina produce un efecto de aceleración del tránsito debido a una acción sobre el receptor de la serotonina 5-HT4, es posible que influya en el fenotipo de estos pacientes. De forma similar, la proporción de ácidos biliares primarios era más elevada en pacientes con SII-D, lo que indica una disfunción en su transformación por la microbiota. Los experimentos in vitro sugirieron que los ácidos biliares primarios aumentan la secreción cólica por lo que también es posible que influyan en el fenotipo.
Por último, la integración de los datos multiómicos reveló un posible nuevo mecanismo en el SII. Los resultados sugieren que en los pacientes con SII se produce un mayor deterioro de los nucleótidos de purinas, y en concreto de la hipoxantina, por parte de la microbiota y de las células del hospedador, lo cual provoca estrés en el colon. Es posible que esto cause una respuesta compensatoria con un aumento de la recuperación de purinas. Niveles bajos de nucleótidos de purinas pueden llevar a una disminución del estado de energía epitelial y de la capacidad para reparar la mucosa intestinal, lo que también podría contribuir a la fisiopatología del SII.
Estos datos sugieren que la microbiota intestinal influye en la fisiopatología del SII, con diferencias entre el SII-E y el SII-D. Por otro lado, estos resultados sugieren que también puede afectar una insuficiencia de nucleótidos de purinas, en concreto, a causa de un consumo excesivo de hipoxantina por parte de la microbiota y de las células del huésped. Estos resultados abren el camino a tratamientos que estimulen la producción de hipoxantina microbiana o que inhiban localmente la xantina oxidasa en el intestino.
Este estudio longitudinal multiómico demuestra por qué son interesantes los estudios longitudinales en humanos y pone de relieve las alteraciones funcionales de la microbiota durante el SII que, posiblemente, estén implicadas en su fisiopatología. Puede que estas nuevas pistas identificadas se conviertan a su vez en nuevas dianas terapéuticas.
Síntesis
Por el Dr François Trottein
Centro de Infección y de Inmunidad de Lille, Instituto Nacional de Salud e Investigación Médica (Inserm) U1019, CNRS UMR 9017, Universidad de Lille, Hospital Universitario de Lille, Instituto Pasteur de Lille, Francia

La nueva enfermedad del coronavirus 19 (SARS-CoV-2) (COVID-19) nos recuerda que las interacciones entre la microbiota intestinal y el sistema inmunitario son fundamentales durante las infecciones respiratorias víricas. Los virus respiratorios pueden desencadenar síntomas gastrointestinales, lo que pone de relieve la importancia del eje pulmón-intestino en la enfermedad. Estudios clínicos y modelos experimentales indican que las infecciones respiratorias víricas agudas alteran la composición y las funciones de la microbiota intestinal, un componente fundamental de la salud humana. En este artículo repasaremos estos importantes cambios y comentaremos las posibles causas de la disbiosis intestinal. Además, presentaremos las consecuencias de una disbiosis intestinal producida durante una infección sobre las consecuencias secundarias de la enfermedad. Por último, propondremos estrategias intervencionistas que podrían utilizarse para tratar la microbiota intestinal y reducir así la gravedad de la enfermedad respiratoria vírica.
viral siguen siendo una gran preocupación a escala mundial, dado que llevan aparejadas considerables consecuencias socioeconómicas y sanitarias. A pesar de las campañas de vacunación y de los medicamentos antivíricos, la gripe sigue siendo la más impactante, en términos de contagios (5-10 % de la población mundial) y muertes (> 600 000 al año). Además de las epidemias anuales, la gripe es la causante de pandemias cada 10-20 años, de las cuales la más famosa es la conocida como «gripe española», de 1918 a 1919 (con 50 millones de defunciones) y la más reciente, la pandemia de gripe H1N1 en 2009 (con una sobremortalidad de 400 000 defunciones) [1]. Las pandemias no se limitan a la gripe, como ha mostrado la COVID- 19 [2]. Es especialmente preocupante el aumento de la frecuencia de pandemias que se han producido en las últimas décadas, un fenómeno explicado en parte por los cambios climáticos y por la actividad humana, en general.
La microbiota intestinal desempeña una función clave en el mantenimiento de la salud humana y es decisiva para el control de las infecciones (respiratorias) [3, 4]. Existen muchos factores que pueden alterar la diversidad y la composición de la microbiota intestinal, y conducir a una disbiosis. Entre ellos, situaciones patológicas como infecciones, trastornos inflamatorios o metabólicos crónicos pueden desencadenar una alteración profunda de la composición y el funcionamiento de la microbiota intestinal. Las modificaciones en las comunidades bacterianas del intestino pueden influir en la evolución de la enfermedad, incluso en órganos alejados (por ejemplo los pulmones) [3, 4], como han demostrado los experimentos de transferencia de microbiota disbiótica. A continuación, resumimos los efectos de las infecciones respiratorias agudas de origen viral en la microbiota intestinal.
Ciertos hallazgos clínicos parecen indicar que durante la infección por gripe se produce una disbiosis de la microbiota. La abundancia relativa de Actinobacteria, Erysipelotrichea, Clostridia y de productores benignos de butirato (familias Lachnospiraceae y Ruminococcaceae) se reduce en pacientes con H1N1. Por otro lado, se desarrollan patógenos oportunistas como Escherichia-Shigella y Prevotella [5]. En modelos experimentales (con ratones) también se produce una disbiosis intestinal transitoria, con un pico en los 5-7 días posteriores a la infección [6-9]. Los cambios se producen en los niveles taxonómicos, sin modificaciones en la diversidad alfa. La infección reduce el crecimiento de las bacterias que favorecen un buen estado de salud, como Lactobacilli, Bifidobacteria y bacterias filamentosas segmentadas. Numerosas especies capaces de transformar por fermentación fibras alimentarias en ácidos grasos de cadena corta (AGCC) se ven afectadas. Por ello, la producción de AGCC se desploma cuando hay infección por gripe [9]. Los bajos niveles de comensales benignos se asocian a la proliferación de bacterias nocivas como Gammaproteobacteria (Escherichia coli) y bacterias que dañan la mucosa como Verrucomicrobia (Akkermansia genus) y Ruminococcus. La infección por SARSCoV- 2 también produce alteraciones en la microbiota intestinal de los pacientes, por ejemplo, una menor abundancia de productores de butirato, como varios géneros bacterianos de las familias Ruminococcaceae y Lachnospiraceae (Roseburia) [5, 10]. Por otro lado, se ha observado una abundancia significativamente mayor de bacterias patógenas oportunistas como Streptococcus (clase Bacilli), Rothia y Actinomyces. Hay que señalar que, en pacientes con COVID-19, también se ha descrito una proliferación de patógenos micóticos oportunistas (géneros Aspergillus y Candida) [11]. En conjunto, las respiraciones infecciosas de origen viral conducen a una reducción de bacterias comensales benignas y al enriquecimiento de agentes patógenos nocivos oportunistas. Las posibles modificaciones en la estructura, la composición y la actividad funcional de la microbiota intestinal pueden ser biomarcadores de la gravedad de la enfermedad.
Es probable que existan varias causas que conduzcan a una disbiosis intestinal durante infecciones respiratorias víricas, como la liberación de citocinas inflamatorias y una reducción de la absorción de comida (Figura 1). La infección provoca una pérdida de peso sustancial debido a la falta de apetito. Experimentos consistentes en alimentar a ratones en pares indican claramente que un rápido descenso en la ingesta de alimento reproduce los cambios de la microbiota intestinal observados durante una gripe [8]. La evidencia reciente sugiere que el FNTa influye en la disbiosis asociada a la inapetencia durante una infección respiratoria vírica [12]. Los interferones de Tipo I y II, que son fundamentales para la respuesta antivírica del huésped, también desempeñan una función en la disbiosis intestinal [5,6].
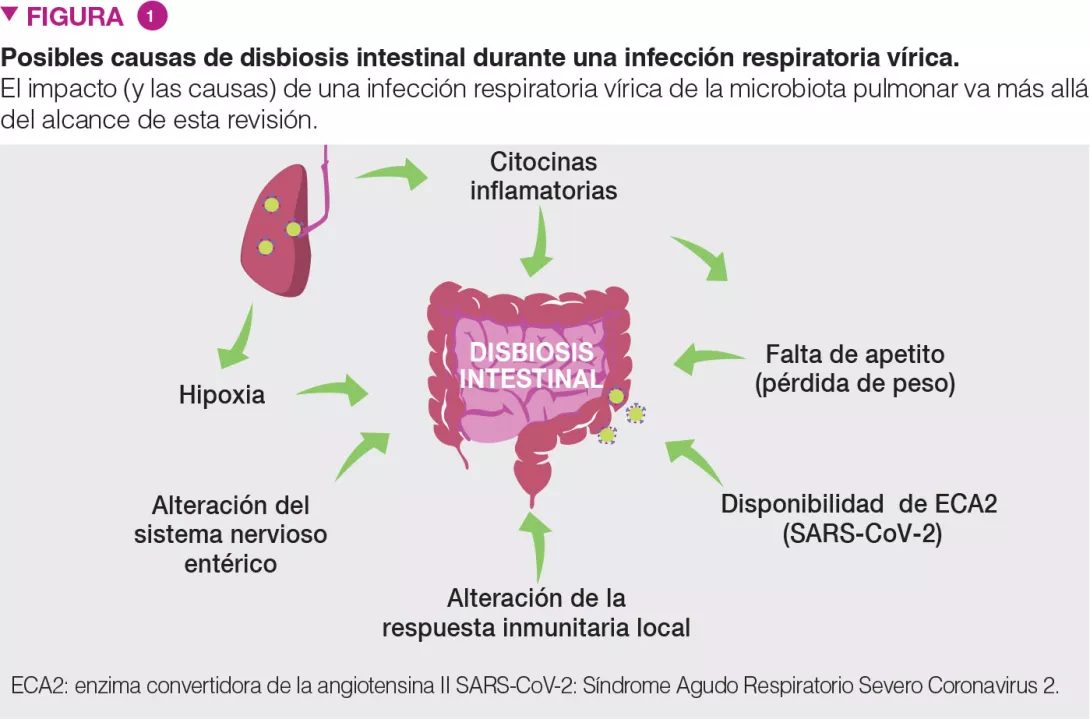
Además, es probable que, en la disbiosis intestinal, intervengan la hipoxia (un síntoma de infección respiratoria vírica aguda), las alteraciones en el sistema nervioso entérico y una respuesta inmunitaria local alterada [13] (Figura 1). En el caso de la COVID-19, además de estos mecanismos, es posible que la replicación viral local influya en la disbiosis intestinal. La enzima convertidora de angiotensina II (ECA2), el receptor del SARS-CoV-2, es determinante para mantener el ecosistema microbiano del intestino. Dada la falta de ACE2 disponible durante la infección por SARS-CoV-2, es de esperar que este factor influya en la composición y el funcionamiento de la microbiota intestinal [13].
La disbiosis intestinal durante una infección respiratoria vírica tiene consecuencias locales y distales, y puede ser un factor clave que contribuya a la gravedad de la enfermedad y a una evolución mortal (Figura 2). Los pacientes con infección respiratoria de origen viral pueden presentar síntomas similares a los de la gastroenteritis, como dolor abdominal, náuseas, vómitos y diarrea. La alteración de la microbiota intestinal puede explicar estos trastornos. También es probable que la microbiota alterada, con la aparición de patobiontes y bacterias que dañan el moco intestinal, intervenga en la inflamación y la alteración de la integridad de la barrera del intestino [6]. A su vez, la permeabilidad de la barrera intestinal puede aumentar las concentraciones de endotoxinas en la sangre, provocando, en última instancia, inflamación, proliferación de citocinas y disfunciones pulmonares [14]. Las infecciones respiratorias de origen viral pueden desembocar en infecciones entéricas secundarias y septicemia. La disbiosis intestinal (y el descenso de AGCC) pueden ser importantes en este contexto. En efecto, los AGCC son decisivos en la homeostasis, la integridad de la barrera intestinal y el control de los patógenos entéricos [15]. Nuestros datos recientes muestran que, además de los trastornos locales, una disbiosis intestinal puede obstaculizar, a distancia, las defensas del huésped en los pulmones [9] (Figura 2). En buenas condiciones de salud, la microbiota intestinal defiende a los pulmones, de manera remota, contra infecciones bacterianas, en parte reforzando la actividad bactericida de los macrófagos pulmonares [16].
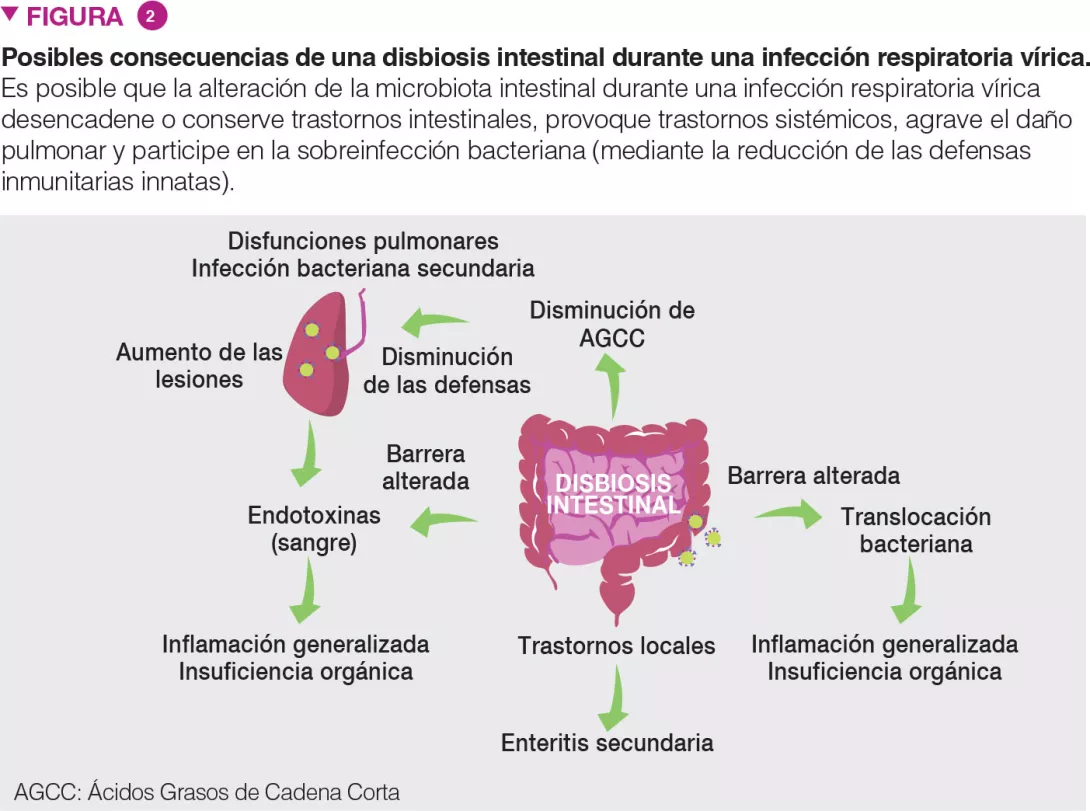
Durante una gripe, este eje se ve alterado y bacterias oportunistas invaden los pulmones, produciendo una sobreinfección bacteriana, que es una de las causas principales de muerte en las epidemias y pandemias de gripe [1]. Hemos mostrado que una menor producción de acetato (el AGCC principal) por la microbiota intestinal es parcialmente responsable de este efecto [9]. Conjuntamente, la disbiosis puede influir en las señales y síntomas gastrointestinales y pulmonares (así como en la mortalidad general) de las infecciones respiratorias víricas. ¿Podemos usar las vías intestino-pulmones como base para controlar mejor la gravedad y la tasa de mortalidad de las infecciones respiratorias víricas?
La microbiota intestinal es vital para las defensas de los pulmones frente a la infecciones respiratorias, por lo que revisten un gran interés estrategias intervencionistas dirigidas a los comensales intestinales, para armar, de manera preventiva, a los pulmones frente a patógenos víricos y proteger a la microbiota de las perturbaciones asociadas con las infecciones víricas (Figure 3). Esto es particularmente importante en personas con un desequilibrio general de microbiota intestinal, como las personas mayores y pacientes con comorbilidades, quienes, como sabemos, están más expuestos a las infecciones.
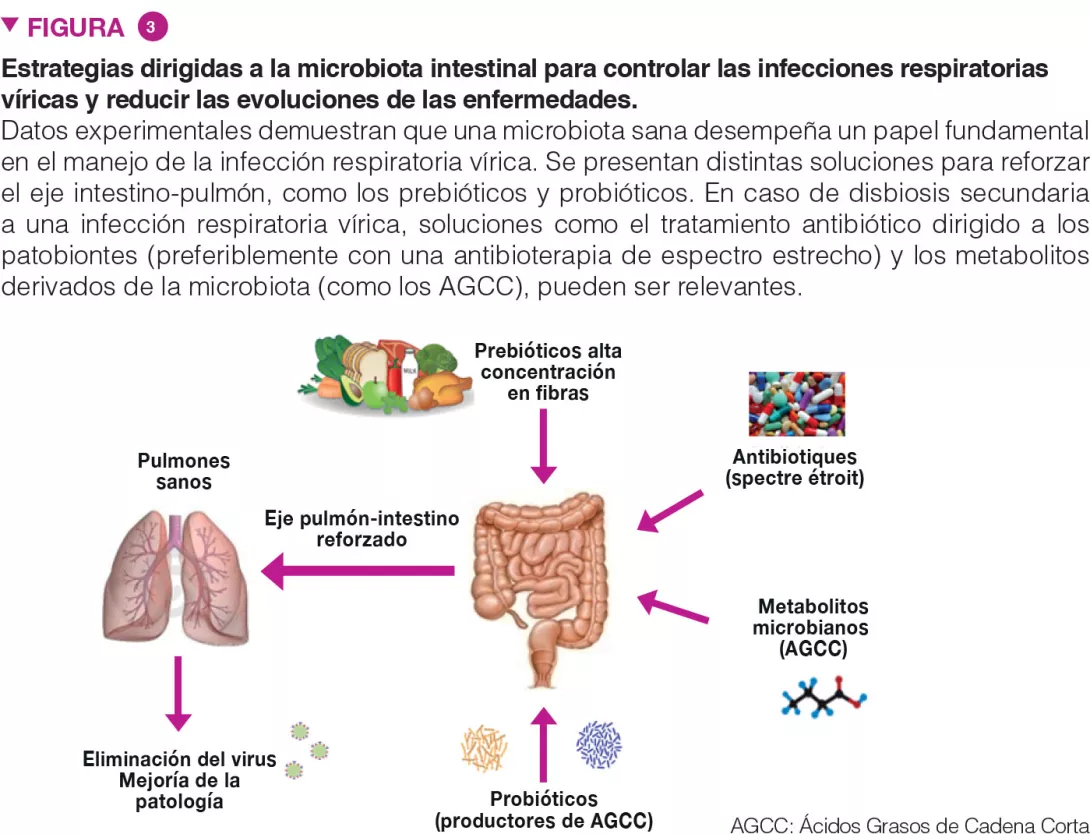
Pueden ser relevantes soluciones como (i) complementos alimenticios destinados a nutrir a nuestros microorganismos beneficiosos (como fibras prebióticas) y (ii) bacterias indígenas (conocidas como probióticos) para reponer los microorganismos benignos que le falten a nuestro intestino y optimizar sus funciones metabólicas. Estas estrategias, especialmente si se personalizan (es decir, según el análisis de la microbiota intestinal en la población «de riesgo»), pueden mejorar los parámetros clínicos y acelerar la recuperación de pacientes que padezcan infecciones respiratorias víricas agudas. Los probióticos, como algunas bifidobacterias y Lactobacillus spp, pueden reducir la gravedad de la gripe, mediante mecanismos aún desconocidos [17]. Un estudio reciente, de gran importancia en la actualidad, demostró que la bacterioterapia oral, junto con la terapia habitual con fármacos, arrojaba pistas prometedoras sobre el manejo de pacientes de COVID-19 [18].
El eje intestino-pulmón es importante durante las infecciones virales de las vías respiratorias. La alteración de la microbiota intestinal influye en complicaciones secundarias de esta enfermedad. Por ello, la microbiota intestinal representa un objetivo que puede limitar la gravedad de la enfermedad. Las soluciones para el abordaje de la enfermedad incluyen complementos alimenticios (como fibras prebióticas) y probióticos.
Síntesis
Por la Pr. Emily P. Balskus
Departamento de Química y Biología Química, Universidad de Harvard, Cambridge, EE. UU.
La microbiota intestinal transforma las estructuras químicas de los compuestos ingeridos, como los fármacos de molécula pequeña administrados por vía oral. Este metabolismo, que puede variar sustancialmente entre pacientes, afecta a la eficacia del fármaco de forma positiva o negativa, y puede influir en su toxicidad. En los últimos 10 años, se ha reconocido cada vez más su posible efecto sobre la variabilidad interindividual en la respuesta a fármacos. A continuación repasaremos este tema, centrándonos en los últimos avances y en su posible impacto futuro sobre el manejo de los pacientes y el descubrimiento de medicamentos.
Los billones de microorganismos que pueblan el intestino humano poseen un número mucho mayor de genes que el genoma del propio huésped. Muchos de estos genes codifican a catalizadores de proteínas (enzimas), que permiten a la microbiota intestinal realizar un amplio abanico de reacciones químicas, ampliando la bioquímica asociada al cuerpo humano. Una característica principal del metabolismo microbiano intestinal es su variabilidad. Así, en una comunidad, tanto varía la composición de la microbiota como las capacidades metabólicas de cada individuo. Conforme identificamos nuevas asociaciones entre la microbiota intestinal y la salud, se hace más importante caracterizar las transformaciones metabólicas microbianas a nivel molecular.
Una de las actividades importantes asociadas a la microbiota intestinal es la capacidad de modificar químicamente las estructuras de los fármacos de molécula pequeña [1]. Los fármacos administrados por vía oral se topan con las bacterias intestinales antes de ser absorbidos en el intestino delgado, o bien en el intestino grueso si su biodisponibilidad por vía oral es baja. Los fármacos administrados por vía oral o inyectados, o sus metabolitos, también llegan a la microbiota si son excretados por la bilis en el intestino. Dado que la actividad farmacológica de un fármaco deriva directamente de su estructura química, el metabolismo microbiano puede tener una gran influencia en la acción del fármaco.
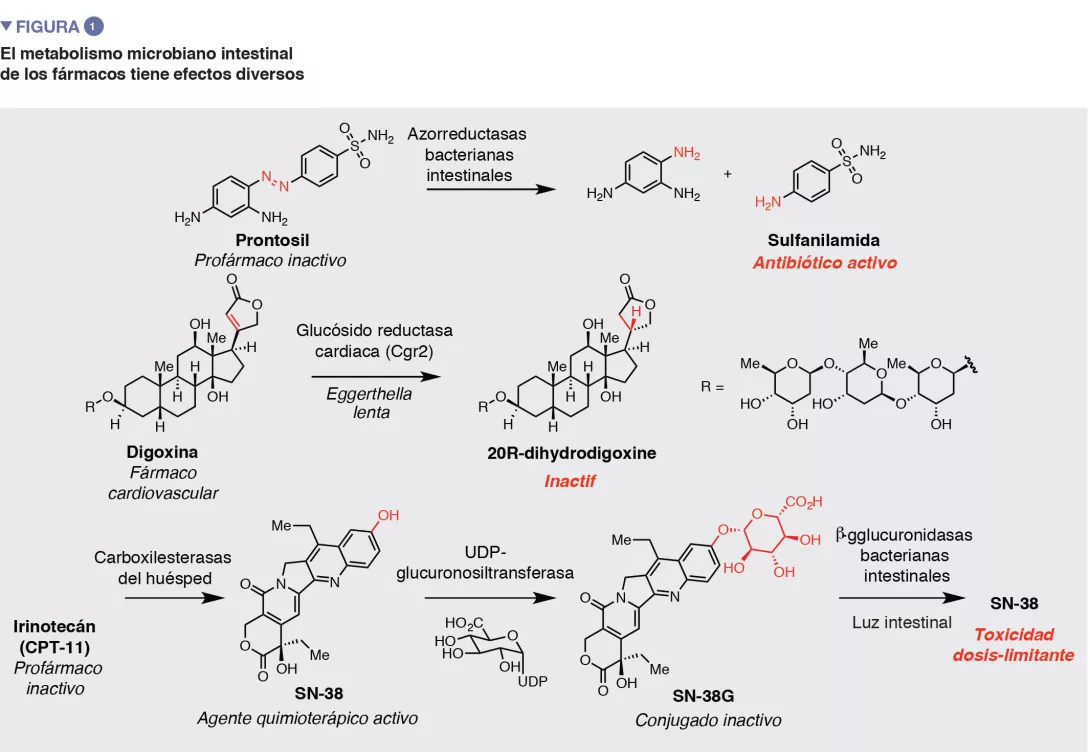
El metabolismo microbiano intestinal tiene distintas consecuencias sobre la acción y eficacia de los medicamentos (Figura 1). Como ilustran los ejemplos sobre fármacos azoicos, el metabolismo microbiano de los «profármacos» (precursores inactivos) puede ser necesario para generar el agente farmacológico activo. Este descubrimiento ha inspirado el diseño de nuevas estrategias para la liberación selectiva de fármacos en el intestino grueso, que dependen de las actividades metabólicas microbianas.
La metabolización que realiza la microbiota intestinal también puede afectar de forma negativa a la actividad de los fármacos, al alterar las interacciones con las dianas terapéuticas del huésped. Un ejemplo es la digoxina, un medicamento para tratar algunas enfermedades cardiacas, hecho a base de un producto natural. En el 5-10 % de los pacientes, la microbiota intestinal reduce el anillo de lactona a, b-insaturada de la digoxina para dar dihidrodigoxina. Esta sutil modificación, realizada por la bacteria intestinal Eggerthella lenta, reduce en gran medida la afinidad de unión con la diana de la digoxina (Na+/K+ ATPasa), lo que conlleva una pérdida de eficacia [2]. Otro buen ejemplo es el tratamiento de primera línea de Parkinson, la levodopa. La conversión de levodopa a dopamina por enzimas en el cerebro del huésped es fundamental para aliviar los síntomas. El metabolismo microbiano intestinal de la levodopa también produce dopamina [3, 4]. Dado que la dopamina generada en la periferia no puede atravesar la barrera hematoencefálica, este efecto podría reducir la cantidad de levodopa que llega al cerebro.
Por último, además de reducir la actividad, las modificaciones químicas creadas por microbios intestinales pueden producir toxicidad no deseada. Por ejemplo, el metabolismo microbiano intestinal ha sido implicado en la letalidad asociada a la administración concomitante del medicamento antivírico brivudina con antineoplásicos de fluoropirimidina. Este desenlace se atribuye a la metabolización microbiana intestinal de sorivudina a bromoviniluracilo. Este metabolito inhibe una enzima clave del huésped implicada en la inactivación del 5-fluorouracilo, lo que aumenta su concentración a niveles mortales.
Los estudios sobre el metabolismo microbiano intestinal de los fármacos comenzaron hace más de 80 años, con el descubrimiento de que uno de los primeros antibi ticos, el prontosil, un azocompuesto inactivo en cultivos bacterianos pero eficaz in vivo, sufré a una reducción por la microbiota intestinal para producir el agente activo sulfanilamida. Desde entonces,
se han descubierto otros ejemplos, a menudo al detectar una variabilidad en la eficacia o la toxicidad de un tratamiento. Sin embargo, a pesar de estos hallazgos, estas relaciones siguen sin tenerse en cuenta a la hora de desarrollar o administrar fármacos.
Una característica importante del metabolismo microbiano intestinal de los fármacos es su variabilidad entre pacientes. Este fenómeno se debe a la variabilidad de la microbiota intestinal. A pesar de que ciertas actividades metabólicas se encuentran en muchos organismos, otras están realizadas por un subconjunto pequeño y de escasa abundancia en la comunidad intestinal. El metabolismo puede variar entre cepas de la misma especie, e incluso entre bacterias estrechamente relacionadas puede haber grandes diferencias en los genomas. Por ese motivo, quizá no sea sorprendente que la composición de la comunidad intestinal suela ser un predictor pobre de su metabolismo, y que el metabolismo de algunos medicamentos sea rápido en ciertas personas e inexistente en otras. Es posible que esta variabilidad tenga consecuencias importantes, pero aún no la comprendemos totalmente en pacientes que toman varios medicamentos de molécula pequeña.
Para comprender completamente el metabolismo microbiano intestinal de los fármacos es necesario vincular las actividades individuales con los microbios, genes y enzimas. Suele ser necesario identificar a microbios específicos metabolizadores de fármacos que permitan realizar estudios farmacodinámicos posteriores. Esto puede lograrse examinando cepas disponibles de microbios intestinales, o bien aislando directamente organismos metabolizadores desde muestras de microbiota intestinal compleja. Luego, es necesario relacionar las transformaciones observadas con genes y enzimas. Esto es fundamental para estudiar el metabolismo en comunidades intestinales complejas, ya que los genes que codifican enzimas metabólicas permiten la detección y la predicción de actividades individuales en genomas microbianos y datos de secuenciación del microbioma. La vinculación del metabolismo de los medicamentos con los genes microbianos puede conseguirse de varias maneras, como el análisis racional de genomas para encontrar enzimas con las capacidades catalíticas necesarias, la secuenciación de ARN para identificar genes que aumentan específicamente en respuesta a un fármaco, y mediante la genómica comparativa, que permite asociar los genes con las capacidades metabólicas.
Hasta 2019, se comunicaron unos 60 ejemplos de metabolismo microbiano intestinal de fármacos. Dos estudios recientes utilizaron técnicas de cribado analítico masivo y de experimentación para realizar investigaciones a gran escala sobre el metabolismo microbiano intestinal de fármacos, lo que permitió ampliar el abanico de transformaciones conocidas. Goodman y colaboradores examinaron a 76 cepas bacterianas aisladas del intestino humano por su capacidad para metabolizar 271 medicamentos de molécula pequeña y descubrieron que dos tercios de estos fármacos habían sido modificados por al menos un organismo [8]. El grupo de Donia realizó un estudio análogo de 575 fármacos, utilizando una muestra del microbioma intestinal de un paciente ex vivo y halló 45 transformaciones nuevas [6]. Estos hallazgos sugieren que el número de fármacos susceptibles de ser metabolizados puede ser mayor de lo que se sabía hasta ahora; sin embargo, la gran mayoría de estas actividades recién descubiertas aún no se ha confirmado in vivo, por lo que sigue sin conocerse su relevancia para los pacientes.
Las reacciones químicas del metabolismo microbiano intestinal de los fármacos, que tienden a ser reductivas e hidrolíticas, suelen ser diferentes de las transformaciones del huésped, que implican una oxidación de los fármacos y una conjugación con metabolitos más polares para facilitar la excreción. El metabolismo microbiano suele tener efectos opuestos en la disponibilidad de los medicamentos, lo que prolonga su circulación en el cuerpo. Sin embargo, las transformaciones microbianas de los fármacos no tienen por qué ser diferentes para influir en la acción del fármaco. Estudios recientes sobre el fármaco antiviral brivudina sugieren que dichas actividades pueden influir en la farmacocin tica del fármaco incluso aunque sean identicas a las del metabolismo del huésped [5].
Una vez que se averigua que la microbiota intestinal transforma un medicamento de molécula pequeña, el siguiente paso lógico es investigar cómo se puede controlar esta actividad, tanto para evaluar las consecuencias del metabolismo sobre la acción del fármaco como para mejorar el tratamiento del paciente, si se observara que este metabolismo es perjudicial. Se han utilizado varios métodos para lograr este objetivo. Con modelos animales gnotobióticos (animales axénicos colonizados de forma controlada con una microbiota específica), se pueden comparar comunidades que contienen bien cepas intestinales metabolizadoras del fármaco o mutantes que carezcan de estas actividades específicas. En los estudios del laboratorio de Goodman sobre brivudina se ilustra muy bien la utilidad de este enfoque [5].
Sin embargo, la manipulación genética supone un desafío en comunidades microbianas complejas nativas, lo que nos conduce a evaluar enfoques alternativos. Una posible estrategia es aprovechar el conocimiento de la fisiología bacteriana del intestino para orientar la manipulación del entorno intestinal mediante intervenciones alimenticias. Por ejemplo, en el caso de la digoxina, Turnbaugh y sus colaboradores observaron que la presencia de la L-arginina disminuye el metabolismo del fármaco por E. lenta [2]. Después, demostraron que al administrar una alimentación rica en proteínas a ratones axénicos colonizados con E. lenta se reducía la inactivación del fármaco in vivo.
Otra estrategia interesante es identificar a moléculas pequeñas que inhiben la actividad de las enzimas bacterianas intestinales metabolizadoras de fármacos, como descubrió el laboratorio de Redinbo en sus estudios sobre el metabolismo del irinotecán. El irinotecán es un profármaco metabolizado por células del huésped en inhibidor activo de la topoisomerasa SN- 38. El huésped metaboliza la SN-38 por glucoronidación, y produce un conjugado inactivo (SN-38G). Este metabolito se excreta en el intestino, donde las enzimas b-glucoronidasa (GUS) bacterianas intestinales eliminan el glucurónido. Esta reactivación produce una toxicidad dosis-limitante en el tubo gastrointestinal. El grupo Redinbo utilizó el cribado analítico masivo para identificar a los inhibidores selectivos de enzimas GUS bacterianas intestinales, y descubrió que prevenían los graves efectos secundarios causados por el irinotecán en un modelo murino [9]. Trabajos posteriores revelaron que estos compuestos aumentaban la eficacia del irinotecán al limitar su toxicidad [10]. En conjunto, estos trabajos han proporcionado una prueba fascinante para abordar de forma terapéutica el metabolismo bacteriano intestinal y ha impulsado nuevas iniciativas para el descubrimiento de inhibidores.
Se calcula que el 70 % de la diversidad microbiana intestinal aún no se ha cultivado, lo que dificulta la caracterizaciòn de sus actividades. Donia y colaboradores
utilizaron metagen mica funcional, que consiste en introducir ADN aislado directamente de una microbiota compleja en un huésped heter logo, para identificar una enzima bacteriana intestinal que metaboliza la hidrocortisona [6]. Recientemente, también se han descubierto enzimas que metabolizan el colesterol en bacterias intestinales no cultivadas, correlacionando la presencia de genes microbianos en microbiomas con datos metabol micos. Ambas estrategias podrían ser útiles para investigar el metabolismo
de fármacos en microorganismos no cultivados.
El desarrollo exitoso de inhibidores de GUS como candidatos terapéuticos demuestra que gracias a una comprensión molecular del metabolismo microbiano intestinal de los fármacos los pacientes podrían salir beneficiados. Otro ámbito que podría transformarse gracias a este conocimiento es la medicina de precisión. Si se comprende cómo los microbios intestinales metabolizan medicamentos específicos, es posible que los médicos puedan utilizar los datos de secuenciación del microbioma o pruebas diagnósticas a partir de la microbiota para decidir cuándo y cómo recetar determinados medicamentos.
Un mayor reconocimiento del valor del metabolismo microbiano intestinal de los medicamentos podría influir incluso en el propio proceso de descubrimiento de los medicamentos. Los farmacéuticos clínicos suelen evitar grupos funcionales que se sabe que son transformados por bacterias intestinales, debido a asociaciones previas con toxicidad y efectos secundarios. Cabe imaginar que se descubrirán transformaciones nuevas e imprevistas en las primeras etapas del desarrollo de un fármaco estudiando los microbios intestinales individuales o las comunidades complejas pacientes para averiguar el metabolismo ex vivo, del mismo modo en que se suele probar el metabolismo de los fármacos candidatos por las enzimas del huésped. Las diferencias en la composición y las funciones de la microbiota intestinal entre modelos animales y humanos deberían tenerse en cuenta en estudios clínicos y preclínicos. Por último, sería recomendable incorporar a los ensayos clínicos la recogida y análisis de muestras de microbioma para evaluar el metabolismo de los fármacos. Es posible que correlacionar el metabolismo con las diferencias de toxicidad y eficacia ayude a interpretar los resultados de dichos ensayos y a definir las poblaciones de pacientes destinatarios.
En resumen, en la última década hemos vivido grandes avances en nuestra comprensión de los mecanismos moleculares subyacentes al metabolismo microbiano intestinal de los fármacos y sus consecuencias sobre la eficacia de estos. Es necesario realizar más estudios para explorar este campo de investigación apasionante y hacer avanzar la medicina de precisión y el descubrimiento de fármacos.
Síntesis
Por la Dra. Dorota Czerucka
iBiología médica, Equipo de ecosistemas e inmunidad, Centro científico de Mónaco, Mónaco
Los seres vivos han evolucionado desde hace millones de años en entornos complejos ocupados por ecosistemas microbianos, lo que los ha llevado a forjar relaciones simbióticas reguladas por el sistema inmunitario. Las nuevas técnicas de secuenciación han revolucionado nuestros conocimientos y nos han permitido poner de relieve que cada individuo alberga una microbiota propia, así como su función en la fisiología del anfitrión y en numerosa s enfermedades, como las infecciones. El diálogo entre la microbiota intestinal y el sistema inmunitario comienza desde la vida fetal. La organización específica de la microbiota –separada del anfitrión por una monocapa celular– supone un notable desafío para el sistema inmunitario, cuya función es reconocer lo «ajeno» como un posible signo de infección para iniciar las respuestas inmunitarias en cadena. Por consiguiente, los intercambios continuos con la microbiota tienen un impacto considerable en el sistema inmunitario del anfitrión. La respuesta inmunitaria, que debe tolerar a la microbiota, también afecta a la composición y la función de la microbiota. Así, los intercambios bidireccionales y constantes entre estas dos entidades moldean tanto la inmunidad del anfitrión como la microbiota intestinal para proteger de infecciones y de numerosas patologías.
La microbiota intestinal es la primera barrera para proteger a la mucosa intestinal frente a los patógenos. Este ecosistema complejo habita de manera estable el tracto gastrointestinal y limita el acceso a los nichos del intestino, así como a los nutrientes necesarios para la multiplicación de bacterias exógenas, mediante el fenómeno de la «resistencia a la colonización » [1] (Figura 1). Los enterocitos, que proporcionan una barrera física entre la luz intestinal y el anfitrión, absorben el agua y los nutrientes, y segregan péptidos antimicrobianos PAM (RegIIIg, b-defensins y cathelicidin) [2]. Gracias al reconocimiento de patrones moleculares asociados a los microorganismos (Microbe-Associated Molecular Patterns, MAMPs) por receptores específicos (como los Toll-Like-Receptors, TLR), estas células serán capaces de transducir la señal en citocinas y quimiocinas para señalar una infección y reclutar células inmunitarias (Figura 2). Las células caliciformes –secretoras de mucosidad– y las células M tienen la capacidad de hacer pasar a la luz intestinal antígenos intactos y capturados al azar provenientes de bacterias comensales, de patógenos o de antígenos alimentarios. A continuación, las células dendríticas (CD) los procesarán y los presentarán a las células de la inmunidad adaptativa. Esta función es fundamental para la tolerancia intestinal y la inducción de respuestas inmunitarias en las mucosas [2]: así, se produce un equilibrio permanente entre las respuestas pro- y antinflamatorias (Figura 2). Esto se ha demostrado en concreto en modelos de ratones con colitis inducida o con falta de receptores TLR: la ausencia de microbiota o de reconocimiento de la misma, reduce la proliferación de células epiteliales intestinales o la reparación de la barrera [2]. Por último, la mucosidad también proporciona una protección, al capturar los PAM que mantienen a los patógenos alejados del epitelio. En un modelo de ratones con carencia de Muc2 (gen codificante de una de las proteínas que componen la mucosidad), se observa un aumento de la translocación de bacterias comensales, y estos animales acaban padeciendo enfermedades inflamatorias intestinales [3].
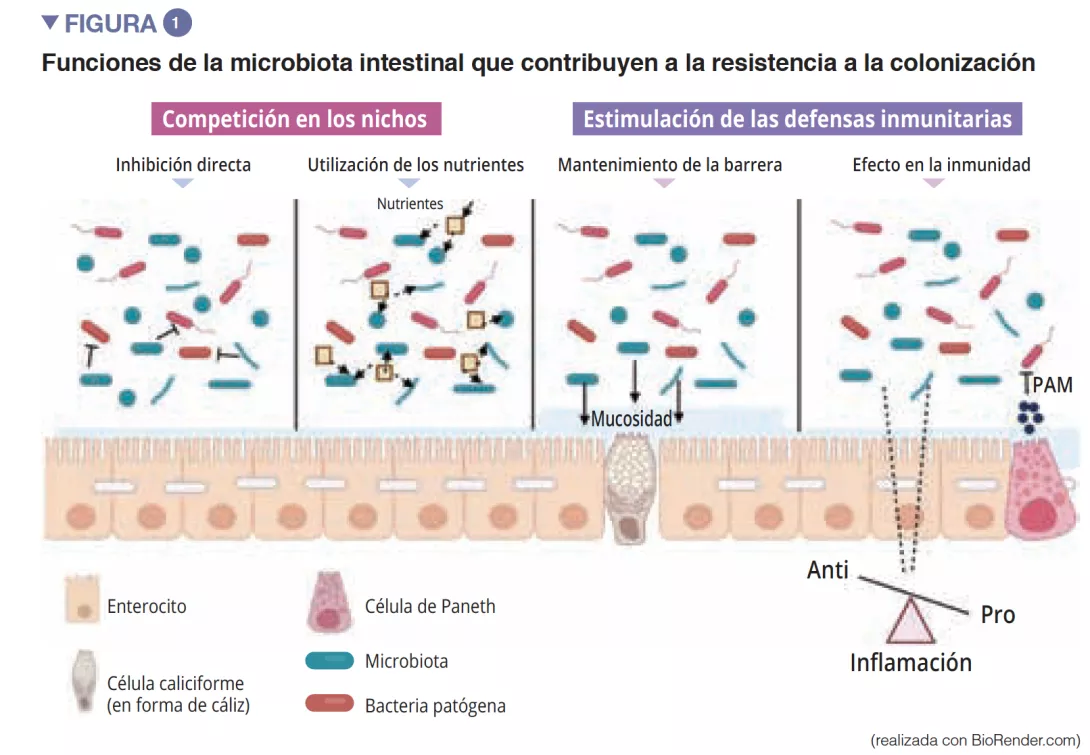
De entre los actores del sistema inmunitario innato que participan en la homeostasis intestinal, las células presentadoras de antígenos (CPA) como los macrófagos (Mj)y las CD, desempeñan una función fundamental. Los Mj y las CD sintetizan la IL-10 y favorecen así la diferenciación en Treg [4] además de la maduración de los linfocitos Th17 mediante la implicación de bacterias comensales: las bacterias segmentadas filamentosas (Segmented Filamentous Bacteria, SFB). Estas tienen la particularidad de adherirse a las células epiteliales intestinales y producir una estimulación activa del sistema inmunitario [5] (Figura 3). Un estudio ha demostrado que la colonización de ratones con estas SFB induce la diferenciación de Th17 y ejerce así una protección frente a Citrobacter rodentium (equivalente en ratones de los EPEC y EHEC). Es posible que esta protección se pueda atribuir a la capacidad de las SFB para estimular la síntesis de IL-22 por Th17, citocina conocida por estimular la síntesis de los PAM [6]. Las CD, por su parte y gracias a una extensión de sus dendritas entre las células epiteliales, son capaces de fagocitar bacterias presentes en la luz intestinal. A continuación, estas bacterias son transportadas hasta los ganglios linfáticos mesentéricos para inducir la producción de IgA secretadas por los plasmocitos [1]). Las células linfoides innatas (Innate Lymphoid Cells, ILC) también desempeñan una función importante en la homeostasis intestinal, relacionada con su capacidad para iniciar y orientar las respuestas inmunitarias intestinales. Más concretamente, las ILC de tipo 3 (ILC3) ocupan un lugar especial en la interacción con la microbiota intestinal. A través de la síntesis de IL-22, estas células estimulan la producción de mucosidad y de PAM, además de la secreción de quimiocinas y el reclutamiento de células polimorfonucleares (PMN) (Figura 2) [1].
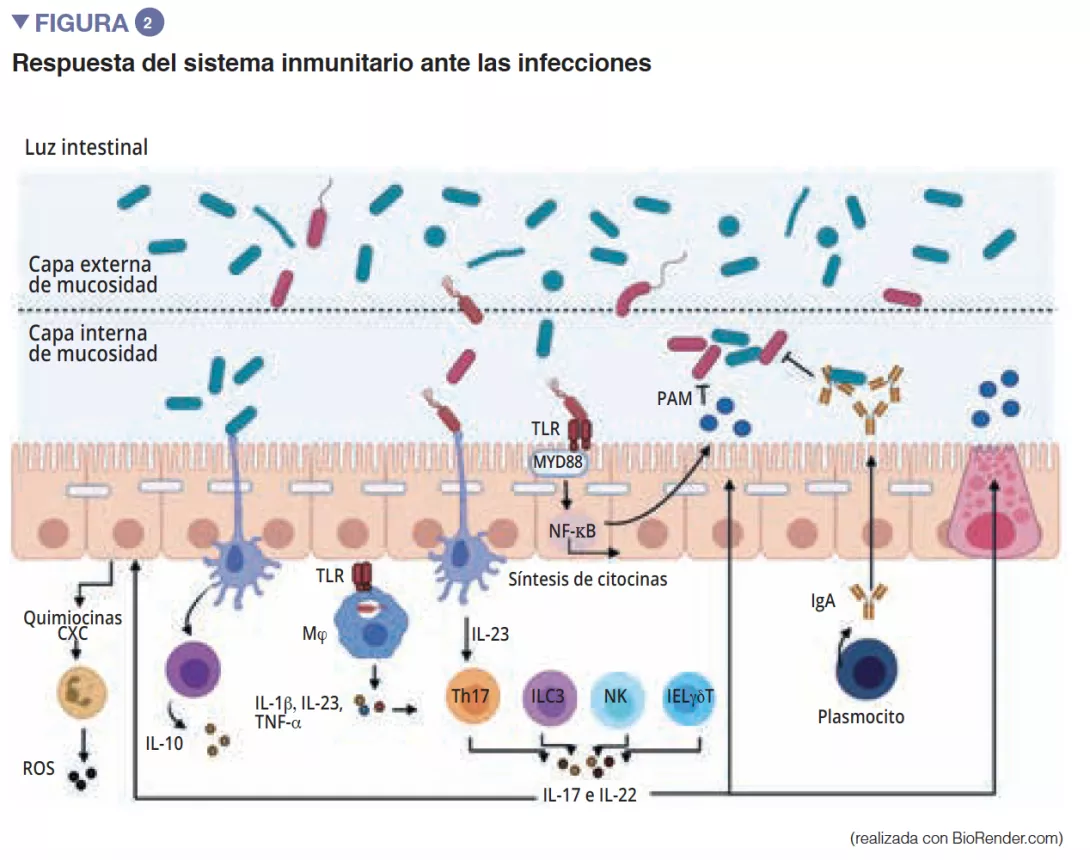
La maduración final del sistema inmunitario adaptativo se caracteriza por el asentamiento en la mucosa intestinal de linfocitos maduros efectores T proinflamatorios (Th17), antinflamatorios (Treg) y linfocitos B (Figura 2). Al margen de los efectos sobre los macrófagos y la diferenciación de los Th17, las SFB estimulan además el desarrollo de los órganos linfoides y participan en la diferenciación de los linfocitos B en células plasmáticas productoras de IgA que contendrán las bacterias patógenas en la mucosidad [5]. Otras bacterias comensales pueden estimular las respuestas inmunitarias adaptativas: una mezcla de 17 cepas de Clostridia, aislada de una muestra fecal humana e introducida en un ratón, induce una respuesta antinflamatoria al estimular los Treg [7]. Faecalibacterium prausnitzii también se ha identificado por su acción antinflamatoria in vitro e in vivo al actuar sobre el factor NF-kB, las CD y Mj, que segrega IL-10 y favorece la diferenciación de los Treg en detrimento de los Th17 [8]. De entre las Bacteroidetes, también se ha observado que Bacteroides fragilis y B. thetaiotaomicron ejercen una actividad antinflamatoria. B. fragilis sintetiza un polisacárido A (PSA) que suprime la producción proinflamatoria de IL-17, y estimula la secreción antinflamatoria de IL-10 Figura 3). En un modelo específico de colitis inducida por Helicobacter hepaticus, PSA estimula el desarrollo de los órganos linfoides, estimula el linfocito Treg y protege a los ratones [9].
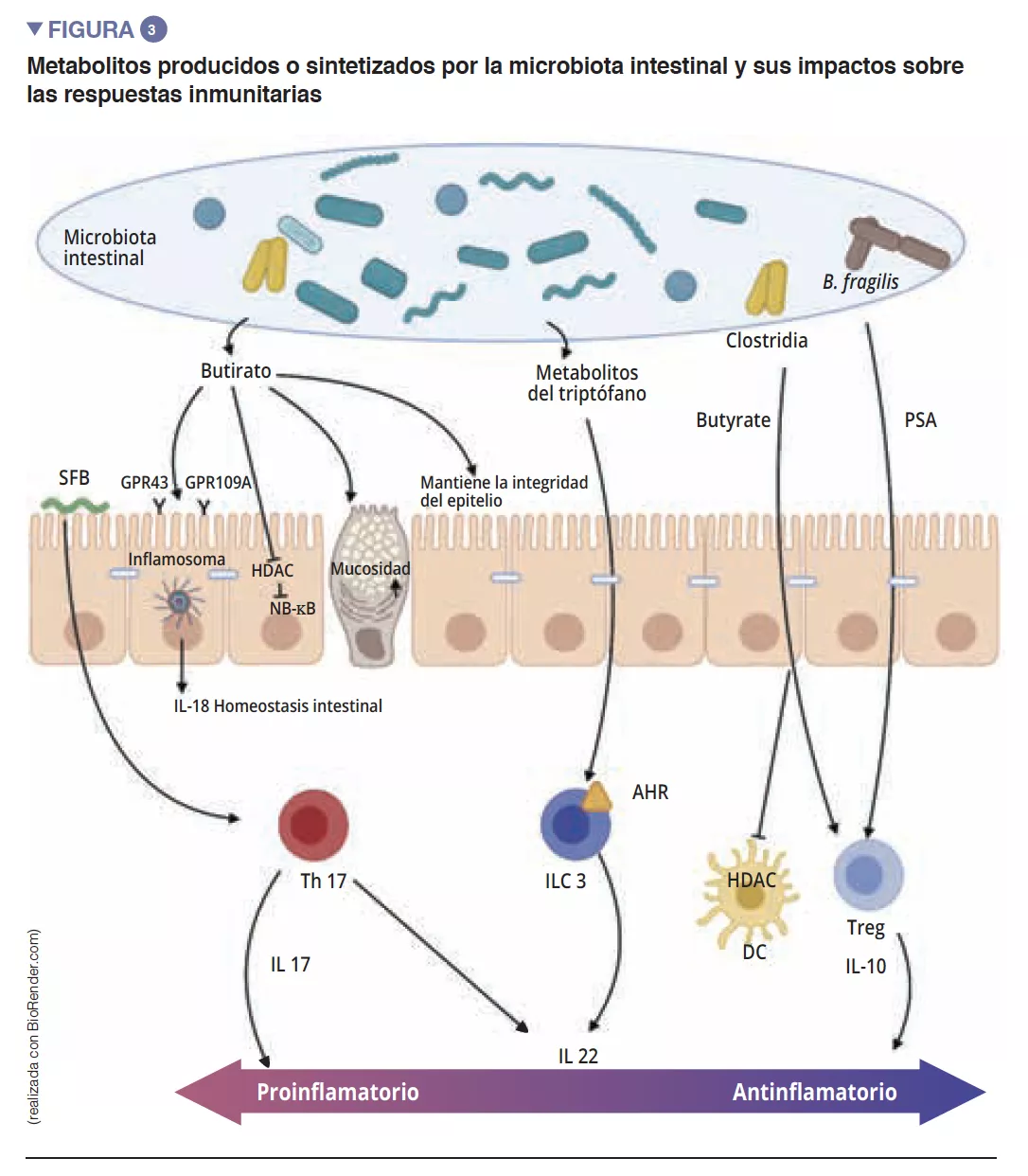
Los ácidos grasos de cadena corta (AGCC), los metabolitos derivados del triptófano y las sales biliares son los principales metabolitos procedentes de la microbiota intestinal que ejercen un efecto protector contra las infecciones [9, 10]. El butirato, el propionato y el succinato son conocidos por sus acciones sobre la homeostasia intestinal, sobre la secreción de mucosidad, pero también sobre las distintas células del sistema inmunitario. El butirato ejerce, entre otros, efectos antinflamatorios y antimicrobianos. En esta acción intervienen los receptores acoplados a las proteínas G (PGR) que se encuentran sobre las células epiteliales y los macrófagos [9]. F. prausnitzii es un gran productor de butirato, lo que puede explicar, en parte, su efecto antinflamatorio. De hecho, inhibe la activación de NF-kB e inhibe así la síntesis de citocinas proinflamatorias IFN-g, TNF-a, IL-1b, IL-8 por parte de los enterocitos [8] (Figura 3). Asimismo, induce modificaciones metabólicas y epigenéticas (a través de las histonas deacetilasas, HDAC) en macrófagos de ratones, amplificando así sus actividades antimicrobianas in vitro et in vivo [11]. Las bacterias comensales también pueden metabolizar el triptófano y producir sustancias antimicrobianas. Así, las Lactobacilli lo utilizan como fuente de energía para sintetizar un indol que se une a los receptores de arilhidrocarburos (AhR) presentes en las ILC3. Los AhR van a inducir primero la secreción de IL-22 por las ILC y posteriormente la secreción de AMP e inducirán una protección frente a infecciones [9].
De entre los virus entéricos, el norovirus y el rotavirus son las principales causas de gastroenteritis [12]. Los virus entéricos infectan distintos tipos celulares: los enterovirus 71 infectan específicamente las células caliciformes, mientras que el tropismo de los rotavirus se dirige principalmente hacia los enterocitos [13] (Figura 4A). La microbiota intestinal actúa como una barrera contra las infecciones virales entéricas. Los virus han evolucionado y se han adaptado a su anfitrión desarrollando mecanismos que les permiten atravesar la barrera intestinal y escapar a la inmunidad de barrera: de hecho, es difícil infectar eficazmente por vía oral a ratones con virus entéricos humanos [13]. La penetración del virus en el enterocito provoca la secreción de interferón (IFN) de tipo III. La detección vírica puede inducir IL-la, que activa las ILC3 para producir la IL-22. Esta IL protege de las infecciones virales entéricas y actúa en sinergia con los IFN de tipo III para inducir la expresión de efectores antivirales y de IL-15. El reconocimiento vírico por parte de los TLR-3 conduce a la activación de la vía NF-kB así como la producción de IL-15. La IL-15 activa los linfocitos citotóxicos (células NK). Los virus que han atravesado la barrera intestinal inducen una producción de IFN de tipo I por los macrófagos de la lámina propia (Figura 4B). Ciertos virus entéricos (rotavirus, reovirus, enterovirus) son capaces de unirse a las bacterias intestinales, favoreciendo la penetración en las células epiteliales intestinales [13]. Las SFB, que permiten una renovación epitelial, proporcionan una protección frente a la infección por rotavirus en el ratón al expulsar las células infectadas [14]. Los ácidos biliares metabolizados por la microbiota intestinal también desempeñan una función al proteger al intestino delgado (pero no al colon) de la infección aguda por el norovirus en el ratón, y favorecen la producción de IFN de tipo III en el intestino delgado [15].
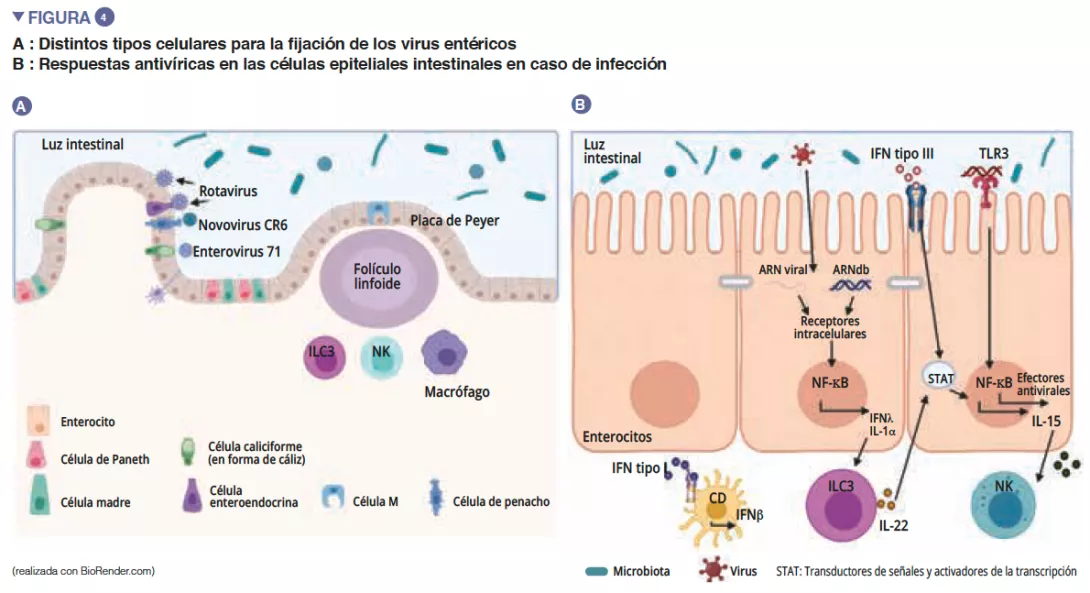
El estudio de las relaciones entre la microbiota intestinal y la inmunidad intestinal supone un gran avance en la investigación en gastroenterología. La homeostasis intestinal se mantiene gracias al reconocimiento de bacterias comensales por parte de células del sistema innato y de células del epitelio intestinal, ya sea por un contacto directo (en el caso de las SFB), o a través de la síntesis de metabolitos de la microbiota. La alteración de la homeostasis (disbiosis intestinal, infecciones, etc.) produce una estimulación de las respuestas innatas y una activación del sistema adaptativo. Una mala «gestión» de la inflamación podría dar lugar a enfermedades como el síndrome del intestino irritable posinfeccioso.
1 Perez-Lopez A, Behnsen J, Nuccio SP, Raffatellu M. Mucosal immunity to pathogenic intestinal bacteria. Nat Rev Immunol 2016 ; 16 : 135-48.
2 Allaire JM, Crowley SM, Law HT, et al. The intestinal epithelium: central coordinator of mucosal immunity. Trends Immunol 2018 ; 39 : 677-96.
3 Van der Sluis M, De Koning BA, De Bruijn AC, et al. Muc2-deficient mice spontaneously develop colitis, indicating that MUC2 is critical for colonic protection. Gastroenterology 2006 ; 131 : 117-29.
4 Kim M, Hill A A, Wu WJ, et al. Intestinal microbes direct CX3CR1+ cells to balance intestinal immunity. Gut Microbes 2018 ; 17 : 151-63.
5 Flanningan KL, Denning TL. Segmented filamentous bacteria-induced immune responses: a balancing act between host protection and autoimmunity. Immunology 2018 ; 154 : 537-46.
6 Ivanov I I, Atarashi K, Manel N, et al. Induction of intestinal Th17 cells by segmented filamentous bacteria. Cell 2009 ; 139 : 485-98.
7 Atarashi K, Tanoue T, Oshima K, et al. Treg induction by rationally selected mixture of Clostridia strains from the human microbiota. Nature 2013 ; 500 : 232-6.
8 Miquel S, Martin R, Rossi O, et al. Faecalibacterium prausnitzi and human intestinal health. Curr Opin Microbiol 2013 ; 16 : 255- 61.
9 Levy M, Blacher E, Elinav E. Microbiome, metabolites and host immunity. Curr Opin Microbiol 2017 ; 35 : 8-15.
10 Michaudel C, Sokol H. The gut microbiota at the service of immunometabolism. Cell Metabolism 2020 ; 32 : 514-23.
11 Schulthess J, Pandey S, Capitani S, et al. The short Chain Fatty Acid Butyrate imprints an antimicrobial program in macrophages. Immunity 2019 ; 50 : 432-45.
12 Bányai K, Estes MK, Martella V, et al. Viral gastroenteritis. Lancet 2018 ; 392 : 175-86.
13 Segrist E, Cherry S. Using diverse model systems to define intestinal epithelial defenses to enteric viral infections. Cell Host Microbe 2020 ; 27 : 329-44.
14 Shi Z, Zou J, Zhang Z, et al. Segmented filamentous bacteria prevent and cure rotavirus infection. Cell 2019 ; 179 : 644-658.e13.
15 Grau KR, Zhu S, Peterson ST, et al. The intestinal regionalization of acute norovirus infection is regulated by the microbiota via bile acid-mediated priming of type III interferon. Nat Microbiol 2020 ; 5 : 84-92.
Síntesis
Por el Pr. Tao Zuo
Instituto de Investigación de Gastroenterología SYSU, Instituto de Gastroenterología de Guangdong, Sexto Hospital Afiliado de la Universidad Sun Yat-Sen, Guangzhou, China
La microbiota intestinal, incluidas las fracciones bacteriana, fúngica y vírica, puebla los intestinos humanos y regula la inmunidad del anfitrión frente a invasiones de patógenos. La gran heterogeneidad en la composición de la microbiota intestinal (MI) en distintas personas puede influir en las respuestas inmunitarias del anfitrión a la infección por SARS-CoV-2, dando lugar a distintos síntomas y evoluciones de la COVID-19. Por otro lado, aunque la infección por SARS-CoV-2 provoca principalmente síntomas respiratorios, también causa una profunda desregulación de la inmunidad general del anfitrión y afecta al aparato digestivo, donde la microbiota intestinal puede verse afectada tanto a corto como a largo plazo. Aquí, vamos a revisar la evidencia actual sobre el impacto de la COVID-19 en la microbiota intestinal humana, así como las asociaciones entre la composición de la microbiota intestinal y la gravedad de la COVID-19.
La COVID-19 es una enfermedad respiratoria causada por un nuevo coronavirus (SARS-CoV-2) que actualmente sigue afectando a decenas de millones de personas en todo el mundo. Aunque la mayoría de los pacientes con COVID-19 presenta síntomas respiratorios, hasta un 20 % muestra síntomas gastrointestinales (GI) como diarrea [1], lo que sugiere que el tubo digestivo es un foco extrapulmonar de la infección por SARS-CoV-2 y de expresión de esta enfermedad. Además, la COVID-19 presenta un amplio abanico de niveles de gravedad, que va desde ser asintomática, leve, grave, hasta crítica con insuficiencia respiratoria e incluso la muerte [2].
El tracto gastrointestinal es el órgano inmunitario más grande en los seres humanos, y desempeña funciones decisivas en la defensa del anfitrión ante infecciones por patógenos. Hay billones de microorganismos que viven y colonizan el intestino humano –bacterias, hongos, virus y otras formas de vida que en conjunto se conocen como microbiota– que regulan la inmunidad del anfitrión. Por tanto, es de vital importancia entender si la microbiota intestinal modula la susceptibilidad del anfitrión a la infección por SARS-CoV-2 y su gravedad, así como el impacto de la infección por SARS CoV-2 en la microbiota intestinal del anfitrión y su efecto posterior a largo plazo en la salud humana.
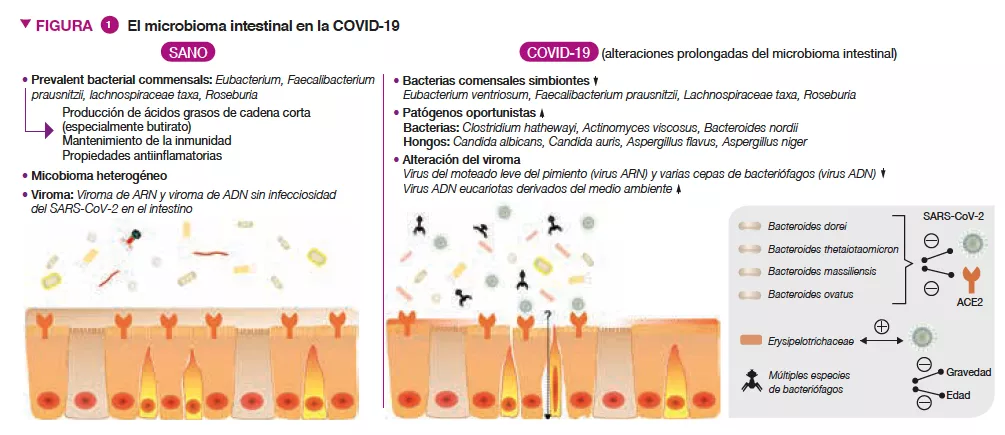
Pacientes con COVID-19 presentaron, en comparación con personas sanas, alteraciones significativas en el microbioma intestinal, caracterizadas por una disminución de bacterias comensales beneficiosas y un aumento de patógenos oportunistas en el intestino (Figura 1) [3]. La disminución de los simbiontes intestinales persistió incluso después de la resolución de la COVID-19. Los valores de referencia (en el momento de la hospitalización) de abundancia para las bacterias Coprobacillus, Clostridium ramosum y C. hathewayi se correlacionaron positivamente con la gravedad de la COVID-19, mientras que había una correlación inversa entre la abundancia de Faecalibacterium prausnitzii (conocida por ser una bacteria antinflamatoria) y la gravedad de la enfermedad. El SARS-CoV-2 utiliza el receptor de la enzima convertidora de angiotensina 2 (ECA2) para entrar en el anfitrión, y este receptor se encuentra altamente expresado tanto en el tracto respiratorio como en el gastrointestinal [4]. La ECA2 es importante para controlar la inflamación intestinal y la ecología microbiana del intestino [5]. Se observó que cuatro especies de Bacteroides, B. dorei, B. thetaiotaomicron, B. massiliensis, y B. ovatus, se asociaban negativamente con la expresión de la ECA2 en el intestino del ratón [6]. Curiosamente, su abundancia en el microbioma fecal también mostró una correlación inversa con la carga vírica fecal de SARS-CoV-2 en pacientes con COVID-19 durante el curso de la enfermedad. Estos resultados sugieren que la COVID-19 perjudica a la MI humana y que es posible que esta module la defensa del anfitrión frente a la infección por SARS-CoV-2.
El tracto gastrointestinal también alberga un amplio número de hongos, conocidos de forma colectiva como micobioma (microbioma fúngico), el cual se ha demostrado que está implicado de forma causal en la formación de la MI y en el desarrollo inmunitario [7]. Pacientes con COVID-19 también presentaban micobiomas intestinales alterados, caracterizados por un enriquecimiento de Candida albicans y configuraciones de micobioma muy heterogéneas (Figura 1) [8]. La diversidad del micobioma fecal en los pacientes con COVID-19 en el momento del alta era 2,5 veces superior a la de personas sanas. Había una fuerte presencia de hongos patógenos oportunistas como Candida albicans, C. auris y Aspergillus flavus en las heces de pacientes con COVID-19 durante el curso de la enfermedad. Se detectaron dos hongos patógenos asociados a síntomas respiratorios, A.lavus y A.niger, en muestras fecales de un subconjunto de pacientes con COVID-19, incluso después de la resolución de la enfermedad. En un 30 % de pacientes con COVID-19 persistieron micobiomas intestinales inestables y disbiosis.
Mediante secuenciación de escopeta de ARN vírico se encontró una firma de infección vírica intestinal activa en el 47 % de los pacientes con COVID-19, incluso en ausencia de síntomas gastrointestinales y después de la eliminación respiratoria del SARS-CoV-2 [9], lo que sugiere una infección «latente» por SARS-CoV-2 en el tracto gastrointestinal y un posible riesgo de transmisión fecal-oral. Los pacientes con esta actividad gastrointestinal del SARSCoV- 2 albergaban composiciones y funciones anómalas de la MI, caracterizadas por una gran abundancia de patógenos oportunistas y una mayor capacidad de biosíntesis de nucleótidos y aminoácidos y del metabolismo de los hidratos de carbono (glucólisis) [9].
El tracto gastrointestinal humano también alberga abundantes elementos víricos / bacteriófagos conocidos de forma colectiva como viroma intestinal. Pacientes con COVID-19 tenían una infrarrepresentación del virus del moteado leve del pimiento (virus ARN) y varias cepas de bacteriófagos (virus ADN) y un aumento de virus de ADN eucariotas derivados del entorno en muestras fecales, en comparación con personas sin COVID-19 (Figura 1) [10]. El viroma fecal en la infección por SARS CoV-2 reveló una mayor capacidad de codificación de genes asociados al estrés, la inflamación y la virulencia. En los valores de referencia del paciente, las abundancias fecales de virus ARN, del virus del mosaico suave del pimiento y de varias especies bacteriófagas se correlacionaban de manera inversa con la gravedad de la COVID-19. Estos virus también estaban asociados negativamente con los niveles en sangre de proteínas proinflamatorias, glóbulos blancos y neutrófilos, lo que indica que es posible que los virus residentes en el intestino modulen la respuesta inmunitaria a la infección por SARS-CoV-2. Entre las especies de virus ADN asociadas a la gravedad de la COVID-19, se observó que el 40 % de las especies tenían una relación inversa con la edad, lo que podría respaldar la observación de que las personas de edad avanzada presentan un mayor riesgo de padecer una COVID-19 más grave.
En resumen, el conjunto de pruebas sugiere que el microbioma intestinal humano (microbiota bacteriana, micobioma y viroma) se altera en caso de COVID-19. Esta alteración persiste incluso después de la resolución de la enfermedad, lo que, a largo plazo, puede suponer una amenaza para la salud del anfitrión. La composición de la microbiota intestinal se asocia con las respuestas inmunitarias del anfitrión y la gravedad de la COVID-19 en la infección por SARS CoV-2. Es necesario investigar más para explorar los efectos a largo plazo de la COVID-19 y para mejorar el microbioma intestinal del anfitrión y su inmunidad ante esta pandemia vírica sin precedentes.
¿Por qué es tan importante la microbiota intestinal para la salud?
En nuestros intestinos viven trillones1 de bacterias que ejercen efectos fascinantes sobre el cuerpo humano. Tratemos de comprender cómo funciona nuestra microbiota intestinal y por qué debemos cuidar de ella.

Es probable que haya oído hablar de la “flora intestinal”. Pues bien, su nombre científico es “microbiota intestinal”. ¿Cuál es la definición de la microbiota intestinal? Se compone de trillones1 de (sidenote: Microorganismos Organismos vivos que son demasiado pequeños para observarlos a simple vista. Incluyen bacterias, virus, hongos, arqueas, protozoos, etc. y se llaman comúnmente «microbios». What is microbiology? Microbiology Society. ) que viven en nuestros intestinos tales como bacterias, virus, hongos (entre otros, levaduras) e incluso parásitos.
Por cierto, “microbiota” y “microbioma” son dos palabras que a menudo se usan indiferentemente aunque tienen significados ligeramente distintos: mientras que la microbiota se refiere básicamente a los microorganismos individuales y responde a la pregunta “¿quién está allí?”, el microbioma se refiere a su genoma, a “lo que llevan dentro”2 y responde a la pregunta “¿qué hacen?” o “¿cuál es su función?
Cada uno de nosotros tiene una microbiota única, como si fuera una huella digital.1 Al nacer, los microorganismos fecales y vaginales transmitidos por la madre durante el parto vaginal, o los microorganismos del entorno en el caso de un parto por cesárea,3 dan inicio a la colonización de la microbiota intestinal.4 Esta tarda alrededor de tres años en constituirse, diversificarse y estabilizarse.5 Una vez alcanzada la edad adulta,6 su composición se mantiene relativamente estable hasta la vejez, cuando sufre de nuevo una profunda transformación que la empobrece ligeramente.7
100 millones 100 millones de millones de «buenas» bacterias viven en nuestros intestinos.
2º se considera a menudo que la microbiota intestinal es nuestro segundo cerebro
La microbiota intestinal puede considerarse como un órgano funcional del cuerpo humano. ¿Por qué debemos cuidar de nuestra microbiota? Trabaja en estrecha colaboración con los intestinos y tiene cuatro funciones importantes:
ayudando las células intestinales a absorber nutrientes (azúcares, aminoácidos, vitaminas…) o a fermentar una pequeña parte de los alimentos. Estos procesos de fermentación producen gas y numerosos metabolitos tales como ácidos grasos de cadena corta que constituyen un auténtico “combustible” para las células del colon.8
por su intervención activa en la producción de moco gastrointestinal, la irrigación de las células intestinales y la actividad enzimática de la mucosa.9
contra patógenos y toxinas.10 Además, ciertas bacterias liberan moléculas antimicrobianas contra bacterias (sidenote: Patógeno un patógeno es un microorganismo que causa, o puede causar, una enfermedad Pirofski LA, Casadevall A. Q and A: What is a pathogen? A question that begs the point. BMC Biol. 2012 Jan 31;10:6. ) , mientras que otras estimulan la producción de moco, protegiendo las células intestinales de los ataques y evitando efectos nocivos sobre el cuerpo.11
en el desarrollo del sistema inmunitario humano. Las bacterias de la flora intestinal participan en la maduración y la activación de las células del sistema inmunitario intestinal, que nos protege de los ataques de patógenos (bacterias y virus). El intestino es el principal reservorio de células inmunes del cuerpo. A su vez, el sistema inmunitario influye en la composición y diversidad de la microbiota.12
La composición de la microbiota intestinal se caracteriza por su gran diversidad (número de especies diferentes presentes en un individuo dado) y por su abundancia (cantidad total de microorganismos presentes). En caso de perturbación de esta composición, se rompe el equilibrio y aparece una disbiosis 13 que puede asociarse con distintas enfermedades.
Numerosos factores pueden afectar a la diversidad y composición de la microbiota intestinal. Entre estos factores cabe mencionar los siguientes:
Factores individuales, tales como:
Factores relacionados con el entorno del individuo, tales como:
La disbiosis, ¿causa o consecuencia de estas enfermedades? Esta pregunta aún no ha sido respondida con certeza por los estudios científicos.
Los cólicos del lactante afectan del 20 al 25% de los bebés de entre 1 y 4 meses de edad.14
La diarrea relacionada con antibióticos se manifiesta en el 5 al 35% de los pacientes que toman antibióticos.15
La diarrea del viajero1 es una infección causada por el consumo de agua o alimentos contaminados. Entre un 3% y un 17% de los pacientes afectados pueden padecer el síndrome del intestino irritable (SII) después de la infección.16
La gastroenteritis suele ser benigna y de origen viral pero es responsable del fallecimiento de más de 200 000 niños al año en el mundo.17
La obesidad es una enfermedad crónica frecuente, grave y costosa que en 2016 afectaba al 13% de la población adulta del mundo (el 11% de los hombres y el 15% de las mujeres).18
El SII es uno de los trastornos gastrointestinales funcionales más frecuentes y se caracteriza por dolor abdominal y alteraciones de los hábitos defecatorios del huésped (estreñimiento, diarrea o ambos de manera alternada). Su prevalencia varía considerablemente entre países.19
La enfermedad de Crohn es una enfermedad inflamatoria del intestino en la que la inflamación puede afectar a todos los niveles del sistema digestivo, desde la boca hasta el ano. Estudios recientes demostraron que la microbiota intestinal desempeña una función importante en la etiopatogenia de esta enfermedad.20
El cáncer de estómago21 y el cáncer colorrectal22 son cánceres gastrointestinales asociados con una disbiosis intestinal.
Sin embargo, el papel de la microbiota humana no se limita al intestino: estudios recientes revelaron que la microbiota intestinal podría intervenir más allá del tracto gastrointestinal. De hecho, la microbiota intestinal se ha asociado con distintas enfermedades extraintestinales como el acné,23 ciertas alergias,24 la obesidad,25 trastornos de ansiedad,26 trastornos del espectro autista,26 etc. ¡Y esto no es todo! La microbiota intestinal también podría intervenir en trastornos neurodegenerativos como la enfermedad de Alzheimer,27 o la de Parkinson.28 De hecho, existe un sistema de comunicación bidireccional entre el intestino y el cerebro denominado “eje intestino-cerebro”, y la microbiota intestinal podría ejercer una influencia en estas interacciones. Esto explica por qué a veces se dice que el intestino es el segundo cerebro.
Ahora conoce el papel central que desempeña la microbiota intestinal en la salud humana. Entonces, ¿cómo podemos cuidar de nuestra propia microbiota?, ¿Cómo reforzar su microbiota intestinal? Numerosos estudios científicos se han interesado en la manera de evitar cualquier perturbación de su composición y mantenerla equilibrada lo mejor posible. 29 Pero la respuesta es complicada: no basta aportar bacterias o levaduras beneficiosas para rellenar o enriquecer la microbiota existente o reemplazar los microorganismos nocivos. En realidad, la idea consiste en ejercer una influencia en la microbiota para ayudarla a funcionar sin problemas, mejorando así la salud del huésped. 30
Dieta: la diversidad y la calidad de los alimentos que ingerimos contribuyen al equilibrio de nuestra microbiota intestina.31,32 Por otro lado, una dieta desequilibrada puede afectar la composición de nuestra microbiota intestinal y provocar ciertas enfermedades.33 ¡Es muy importante saber qué tipos de alimentos tienen efectos beneficiosos o negativos para mantener nuestro intestino en forma!34
Probióticos: los probióticos son microorganismos vivos que, si se administran en cantidades adecuadas, producen efectos beneficiosos en la salud del huésped.35,36
Prebióticos: los prebióticos son fibras alimentarias no digeribles específicas que mejoran la salud. Los microorganismos beneficiosos de la microbiota del huésped los utilizan de manera selectiva.37,38 Dado que algunos alimentos tienen un contenido particularmente elevado de prebióticos, es importante prestar atención a nuestra dieta. Ciertos productos específicos que combinan prebióticos y probióticos se denominan simbióticos.39
Trasplante: al igual que otros órganos, la microbiota intestinal puede trasplantarse a un individuo para tratar de restablecer el equilibrio de su ecosistema microbiano intestinal.32 Por el momento, esta estrategia terapéutica, conocida como trasplante de microbiota fecal (TMF), solo ha sido aprobada para tratar la infección recurrente por Clostridioides difficile32, pero se están realizando investigaciones para evaluar sus efectos en otras afecciones específicas como la enfermedad de Crohn, la colitis ulcerosa, el síndrome del intestino irritable o enfermedades metabólicas.32
Ahora que ha leído todo lo que necesita saber sobre la microbiota intestinal, también debe tener en mente que hay (sidenote: Microorganismos Organismos vivos que son demasiado pequeños para observarlos a simple vista. Incluyen bacterias, virus, hongos, arqueas, protozoos, etc. y se llaman comúnmente «microbios». What is microbiology? Microbiology Society. ) vivos en todas las partes del cuerpo: en la piel,40 las vías urinarias,41 la vagina,42 la boca,43 los oídos,44 los pulmones45 y, al igual que sucede en el intestino, los microorganismos que allí viven desempeñan un papel crucial en el funcionamiento de los respectivos órganos y el mantenimiento de una buena salud. Si desea más información, visite esta página.
Toda la información contenida en este artículo procede de fuentes científicas autorizadas. Recuerde que no es exhaustiva. Esta es la lista de los estudios de donde hemos extraído toda esta información.
11 Sokol H. Microbiota and barrier effect. In: Marteau P, Dore J, eds. Gut Microbiota: A Full-Fledged Organ. Paris: John Libby Eurotext; 2017:65-71.
17 Stuempfig ND, Seroy J. Viral Gastroenteritis. In: StatPearls. Treasure Island (FL): StatPearls
18 WHO. Fact sheets on obesity and overweight June 2021
27 Qian XH, Song XX, Liu XL, et al. Inflammatory pathways in Alzheimer's disease mediated by gut microbiota. Ageing Res Rev. 2021 Mar 9;68:101317.
