Metagenómica del viroma fecal: efecto acumulativo de la cantidad de gluten y de enterovirus en el riesgo de autoinmunidad de la enfermedad celíaca en niños con predisposición: el estudio Teddy
Artículo comentado - Niño
Por el Pr. Emmanuel Mas
Gastroenterología y nutricíon, Hospital de Niños, Toulouse, Francia
Comentario del artículo original de Lindfors et al. (Gut 2019) [1]
Se han sugerido varios facilitadores medioambientales de la enfermedad celíaca (EC) como un consumo de gluten más elevado, infecciones gastrointestinales frecuentes, y adenovirus, enterovirus, rotavirus y reovirus. Sin embargo, no se sabe si existe una interacción entre la cantidad de gluten ingerido y las exposiciones virales en el desarrollo de la EC. El objetivo de este estudio era averiguar si diferentes exposiciones virales, solas o asociadas al gluten, aumentan el riesgo de autoinmunidad de la EC en niños genéticamente predispuestos. Su conclusión es que una exposición frecuente a enterovirus entre los 1 y 2 años se asocia, efectivamente, a un mayor riesgo de autoinmunidad de la EC. El efecto acumulativo de la interacción entre los enterovirus y un consumo de gluten más elevado podrían ser dos de las causas.
¿Qué se sabe ya sobre este tema?
La enfermedad celíaca es una patología autoinmunitaria que aparece en personas genéticamente predispuestas con un genotipo HLA DQ2 y/o DQ8 positivo. Se caracteriza por la presencia de una atrofia vellositaria y de una infiltración linfocitaria en el epitelio del intestino delgado. El gluten presente en la alimentación induce una respuesta autoinmunitaria dirigida contra la transglutaminasa tisular. La aparición de anticuerpos antitransglutaminasa (TG) define la autoinmunidad de la enfermedad celíaca.
El aumento de la incidencia de las enfermedades autoinmunitarias lleva a sospechar que hay una influencia de factores medioambientales en su patogénesis. Ciertos estudios observacionales sugieren que las infecciones virales podrían inducir una ruptura de la tolerancia oral al gluten y la aparición de la enfermedad celíaca
¿Cuáles son los principales resultados aportados por este estudio?
Se trata de un estudio caso-control realizado en la cohorte de nacimiento TEDDY (The Environmental Determinants of Diabetes in the Young) compuesta por 8676 niños menores de 4 meses y medio, y con un seguimiento durante 15 años. El objetivo principal de esta cohorte es identificar a los factores genéticos y medioambientales asociados a la diabetes de tipo 1 y a la enfermedad celíaca. Después de juntar de dos en dos a los niños en función de sus antecedentes familiares de diabetes de tipo 1, sexo y lugar de inclusión en el estudio, se hizo el seguimiento de 83 pares (niños con predisposición y control) en el análisis final, para los cuales los datos del viroma fecal estaban disponibles después de la introducción del gluten. Entre esos pares, 16 tenían antecedentes familiares de diabetes de tipo 1. Durante el seguimiento, en 28 casos de autoinmunidad de la enfermedad celíaca apareció la enfermedad.
Cada mes se recogieron muestras fecales, desde los 3 meses hasta los 2 años; se hizo una búsqueda de enterovirus, adenovirus, astrovirus, norovirus, reovirus y rotavirus. Cada 3 meses, se enviaba un cuestionario alimenticio para recoger información sobre la lactancia materna y la edad de introducción de alimentos que contienen gluten. Un registro alimenticio durante 3 días permitió calcular las cantidades de gluten ingeridas a los 6, 9, 12, 18 y 24 meses.
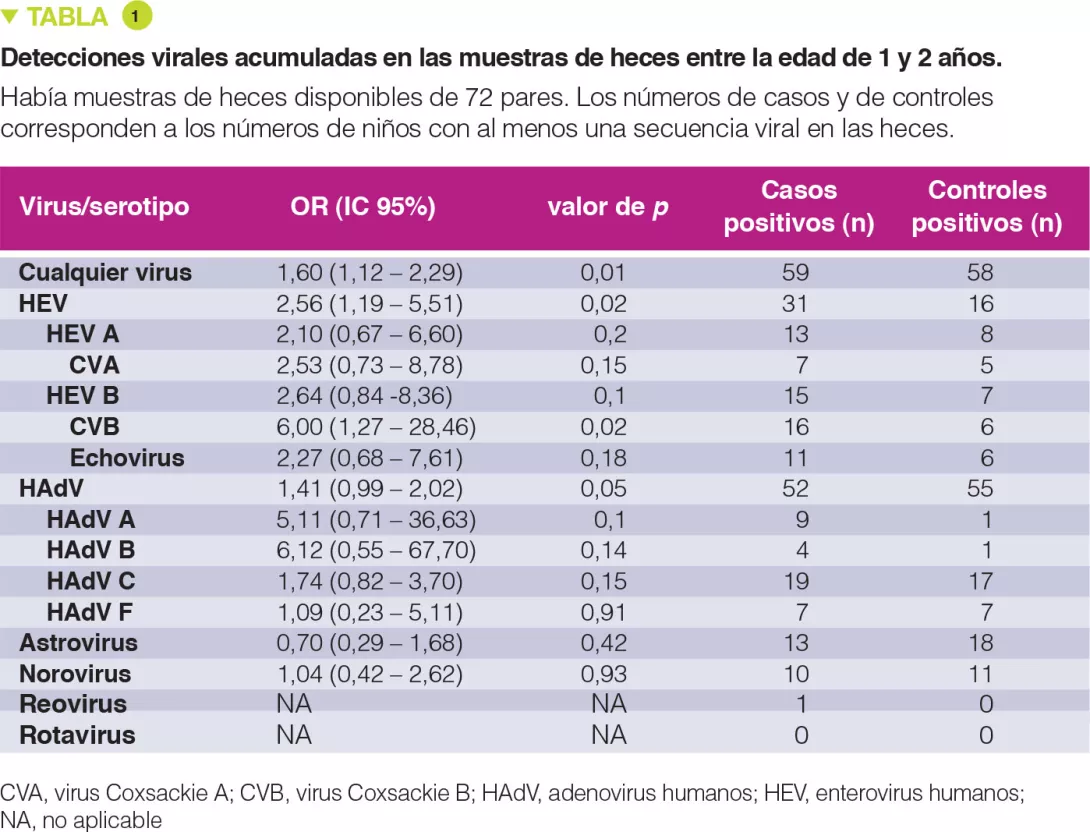
El porcentaje de muestras de heces positivas para cualquier virus variaba del 22 al 50 %, sin pico relacionado con la edad, y el de los enterovirus del 0 al 21 % después de 6 meses. Entre los 1 y 2 años, se detectaron enterovirus en 31 casos frente a 16 en los controles (Tabla 1). El número acumulado de muestras de heces positivas para cualquier virus estaba asociado a un mayor riesgo de autoinmunidad de la enfermedad celíaca (OR 1,60; p = 0,01), con una mayor asociación en el caso de los enterovirus (OR 2,56; p = 0,02).
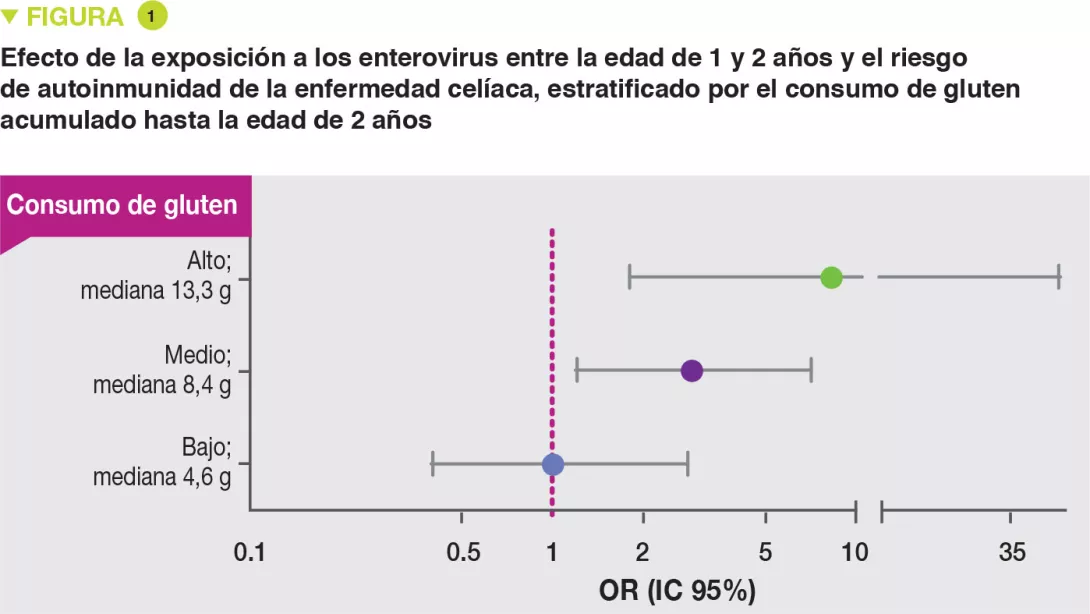
El riesgo de autoinmunidad de la enfermedad celíaca no aumentaba con las infecciones virales que aparecían después de la edad de introducción del gluten, cuando se mantenía la lactancia materna. En cambio, después del destete, en las muestras de heces recogidas entre los 1 y los 2 años, cuando se había introducido el gluten, existía una relación entre el número acumulado de virus (OR 1,41; p = 0,05) y de enterovirus encontrados (OR 2,47; p = 0,03) y el riesgo de autoinmunidad de la enfermedad celíaca. Existía una interacción significativa entre la presencia de enterovirus encontrados entre los 1 y los 2 años y la cantidad integrada de gluten hasta los 2 años sobre el riesgo de autoinmunidad de la enfermedad celíaca (p = 0,30). Este riesgo aumentaba con la cantidad ingerida de gluten: alta (OR 8,3), media (OR 2,9) y baja (OR 1,0) (Figura 1).
Puntos clave
-
Los factores medioambientales influyen en la enfermedad celíaca.
-
La exposición a los enterovirus es un factor de riesgo de aparición de anticuerpos de antitransglutaminasas en niños HLA DQ y/o DQ8 positivos.
-
Este riesgo se incrementa si hay una aportación importante de gluten en la alimentación.
¿Cuáles son las consecuencias en la práctica?
Los resultados de este estudio son interesantes para prevenir la aparición de auto-anticuerpos de la enfermedad celíaca en niños con riesgo genético. Para ello, habría que prestar atención a las cantidades ingeridas de gluten, en concreto en caso de exposición a los enterovirus, sobre todo cuando el niño ya no está tomando leche materna.
Conclusión
Este estudio ha demostrado que existía una asociación entre las exposiciones digestivas a los enterovirus y el riesgo de autoinmunidad de la enfermedad celíaca en niños con riesgo genético. Este riesgo aumenta con la cantidad de gluten ingerido.