Microbiote intestinal #10
Par le Pr. Markku Voutilainen
Faculté de médecine de l’Université de Turku ; gastro-entérologie, Hôpital universitaire de Turku, Finlande


Par le Pr. Markku Voutilainen
Faculté de médecine de l’Université de Turku ; gastro-entérologie, Hôpital universitaire de Turku, Finlande

Pour mieux comprendre l’impact du microbiote intestinal sur la progression/sévérité de la maladie de Parkinson (MP) et sur les agrégats d’α-synucléine (α-syn) dans les corps de Lewy, les auteurs ont utilisé un modèle nématode Caenorhabditis elegans. Ils ont remarqué que les spores et les cellules végétatives d’une souche de Bacillus subtilis induisaient la formation d’un biofilm dans l’intestin du ver ainsi que la libération de métabolites bactériens. Ainsi, des voies protectrices telles que le métabolisme des sphingolipides ont été régulées de manière différenciée ; une élimination des agrégats d’α-syn préformés et une inhibition de l’agrégation des α-syn ont été observées chez les animaux jeunes et vieux. D’après les auteurs, les effets de cette souche en complément alimentaire devraient être davantage explorés dans le traitement de la MP.
Le microbiote intestinal pourrait avoir un impact sur l’efficacité et la toxicité des traitements anticancéreux. D’après les résultats dont on dispose actuellement, il influence significativement la réponse au traitement par inhibiteur de point de contrôle immunitaire (Immune Checkpoint Inhibitor, ICI). L’étude a évalué l’impact de l’utilisation des antibiotiques, des inhibiteurs de la pompe à protons (IPP), des corticoïdes et des opioïdes sur la réponse thérapeutique au traitement par ICI, définie d’après les critères iRECIST1. Elle a montré que le recours aux antibiotiques en soi n’était pas associé à une réduction de l’efficacité du traitement par ICI, mais que des cures multiples ou prolongées d’antibiothérapie altéraient les résultats de l’immunothérapie. C’est la première étude qui montre que l’utilisation concomitante d’opioïdes, mais pas d’IPP ou de corticoïdes, est associée à de moins bons résultats du traitement par ICI.
Par le Pr. Markku Voutilainen
Faculté de médecine de l’Université de Turku ; gastro-entérologie, Hôpital universitaire de Turku, Finlande
Le meibum empêche l’évaporation du film lacrymal de l’oeil, permet l’homéostasie de la surface oculaire et possède son propre microbiote.
Mais ce microbiote et celui des surfaces oculaires changent-ils avec l’âge ? Cette étude montre que les échantillons de peau palpébrale prélevés chez les sujets jeunes présentaient une faible diversité α (indice de Shannon) et que Probionibacterium acnes et Staphylococcus epidermidis étaient les espèces dominantes. Les microbiotes du meibum et du sac conjonctival étaient différents de celui de la peau, et étaient caractérisés par un indice de diversité α élevé avec un grand nombre d’espèces bactériennes. Chez les sujets âgés, Corynebacterium sp. et les espèces appartenant à la famille des Neisseriaceae prédominaient au niveau de la peau palpébrale et l’indice de Shannon était significativement réduit au niveau du meibum et du sac conjonctival. Les auteurs concluent que le microbiote du meibum est effectivement altéré avec l’âge, de la même manière chez les hommes que chez les femmes.
On sait que le microbiote intestinal évolue avec l’âge, et on observe le même phénomène avec les microbiotes buccal et cutané.
Mais lequel permet le mieux de prédire le vieillissement ? Les auteurs ont évalué la diversité du microbiote sur près de 9 000 échantillons prélevés sur la peau, dans la salive et les intestins de sujets sains dans 10 études. Les taxa enrichis chez les sujets jeunes (18 à 30 ans) semblaient plus abondants et répandus que les taxa enrichis chez les sujets âgés (> 60 ans) ; le vieillissement pourrait être lié à une perte de taxa clés. En comparaison aux microbiotes intestinal et buccal, celui de la peau était le meilleur prédicteur de l’âge (moyenne 3,8 ans ± 0,45 écart type). Les auteurs ont identifié des genres et familles comprenant des bactéries anaérobies (Mycoplasma, Enterobacteriaceae, Pasteurellaceae) qui étaient corrélées négativement avec l’âge. Les modifications liées à l’âge de la physiologie de la peau (diminution de la production de sébum, augmentation de la sécheresse cutanée) et des réactions immunitaires de l’hôte pourraient être à l’origine de ces modifications du microbiote.
Article commenté - Rubrique adulte
Pr. Harry Sokol
Gastro-entérologie et nutrition, Hôpital Saint-Antoine, Paris, France
Bacteriophages infecting bacteria, illustration. A bacteriophage, or phage, is a virus that infects bacteria.
Commentaire de l’article original de Clooney et al. (Cell Host & Microbe 2019) [1]
On pense actuellement que le virome intestinal humain a un impact significatif sur le microbiote et la santé humaine. Cependant, la plupart des analyses ont été effectuées sur une fraction limitée de virus connus. En réalisant une analyse du virome entier sur une cohorte publiée de maladie inflammatoire chronique de l’intestin (MICI) et des nouvelles données sur la rectocolite hémorragique, les auteurs ont mis en lumière la composante virale du microbiote intestinal humain dans les MICI au-delà de la minorité identifiable. Les auteurs y ont observé des changements spécifiques au cours des MICI et une augmentation du nombre de séquences de phages tempérés chez les patients atteints de la maladie de Crohn. Contrairement aux méthodes antérieures dépendantes de la base de données, aucun changement dans la richesse virale n’a été observé. Chez les sujets atteints de MICI, les changements dans la composition du virome reflétaient des altérations de la composition bactérienne. De plus, l’association des données du bactériome et du virome améliorait la puissance de classification entre la santé et la maladie. Cette approche de l’analyse des génomes de virus entiers à travers plusieurs cohortes met en évidence des signaux significatifs au cours des MICI, qui pourraient être cruciaux pour le développement de futurs biomarqueurs et traitements.
Le virome est probablement l’une des principales forces qui façonnent le microbiote intestinal humain, mais il s’agit peut-être aussi de sa composante la moins bien comprise. Il est dominé par les bactériophages (phages), qui jouent un rôle vital dans de nombreuses communautés microbiennes en stimulant la diversité, en aidant au renouvellement des nutriments et en facilitant le transfert horizontal de gènes. Comprendre le rôle des bactériophages dans les structures des communautés microbiennes sera essentiel si nous voulons comprendre ou contrôler les altérations du microbiome intestinal humain qui sont associées à de nombreuses maladies. De nombreuses bactéries intestinales (et des hôtes potentiels de phages) restent difficiles à cultiver, ce qui signifie que l’analyse du virome dépend fortement du séquençage métagénomique et des approches bio-informatiques.
Une large majorité du virome intestinal ne peut être étudié car il est absent des bases de données.
Une méthode indépendante des bases de données développée ici permet d’appréhender le virome intestinal dans sa globalité.
Le virome intestinal est altéré dans les MICI, moins stable et dominé par des phages tempérés.
Le virome intestinal pourrait être utilisé comme biomarqueur ou cible thérapeutique dans le futur.
Cependant, il n’existe pas de gènes marqueurs universels pour les phages (similaires à l’ARNr 16S pour les bactéries) et il y a un manque d’informations taxonomiques avec des bases de données pauvres, ce qui signifie que des méthodes indépendantes des bases de données sont nécessaires. Les premières études métagénomiques ont mis en évidence la diversité du virome intestinal humain, mais n’ont pu classer qu’une fraction mineure (2 %) de l’ADN séquencé [2]. Les améliorations des technologies de séquençage à haut débit ont permis au virome d’être analysé avec un niveau de détail inédit. Il a été confirmé que le virome est incroyablement diversifié, que la majorité ne s’aligne pas sur les séquences connues dans les bases de données (appelée la matière noire virale), et que la composition est unique aux individus..
Bien que l’étiologie des MICI reste floue, ces maladies sont multifactorielles et associées à des altérations du microbiome intestinal. Un ensemble de données émergent prouvant que le virome intestinal est altéré dans les MICI [3] avec une diversité globale accrue et une abondance relative accrue de l’ordre des Caudovirales. Pourtant, presque tous les résultats ont été tirés des changements de composition de la fraction identifiable du virome, qui peuvent représenter aussi peu que 15 % des données [3]. Cela limite la compréhension globale du virome et entrave l’identification des biomarqueurs potentiels de la maladie. Une méthode d’analyse indépendante de la base de données est essentielle si l’on souhaite caractériser pleinement les changements du virome intestinal.
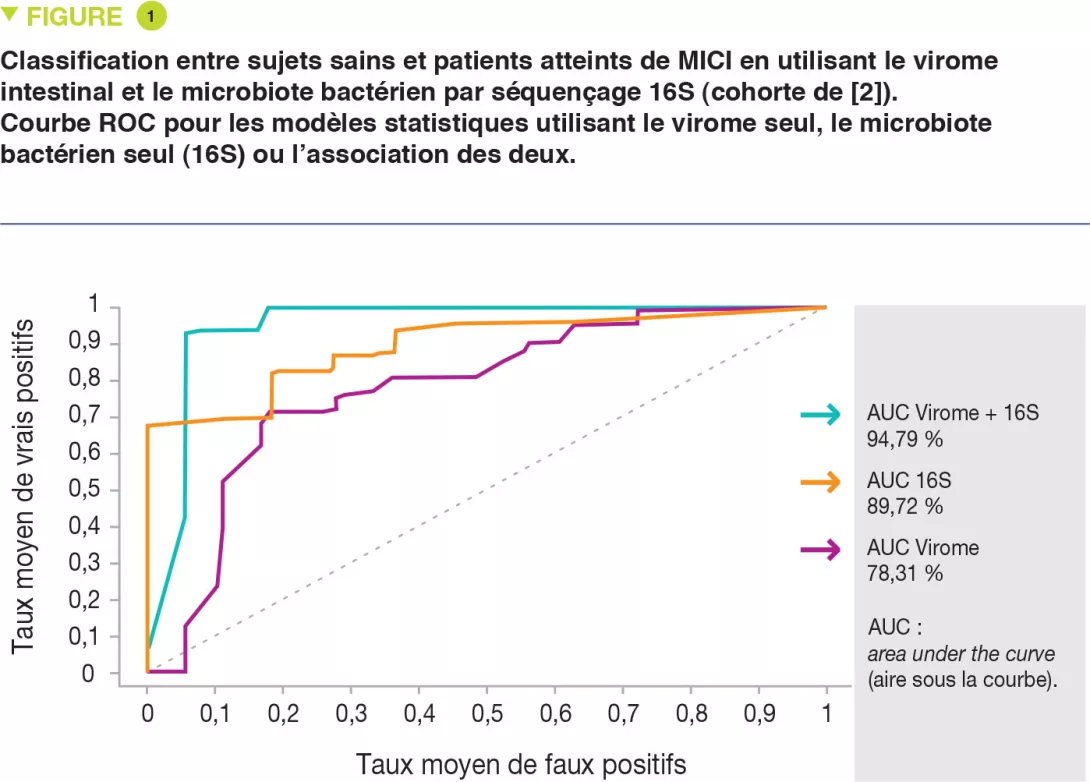
Dans le cadre de cette étude, les auteurs ont réanalysé un ensemble de données- clés publiées précédemment [3] obtenues à partir de sujets avec maladie de Crohn et rectocolite hémorragique et des témoins sains. Le problème du niveau élevé de variation interindividuelle a été surmonté en utilisant l’homologie des protéines et un algorithme particulier (cluster de Markov) pour regrouper les séquences virales en rangs taxonomiques supposés. De cette façon, il a été possible de décrire les changements de composition à travers le virome entier au-delà de la minorité connue. Les auteurs proposent que, contrairement au core virome des sujets sains constitué de phages virulents, le virome des patients atteints de MICI est altéré, moins stable et dominé par des phages tempérés. Ils montrent que les altérations du virome miment celles du bactériome et que lorsqu’elles sont utilisées ensemble, elles offrent une meilleure efficacité pour différencier les patients atteints de MICI de sujets sains (Figure 1). Les résultats ont été validés dans une cohorte longitudinale de patients atteints de RCH. Cette approche, indépendante des bases de données, pourrait être utilisée pour éclairer la matière noire virale de nombreuses études déjà publiées.
Ces résultats confirment que le virome intestinal est altéré dans les MICI et que celui- ci peut être associé à des éléments du microbiote bactérien ; il pourrait être utilisé comme biomarqueur diagnostique, voire pronostique. D’autre part, les altérations observées suggèrent que le virome pourrait être en partie responsable des altérations du microbiote bactérien observé dans les MICI. Dans ce sens, le virome intestinal pourrait donc être une future cible ou un futur outil thérapeutique dans les MICI.
En utilisant une méthode indépendante des bases de données, le virome intestinal est étudié dans sa globalité alors qu’habituellement, seule une minorité est analysée. Alors que le core virome des sujets sains est constitué de phages virulents, le virome des patients atteints de MICI est altéré, moins stable et dominé par des phages tempérés. Le virome associé au microbiote bactérien permet de différencier les patients atteints de MICI de sujets sains de manière plus efficace qu’en utilisant exclusivement le virome ou le microbiote bactérien. Ces résultats ouvrent de nouvelles perspectives pour l’utilisation du virome comme biomarqueur ou cible thérapeutique dans les MICI.
Par le Pr. Markku Voutilainen
Faculté de médecine de l’Université de Turku ; gastro-entérologie, Hôpital universitaire de Turku, Finlande
Les microbiotes intestinal et vaginal pourraient-ils prédire le stade de l’endométriose ?
D’après cette étude, aucune différence n’a été détectée entre les échantillons des patientes et ceux des sujets témoins sains en termes de phases folliculaire et menstruelle du cycle menstruel. Au niveau individuel, la répartition des types d’états communautaires (community state types, CST) était significativement différente entre les deux phases. Chez les patientes, les unités taxonomiques opérationnelles (Operational Taxonomic Units, OTU) du genre Anaerococcus (en particulier A. lactolyticus et A. degenerii) étaient significativement différentes entre les stades 1-2 et les stades 3-4 de la maladie (classification révisée de l’American Society for Reproductive Medicine, rASRM). Les auteurs concluent que le microbiote vaginal pourrait prédire le stade de la maladie.
La dysbiose vaginale pourrait être liée à l’apparition, à la progression et à la stabilité du cancer du col de l’utérus (CCU), mais on ne sait pas si la relation entre les deux est causale ou corrélative.
Afin de déterminer si les bactéries cervico-vaginales ont également un effet anticancéreux, l’utilisation des micro-organismes dans le traitement du cancer (en particulier le CCU) a été discutée dans cet article : probiotiques, immunothérapie à base de bactéries, toxines et spores bactériennes, vecteurs en thérapie génique, et inhibiteurs de l’angiogenèse tumorale. Certaines bactéries inhibent le CCU en activant les lymphocytes NK et la maturation des cellules dendritiques. Les autres mécanismes sont la production de composés cytotoxiques, la régulation de la différenciation des cellules immunitaires, et l’inhibition de la migration des cellules cancéreuses. En conclusion, les bactéries génétiquement modifiées pourraient constituer un traitement efficace du CCU à l’avenir. Des études portant sur une taille d’échantillon plus importante sont nécessaires pour évaluer cette option..
Retour de congrès
Par le Pr. Kristin Verbeke
Laboratoire Digestion et Absorption, Centre de recherche translationnelle sur les troubles gastro-intestinaux (TARGID), Département de médecine clinique et expérimentale, Louvain, Belgique

C’est dans des circonstances exceptionnelles en raison de l’épidémie de Covid-19 que le Gut Microbiota for Health World Summit de cette année s’est tenu à Madrid. Dans la mesure où de nombreux participants n’ont pas pu assister physiquement à l’événement, ce retour de congrès est un peu plus approfondi que ceux habituellement présentés dans cette Newsletter.
La 9e édition de ce congrès a abordé différents aspects du microbiote, notamment les facteurs alimentaires et non alimentaires qui façonnent le microbiote intestinal ainsi que le rôle du microbiote intestinal dans la fonction cérébrale et la modulation du système immunitaire. Lors d’une conférence inaugurale, Colin Hill (Cork, Irlande) a évoqué les obstacles à la traduction clinique de la recherche sur le microbiote. Il a appelé à adopter les définitions des consensus et à utiliser un langage précis. L’objectif est une approche plus rigoureuse, davantage basée sur la quantification exacte de bactéries plutôt que sur des proportions et des abondances relatives. Pour cela, il faut prendre en compte la complexité liée à l’individualité du microbiote et maîtriser celle des chiffres afin de ne pas noyer le lecteur.
Il a également fait remarquer que le choix de la méthodologie, notamment entre méthodes biologiques et in silico, affecte significativement les résultats.
Par exemple, le microbiote fécal n’est qu’une approximation très vague du microbiote intestinal, la durée du transit intestinal affecte la composition du microbiote et le passage d’une méthode in silico à une méthode in vivo n’est pas toujours simple. La recherche sur le microbiote devrait être une science appliquant des normes strictes plutôt qu’un système de croyances.
Des études transversales antérieures indiquaient déjà que la composition du microbiote fécal dépendait du régime alimentaire. En effet, les personnes qui suivent un régime alimentaire à base de plantes ont un microbiote plus diversifié, avec une proportion plus importante de bactéries productrices d’acides gras à chaîne courte (AGCC) que les personnes qui ont une alimentation occidentale riche en glucides raffinés et en graisses. Le passage d’une alimentation américaine standard à une alimentation végétale a modifié le microbiote de sujets obèses et considérablement amélioré leurs paramètres métaboliques, comme l’a indiqué Hana Kahleova (Washington DC, États- Unis). Les participants qui sont passés à une alimentation végétale pendant 16 semaines ont perdu 5,8 kg, dont environ deux tiers de graisses, et leur sensibilité à l’insuline était améliorée rapport au groupe témoin qui n’a pas adapté son régime alimentaire. Les taux fécaux de Bacteroidetes et de Faecalibacterium prausnitzii ont augmenté avec l’alimentation végétale, alors que les espèces Bacteroides fragilis ont diminué avec les deux régimes, mais dans une moindre mesure avec l’alimentation végétale. En outre, les modifications de la composition bactérienne étaient corrélées aux modifications des paramètres métaboliques.

Comme l’a souligné Rinse K. Weersma (Groningue, Pays-Bas), les médicaments interagissent avec le microbiote intestinal de différentes manières. Certains médicaments, comme les inhibiteurs de la pompe à protons (IPP) affectent la composition et la fonction du microbiote. L’augmentation locale du pH intestinal provoquée par la prise d’IPP entraîne une « oralisation » du microbiote intestinal du fait de la pénétration des bactéries d’origine buccale plus en profondeur dans le tractus gastro-intestinal. La metformine, un antidiabétique oral, affecte également la composition du microbiote intestinal avec un nombre accru d’Akkermansia muciniphila et une plus grande production d’AGCC, ce qui contribue à son effet antihyperglycémiant. Les immunothérapies n’ont pas directement d’impact sur le microbiote, mais comme celui-ci est impliqué dans l’homéostasie immunitaire, il détermine indirectement la réponse à ces médicaments anticancéreux. De plus, le microbiote modifie également l’activité de certains médicaments en les activant ou en les inactivant, ou alors en influençant leur toxicité. Par exemple, la transformation de la lévodopa par le microbiote intestinal réduit sa biodisponibilité pour le cerveau, ce qui pourrait en partie expliquer la variabilité des réponses des patients à ce médicament. Pour être actif, le promédicament sulfasalazine doit être clivé par azo-réduction bactérienne dans le côlon en 5-ASA et en sulfapyridine, alors que la digoxine, un glucoside cardiotonique, est inactivée par métabolisme microbien. Pour finir, la transformation par le microbiote intestinal de l’antiviral oral brivudine en bromovinyluracile joue un rôle dans la toxicité de ce médicament.
Athanasios Typas (Heidelberg, Allemagne) a souligné l’effet majeur des médicaments non antibiotiques sur le microbiote. Un criblage in vitro de 1 200 médicaments commercialisés visà- vis de 40 souches bactériennes intestinales représentatives a montré qu’au moins un quart des médicaments non antibiotiques ayant pour cible des cellules humaines inhibaient au moins une souche [1]. Cette inhibition in vitro se reflétait dans les effets secondaires de ces médicaments chez l’homme et concordait avec les essais cliniques existants, ce qui montre la pertinence de cette stratégie de criblage. Il est intéressant de noter qu’il existait un chevauchement important au niveau de la sensibilité des souches bactériennes intestinales aux médicaments ciblant des cellules humaines et aux antibiotiques, ce qui a été attribué au fait que les mêmes pompes, transporteurs et mécanismes de détoxification sont utilisés pour les deux groupes de médicaments. Ces résultats suggèrent que la polymédication pourrait constituer un facteur important de résistance aux antibiotiques.
Ces 30 dernières années, le traitement des maladies mentales a très peu progressé. John Cryan (Cork, Irlande) a expliqué que le microbiote intestinal pourrait constituer une nouvelle cible pour améliorer la santé mentale, même si ce domaine n’en est qu’à ses balbutiements, car le microbiote intestinal affecte la santé mentale à différents stades de la vie. Le mode d’accouchement, qui a des conséquences sur le microbiote intestinal, a été associé à des troubles neurodéveloppementaux [2]. Les souris nées par césarienne présentent une réponse accrue au stress, une plus grande anxiété et des déficiences en matière de sociabilité. Ces effets peuvent être inversés en agissant sur le microbiote intestinal. Le fait que les souris axéniques présentent également un développement cérébral inapproprié, notamment avec des déficiences au niveau de la mémoire de la peur, une augmentation des douleurs viscérales et des déficiences sociales, confirme le rôle du microbiote gastro-intestinal. De plus, au début de l’adolescence, le cerveau est sensible aux signaux microbiens. Les souris ayant reçu une alimentation riche en graisses pendant l’adolescence présentaient des différences durables en termes de composition du microbiote intestinal à l’âge adulte, ainsi que des différences au niveau de l’expression de gènes en lien avec la neuro-inflammation ou la neurotransmission, même si aucun trouble du comportement avéré n’a été observé à l’âge adulte [3]. Chez des souris mâles âgées, des modifications du microbiote pour adopter un profil précédemment associé à des maladies inflammatoires ont été corrélées avec une plus grande perméabilité intestinale, une inflammation périphérique et des troubles du comportement, notamment des déficiences au niveau de la mémoire spatiale et un comportement plus anxieux.
Souvent appelée « hormone du bonheur », la sérotonine a en fait une fonction biologique beaucoup plus complexe. Elle est en effet impliquée dans la densité osseuse ainsi que dans les fonctions neuronale, plaquettaire et gastro-intestinale, ce qui en fait une option intéressante pour améliorer la santé. La sérotonine est produite en majeure partie dans les tissus gastro-intestinaux. Jonathan Lynch (Los Angeles, États-Unis) a indiqué que le microbiote intestinal joue un rôle essentiel dans la régulation de la production de sérotonine par l’hôte. En particulier, les bactéries sporulées indigènes stimulent la biosynthèse de sérotonine par la production de métabolites solubles qui transmettent directement les signaux aux cellules coliques. Cette induction de sérotonine médiée par les bactéries régule la motilité gastro-intestinale et la fonction plaquettaire chez la souris [4]. En outre, les concentrations de sérotonine dans la lumière intestinale modulent également la colonisation bactérienne dans l’intestin. L’abondance relative des bactéries sporulées, en particulier de Turicibacter sanguinis, augmente quand les taux de sérotonine dans la lumière intestinale sont élevés. T. sanguinis exprime un récepteur homologue au SERT (transporteur de la sérotonine des mammifères) qui permet d’importer la sérotonine, entraînant l’expression de facteurs de sporulation et de transporteurs membranaires. Ces effets sont inversés par l’exposition à la fluoxétine, un inhibiteur de la recapture de la sérotonine.
À la naissance, les nouveau-nés acquièrent des micro-organismes par transmission verticale de leur mère. Cette colonisation postnatale serait le principal stimulus pour le développement et la maturation du système immunitaire. À l’aide d’un modèle de colonisation transitoire de souris gravides, Kathy McCoy (Calgary, Canada) a démontré que, déjà pendant la gestation, le microbiote intestinal de la mère façonne la fonction du système immunitaire des petits. Les souriceaux axéniques nés de mères colonisées de manière transitoire présentaient des taux plus élevés de cellules de l’immunité innée dans l’intestin et une expression accrue de gènes codant pour les peptides antibactériens épithéliaux et le métabolisme des molécules microbiennes par rapport aux petits nés de mères axéniques [5]. Cette éducation du système immunitaire médiée par le microbiote intestinal maternel repose sur les anticorps de la mère transmis aux petits pendant la gestation et par l’intermédiaire du lait. De plus, le microbiote intestinal de la mère protège les petits d’une inflammation excessive. L’administration de lipopolysaccharides (LPS) a déclenché une réponse inflammatoire très importante chez les petits nés de mères axéniques, alors que cette réponse était atténuée chez les petits de mères colonisées.
La période comprise entre la naissance et le sevrage, c’est-à-dire au moment de l’introduction d’une alimentation plus diversifiée, est importante pour l’ontogenèse du système immunitaire, comme l’a souligné Gérard Eberl (Paris, France). L’expansion du microbiote intestinal qui se produit lors du sevrage induit une réponse immunitaire forte associée à l’induction des lymphocytes T régulateurs [6]. L’exposition de souris axéniques aux micro-organismes avant le sevrage entraîne cette réaction immunitaire (normale), alors qu’aucune réaction ne se produit quand les souris ne sont exposées aux micro-organismes qu’après le sevrage, ce qui indique que le système immunitaire doit être exposé aux micro-organismes dans une fenêtre de temps spécifique.
L’empreinte pathologique générée a entraîné plus tard une susceptibilité accrue aux pathologies immunitaires. Le fonctionnement de la mémoire du système immunitaire doit encore être éclairci. Des données dans la littérature suggèrent une régulation épigénétique des gènes immunitaires et l’empreinte de l’expression des gènes dans les cellules myéloïdes ou stromales. L’hypothèse la plus probable est qu’une empreinte soit retrouvée dans de nombreuses cellules différentes, mais la signification de ce phénomène doit encore être analysée.
Article commenté - Rubrique enfant
Par le Pr. Emmanuel Mas
Gastro-entérologie et nutrition, Hôpital des Enfants, Toulouse, France
Commentaire de l’article original de Lindfors et al. (Gut 2019) [1]
Une consommation de gluten plus élevée, de fréquentes infections gastrointestinales et les adénovirus, entérovirus, rotavirus et réovirus ont été suggérés comme déclencheurs environnementaux de la maladie coeliaque (MC). Cependant, le lien entre la quantité de gluten ingérée et les expositions virales dans le développement de la maladie reste inconnu. Le but de cette étude était de savoir si des expositions virales distinctes, seules ou associées au gluten, augmentaient le risque d’auto-immunité de la MC chez les enfants génétiquement prédisposés. Elle conclut que l’exposition fréquente à des entérovirus entre 1 et 2 ans est effectivement associée à un risque accru d’auto-immunité de MC, l’effet cumulatif de l’interaction entre les entérovirus et une consommation de gluten plus élevée étant en cause.
La maladie coeliaque est une pathologie auto-immune qui survient chez des personnes génétiquement prédisposées de génotype HLA DQ2 et/ou DQ8 positif. Elle est caractérisée par la présence d’une atrophie villositaire et d’une infiltration lymphocytaire au niveau de l’épithélium de l’intestin grêle. Le gluten présent dans l’alimentation induit une réponse auto-immune dirigée contre la transglutaminase tissulaire. L’apparition d’anticorps anti-transglutaminase (TG) définit l’auto-immunité de la maladie coeliaque.
L’augmentation de l’incidence des maladies auto-immunes fait suspecter le rôle de facteurs environnementaux dans leur pathogénèse. Des études observationnelles suggèrent que des infections virales pourraient induire une rupture de la tolérance orale du gluten et le développement de la maladie coeliaque.
Il s’agit d’une étude cas-contrôle nichée dans la cohorte de naissance TEDDY (The Environmental Determinants of Diabetes in the Young) qui a inclus 8 676 enfants avant l’âge de 4 mois et demi avec un suivi de 15 ans. L’objectif principal de cette cohorte est d’identifier les facteurs génétiques et environnementaux associés au diabète de type 1 et à la maladie coeliaque. Après appariement en fonction des antécédents familiaux de diabète de type 1, sexe et site d’inclusion dans l’étude, 83 paires (enfants avec prédisposition et cas contrôle) ont été retenues dans l’analyse finale pour lesquelles les données du virome fécal étaient disponibles après introduction du gluten. Parmi ces paires, 16 avaient des antécédents familiaux de diabète de type 1. Lors du suivi, 28 des cas d’auto-immunité de maladie coeliaque ont développé la maladie.
De 3 mois à 2 ans, des échantillons de selles ont été recueillis tous les mois ; une recherche d’entérovirus, adénovirus, astrovirus, norovirus, réovirus et rotavirus a été effectuée. Tous les 3 mois, un questionnaire alimentaire était adressé pour recueillir les informations sur l’allaitement maternel et l’âge d’introduction des aliments contenant du gluten. Un relevé alimentaire sur 3 jours a permis de calculer les quantités de gluten ingérées à 6, 9, 12, 18 et 24 mois.
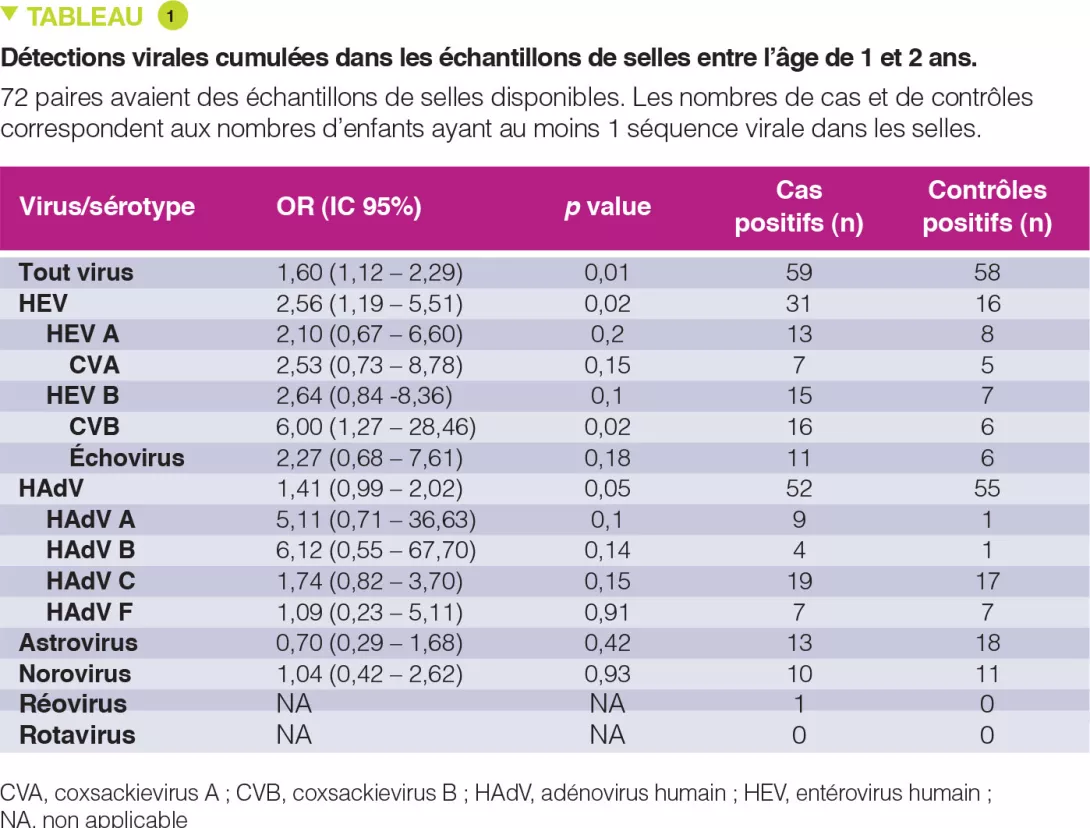
Le pourcentage d’échantillons de selles positives pour n’importe quel virus variait de 22 à 50 %, sans pic lié à l’âge, et celui des entérovirus de 0 à 21 % après 6 mois. Entre 1 et 2 ans, des entérovirus ont été détectés chez 31 cas vs 16 contrôles (Tableau 1). Le nombre cumulé d’échantillons de selles positifs pour n’importe quel virus était associé à un risque augmenté d’auto-immunité de maladie coeliaque (OR 1,60 ; p = 0,01), avec une association la plus forte pour les entérovirus (OR 2,56 ; p = 0,02).
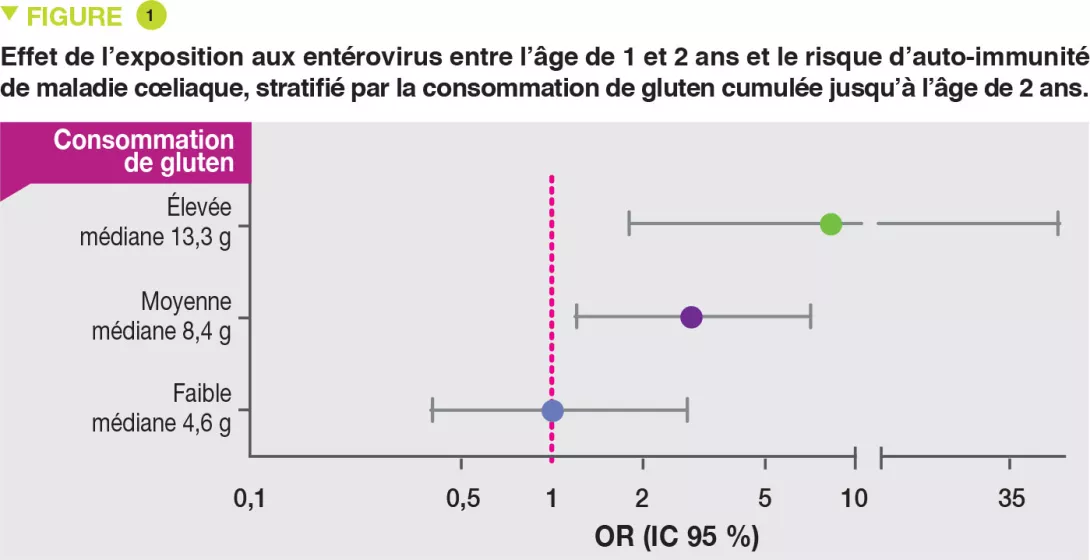
Le risque d’auto-immunité de maladie coeliaque n’était pas augmenté par les infections virales survenant après l’âge d’introduction du gluten lorsque l’allaitement maternel était poursuivi. En revanche, après sevrage de l’allaitement maternel, sur les échantillons de selles recueillis entre 1 et 2 ans alors que le gluten avait été introduit, il existait une association entre le nombre cumulé de virus retrouvés (OR 1,41 ; p = 0,05) ainsi que d’entérovirus retrouvés (OR 2,47 ; p = 0,03) et le risque d’auto-immunité de maladie coeliaque. Il y avait une interaction significative entre la présence d’entérovirus retrouvés entre 1 et 2 ans et la quantité ingérée de gluten jusqu’à 2 ans sur le risque d’auto-immunité de maladie coeliaque (p = 0,03). Ce risque augmentait avec la quantité de gluten ingérée : élevée (OR 8,3), moyenne (OR 2,9) et faible (OR 1,0) (Figure 1).
Les facteurs environnementaux jouent un rôle dans la maladie cœliaque
L’exposition aux entérovirus est un facteur de risque de développer des anticorps anti-transglutaminases chez des enfants HLA DQ2 et/ou DQ8 positifs
Ce risque est potentialisé par des apports importants en gluten dans l’alimentation
Les résultats de cette étude ont un intérêt pour prévenir l’apparition d’auto-anticorps de la maladie coeliaque chez des enfants à risque. Pour cela, il faudrait faire attention aux quantités de gluten ingérées, notamment en cas d’exposition aux entérovirus, surtout lorsque l’enfant n’est plus sous allaitement maternel.
Cette étude a montré qu’il existait une association entre les expositions digestives à entérovirus et le risque d’autoimmunité de maladie coeliaque chez des enfants génétiquement prédisposés. Ce risque est majoré par la quantité de gluten ingérée.
Retour de congrès
Par le Pr Magnus Simrén
Département de Médecine moléculaire et clinique, Institut de Médecine Académie Sahlgrenska, Université de Göteborg, Göteborg, Suède

En raison de la pandémie qui sévit actuellement, l’édition 2020 du congrès UEG Week s’est tenue pour la première fois en mode virtuel. Mais comme les années précédentes, cet événement a été l’occasion de présenter de nombreux abstracts de qualité, dont une grande partie était consacrée au rôle des micro-organismes dans la santé et la maladie.
Le microbiote intestinal a été associé à un grand nombre de maladies, mais on n’a toujours pas défini clairement ce qu’est un microbiote « sain » ou « malade ». Une vaste étude néerlandaise en population (OP178 R Gacesa et al.) a mis en évidence l’existence de profils microbiens communs entre différentes maladies (notamment les maladies inflammatoires chroniques de l’intestin [MICI], le syndrome de l’intestin irritable [SII], l’asthme, le diabète et les troubles mentaux), permettant ainsi de définir des groupes de fonctions et de micro-organismes intestinaux liés à la santé et à la maladie. En effet, le microbiote associé aux maladies était caractérisé par une augmentation significative de la prévalence et de l’abondance de pathogènes opportunistes des genres Clostridium, Gordonibacter et Eggerthella, par une réduction du catabolisme des glucides et de la synthèse des acides aminés et des vitamines, et par une augmentation de la synthèse des acides gras à chaîne longue. Le microbiote sain montrait quant à lui une abondance importante de bactéries commensales productrices de butyrate des genres Alistipes, Roseburia, Faecalibacterium et Butyrivibrio. Les auteurs ont également montré que le microbiote était principalement façonné par l’environnement et le mode de vie, et ils ont donc conclu que l’on peut promouvoir la modulation du microbiote par le biais de l’amélioration du régime alimentaire, du mode de vie et de l’environnement ainsi que de l’utilisation de probiotiques pour améliorer la santé. Une étude de suivi longitudinale (OP201 L Chen et al.) a également montré que les variations microbiennes au cours du temps semblaient déterminées par les expositions environnementales et pouvaient avoir un impact sur la santé métabolique de l’hôte.
La restriction de la consommation de lactose constitue la pierre angulaire du traitement des troubles gastro-intestinaux (GI) chez les sujets présentant une malabsorption du lactose en raison d’un déficit en lactase. Cependant, les symptômes intestinaux tels que les flatulences, les ballonnements et les diarrhées qui surviennent après l’ingestion de lactose chez ces sujets sont de sévérité très variable, pour une raison encore indéterminée. Des analyses du Dutch Microbiome Project (OP177 MDF Brandao Gois et al.) ont permis de démontrer que le microbiote intestinal servait vraisemblablement de médiateur entre l’ingestion de produits laitiers et la survenue des symptômes intestinaux chez les sujets présentant un déficit en lactase, avec un rôle potentiel du genre Bifidobacterium. La modulation de la composition du microbiote intestinal pourrait donc avoir un impact sur la sensibilité aux produits laitiers chez les sujets présentant une malabsorption du lactose.
Bien que l’on n’ait pas encore déterminé exactement les mécanismes expliquant les symptômes GI liés à l’alimentation chez les patients atteints d’un SII, différents ajustements alimentaires permettent d’améliorer les symptômes GI dans des sous-groupes de patients. Une analyse post-hoc d’un essai clinique précédemment publié (P0786 E Colomier et al.) a mis en évidence des profils de facteurs psychologiques, nutritionnels et microbiens pouvant prédire la réponse à la fois au régime traditionnel basé sur les recommandations du NICE (National Institute for Health and Care Excellence) pour le SII et au régime pauvre en oligo-, di-, monosaccharides et polyols fermentescibles (FODMAP, Fermentable Oligo-, Di-, Monosaccharides, and Polyols) pour des symptômes spécifiques. Cela montre qu’il sera possible, dans un avenir proche, de personnaliser les conseils en matière de traitement alimentaire dans le SII.
Les micro-organismes intestinaux et leurs métabolites sont impliqués dans la physiopathologie d’un certain nombre de maladies intestinales, dont le SII et les MICI, comme le montrent plusieurs abstracts présentés au congrès UEG Week 2020. Dans les MICI, une vaste étude de cohorte a confirmé la présence d’une dysbiose intestinale dans la rectocolite hémorragique (RCH) et la maladie de Crohn (MC) (OP002 A Vich Vila et al.), et elle a montré que celle-ci se répercutait sur le profil métabolique fécal qui pourrait alors servir de biomarqueur pour faire la distinction entre une MICI et une absence de MICI et entre la RCH et la MC. En effet, les métabolites liés à la synthèse des sphingolipides étaient plus nombreux dans les MICI, alors qu’on observait une diminution des métabolites acides gras. Une étude de preuve du concept (OP045 L Oliver et al.) a également montré qu’une combinaison de quatre marqueurs microbiens (Faecalibacterium prausnitzii et l’un de ses phylogroupes (PHG-II), Ruminococcus sp. et Methanobrevibacter smithii) pouvait prédire la réponse au traitement anti-TNF avec une valeur prédictive positive de 100 % et une valeur prédictive négative de 75 %. Cela montre que, dans un avenir proche, les analyses du microbiote pourront être utilisées pour personnaliser le traitement dans les MICI. Le rôle du microbiote intestinal dans le SII a été souligné dans plusieurs abstracts, notamment dans une étude confirmant les effets positifs à long terme de la transplantation de microbiote fécal dans le SII (OP059 M El-Salhy et al.), qui a été associée à des modifications du profil des bactéries et acides gras à chaîne courte fécaux et à une augmentation des cellules entéroendocrines (P0783 M El Salhy et al.). Une autre étude a également mis en évidence l’existence d’un profil microenvironnemental intestinal distinct dans le SII en lien avec le trouble du transit prédominant du patient (P0651 C Iribarren et al.), la distinction entre les sujets atteints du SII et les sujets sains et entre les sous-types de SII (SII avec diarrhée versus SII avec constipation) étant principalement déterminée par les métabolites impliqués notamment dans le métabolisme des acides aminés et certaines fonctions cellulaires et moléculaires. Il semble donc que l’action des micro-organismes soit plus importante que la composition en tant que telle. Des abstracts ont également porté sur les modèles animaux pertinents dans la physiopathologie du SII. Ces études ont souligné l’importance du microbiote intestinal dans le développement des interactions anormales entre le cerveau et l’intestin (P0052 M Constante et al.), ainsi que le rôle du stress dans l’induction de la dysbiose intestinale et de l’hypersensibilité viscérale (OP056 C Petitfils et al.). Ces études sont essentielles pour nous permettre de mieux comprendre les interactions intestin-cerveau dans le SII et le rôle des micro-organismes intestinaux et de leurs métabolites dans ces interactions, et s’inscrivent parfaitement dans la démarche de qualifier désormais le SII et les autres troubles GI fonctionnels de « troubles des interactions intestin-cerveau ».
Pour finir, des études sur le microbiote intestinal dans les maladies extra-intestinales ont également été présentées. Des altérations du microbiote intestinal ont été mises en évidence chez des greffés rénaux et hépatiques (OP180 JC Swarte et al. et OP112 y Li et al.). Les patients souffrant d’insuffisance rénale terminale étaient caractérisés par une faible diversité microbienne intestinale et une richesse accrue en facteurs de virulence et en gènes de résistance aux antibiotiques. La diversité microbienne a encore diminué après la transplantation rénale et la composition du microbiote intestinal ne s’est pas rétablie. En outre, les médicaments immunosuppresseurs ont eu un effet majeur sur la composition du microbiote intestinal. Les auteurs ont concédé que ces modifications pourraient avoir des implications importantes pour le devenir de la transplantation rénale. Des observations similaires concernant la diversité microbienne, la composition du microbiote intestinal et l’effet des immunosuppresseurs ont également été faites chez les transplantés hépatiques et étonnamment, la diversité microbienne a été associée à la survie post-transplantation, ce qui pourrait en faire un biomarqueur ou une cible thérapeutique.
En résumé, les abstracts présentés à l’édition 2020 du congrès UEG Week montrent clairement que le microbiote intestinal joue un rôle très important dans plusieurs maladies et dans la santé. La meilleure compréhension du rôle des micro-organismes intestinaux et de leurs métabolites dans différentes maladies a un impact important sur la prise en charge aujourd’hui et son influence sera encore plus grande dans un avenir proche.
Article commenté - rubrique adulte
Par le Pr. Harry Sokol
Gastro-entérologie et nutrition, Hôpital Saint-Antoine, Paris, France

Commentaire de l’article de Ghosh et al. Gut 2020 [1]
Le vieillissement s’accompagne d’une détérioration de multiples fonctions organiques et d’une inflammation, qui, ensemble, contribuent à la fragilité. Il a déjà été montré, par les auteurs et d’autres équipes, que la fragilité était associée aux altérations du microbiote intestinal, d’autant plus dans un contexte de régime alimentaire peu varié. Le régime méditerranéen est reconnu comme étant associé à une bonne santé. Ici, les auteurs ont cherché à savoir si 12 mois de régime méditerranéen, reconnu associé à une bonne santé, pouvait modifier le microbiote intestinal, réduire la fragilité et améliorer les fonctions cognitives. Le profil du microbiote intestinal a été établi chez des sujets non fragiles ou pré-fragiles dans cinq pays européens avant et après l’adoption d’un régime méditerranéen pendant 1 an adapté aux sujets âgés (régime NU-AGE). Les résultats montrent qu’il est possible d’améliorer le régime alimentaire habituel afin de moduler le microbiote intestinal, ce qui favorise un vieillissement en meilleure santé.
La fragilité qui accompagne le vieillissement implique la défaillance de plusieurs systèmes physiologiques et une activation persistante de la réponse inflammatoire immunitaire innée. La fragilité peut inclure le développement d’une inflammation chronique de bas grade, une altération de la fonction cognitive, une sarcopénie et le développement de maladies chroniques comme le diabète et l’athérosclérose. La modification des schémas alimentaires telle que l’adoption d’un régime méditerranéen a été suggérée comme une stratégie thérapeutique majeure pour lutter contre la fragilité [2]. Le régime méditerranéen se caractérise par une consommation accrue de légumes, légumineuses, fruits, noix, huile d’olive, poisson et une faible consommation de viande rouge, produits laitiers et graisses saturées. L’adhésion à ce type d’alimentation est associée à une mortalité réduite et une activité antioxydante accrue, ainsi qu’à une réduction de l’incidence de plusieurs maladies et de l’inflammation.
Plusieurs études ont montré que l’adoption de ce régime est liée à une diminution de la fragilité. Au-delà de l’association négative avec la maladie, une adhésion plus élevée au régime méditerranéen a été associée à des changements bénéfiques dans la composition du microbiote intestinal (réduction de l’abondance des protéobacteries, augmentation des niveaux de production d’acides gras à chaîne courte [AGCC]). Pourtant dans le monde, peu de sujets âgés suivent ce régime et un grand nombre pâtissent d’une alimentation restreinte associée à un microbiote intestinal peu diversifié. Changer cela représente un défi majeur, notamment concernant les personnes en institution.
Dans des travaux antérieurs, les auteurs ont utilisé une analyse bio-informatique pour identifier des taxons microbiens spécifiques qui sont progressivement perdus lors de la transition d’un microbiote diversifié de sujets sains à un microbiote peu diversifié de sujets fragiles. Dans une étude récente portant sur une intervention alimentaire de 6 mois avec supplémentation par 5 prébiotiques (jusqu’à 20 g/jour) chez des personnes âgées, plusieurs taxons microbiens ont été modifiés, mais aucun changement n’a été noté dans la diversité globale du microbiote ou concernant les marqueurs inflammatoires. Les auteurs ont donc estimé qu’une intervention diététique plus drastique était nécessaire. Le projet d’intervention alimentaire NU-AGE visait à étudier l’effet de l’administration d’un régime méditerranéen personnalisé pendant 12 mois dans une large cohorte de plus de 1 200 personnes âgées de 65 à 79 ans, réparties dans cinq pays européens. Une association significative a été observée entre une adhésion accrue au régime méditerranéen et une capacité cognitive globale et une mémoire épisodique améliorées [3].
De plus, il a été démontré qu’une adhésion plus élevée réduisait le taux de perte osseuse chez les personnes atteintes d’ostéoporose et améliorait la fonction immunitaire innée, la tension artérielle et la rigidité artérielle [4-6]. Dans la présente étude, les auteurs ont analysé le microbiote intestinal d’un sous-groupe de sujets de l’étude.
Globalement, 612 sujets ont été analysés (289 témoins : 145 hommes, 144 femmes et 323 sous régime méditerranéen : 141 hommes, 182 femmes). À l’état basal, des différences en termes nutritionnel et de microbiote étaient observées entre les différents pays. Des associations entre le régime méditerranéen et le microbiote intestinal ont été mises en évidence. Parmi les taxons associés à une bonne adhésion au régime méditerranéen (DietPositive), on trouve une surreprésentation d’espèces comme par exemple Faecalibacterium prausnitzii, Eubacterium et Roseburia, dont une majorité sont associées à une bonne santé (incluant la production d’AGCC et des effets anti-inflammatoires). Inversement, des taxons ont été déplétés en cas d’adhésion forte à ce régime et certains ont été associés avec le diabète de type 2, le cancer colorectal, la cirrhose ou les maladies inflammatoires chroniques de l’intestin. Ces résultats suggèrent collectivement que l’adhésion au régime méditerranéen peut moduler le microbiote dans une direction positivement associée à la santé.
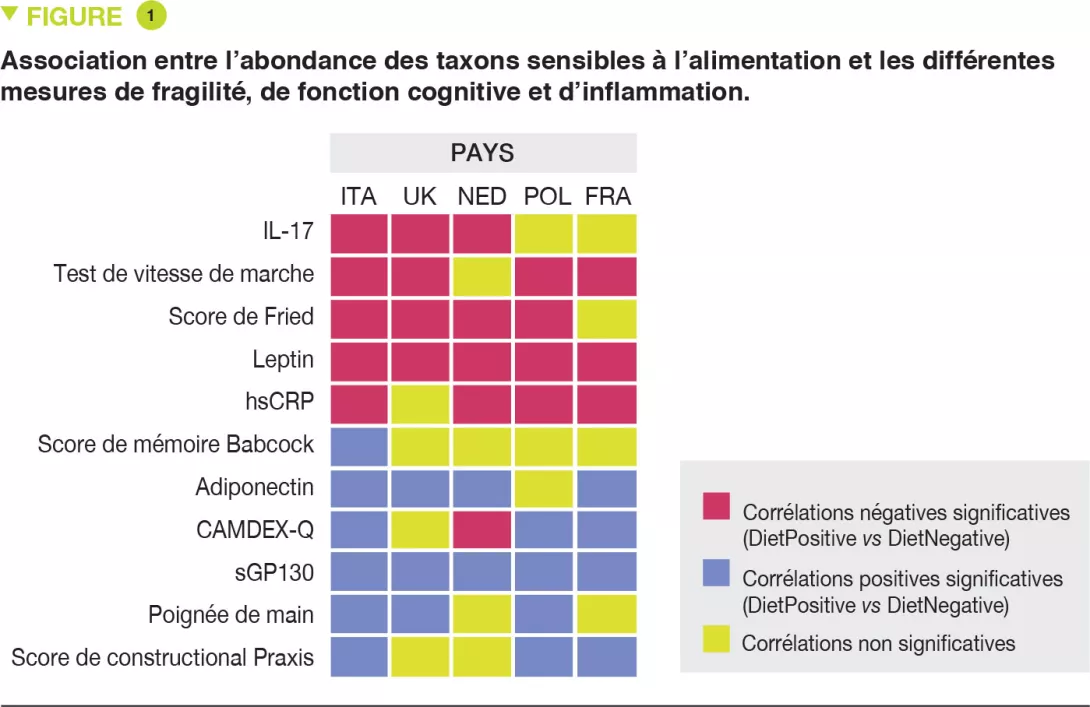
Enfin, les auteurs ont observé que l’abondance des taxons DietPositive étaient négativement corrélés à certains marqueurs inflammatoires (CRP ultrasensible – hsCRP et IL-17), et aux scores cliniques augmentés en cas de fragilité (scores de Fried, test de vitesse de marche). Au contraire, l’abondance de ces taxons était corrélée positivement à l’amélioration des fonctions cognitives (score de Constructional Praxis, Score de mémoire de Babcock) et à la réduction de fragilité (poignée de main) et de deux marqueurs anti-inflammatoires (adiponectine et sGP130). La tendance opposée était observée avec les taxons DietNegative (Figure 1). L’analyse des profils de métabolites microbiens inférés a indiqué que le changement de microbiote modulé par l’alimentation était associé à une augmentation de la production d’acides gras à chaîne courte/ramifiée et à une production plus faible d’acides biliaires secondaires, de p-crésols, d’éthanol et de dioxyde de carbone.
Le vieillissement est associé à une détérioration de nombreuses fonctions et à une inflammation qui conduisent à la fragilité
Un régime alimentaire méditerranéen induit une augmentation de l’abondance de taxons bactériens qui sont associés à une diminution de la fragilité
Le rôle d’une augmentation de la production de certains métabolites bactériens, notamment des AGCC est suggéré dans les effets positifs du régime
Ces résultats confirment que l’alimentation est un moyen efficace pour agir sur l’état de santé, au moins en partie, via une modulation du microbiote intestinal. On peut bien sûr conseiller d’adopter un régime méditerranéen chez les sujets âgés, mais la faisabilité d’une telle intervention nutritionnelle sur le long terme pose question. Cette étude ayant identifié des bactéries associées aux effets bénéfiques du régime méditerranéen, il ouvre la voie à leur utilisation sous forme de probiotiques de nouvelle génération. Ce type d’approche basée sur des bactéries issues du microbiote intestinal devrait être testé dans cette indication.
Cette étude met en lumière les interactions complexes entre alimentation, microbiote intestinal et santé. Elle suggère que les effets bénéfiques d’un régime méditerranéen sur la santé des sujets âgés passent, au moins en partie, par une modulation du microbiote intestinal.

"Bonne science - à lire absolument..." -@ethicos2013 (De Biocodex Microbiota Institute sur X)
1. Ghosh TS, Rampelli S, Jeffery IB, et al. Mediterranean diet intervention alters the gut microbiome in older people reducing frailty and improving health status: the NU-AGE 1-year dietary intervention across five European countries. Gut 2020 [Epub ahead of print].
2. An R, Wilms E, Masclee AAM, et al. Age-dependent changes in GI physiology and microbiota: time to reconsider? Gut 2018 ; 67 : 2213–22.
3. Marseglia A, Xu W, Fratiglioni L, et al. Effect of the NU-AGE diet on cognitive functioning in older adults: a randomized controlled trial. Front Physiol 2018 ; 9 : 349.
4. Jennings A, Cashman KD, Gillings R, et al. A Mediterranean-like dietary pattern with vitamin D3 (10 μg/d) supplements reduced the rate of bone loss in older Europeans with osteoporosis at baseline: results of a 1-y randomized controlled trial. Am J Clin Nutr 2018 ; 108 : 633-40.
5. Maijo M, Ivory K, Clements SJ, et al. One-year consumption of a Mediterranean-like dietary pattern with vitamin D3 supplements induced small scale but extensive changes of immune cell phenotype, co-receptor expression and innate immune responses in healthy elderly subjects: results from the United Kingdom arm of the NU-AGE trial. Front Physiol 2018 ; 9 : 997.
6. Jennings A, Berendsen AM, de Groot LCPGM, et al. Mediterranean-style diet improves systolic blood pressure and arterial stiffness in older adults. Hypertension 2019 ; 73 : 578-86.
Synthèse
Par le Pr. Francisco Guarner
Unité de recherche sur le système digestif, Hôpital université Vall d’Hebron, Barcelone, Espagne

Le mésusage massif des antibiotiques peut être à l’origine d’infections bactériennes multirésistantes, mais aussi de l’expansion de maladies chroniques non transmissibles. Les antibiotiques et les vaccins ont, plus que n’importe quelle autre innovation médicale, permis d’allonger l’espérance de vie. D’après des experts nommés par le British Medical Journal, les antibiotiques figurent parmi les découvertes médicales les plus reconnues[1]. L’introduction de mesures destinées à prévenir et à traiter les maladies infectieuses a donné lieu à des changements colossaux. En effet, les infections étaient auparavant à l’origine de 30 % des décès, principalement chez les enfants de moins de 5 ans ; à la fin du XXe siècle, elles étaient responsables de moins de 4 % de la mortalité seulement [1]. Cependant, deux gros sujets de préoccupation ont récemment surgi. Premièrement, les traitements d’un nombre croissant d’infections deviennent moins efficaces en raison d’une résistance aux antibiotiques. Ce phénomène constitue aujourd’hui une menace majeure pour la santé humaine, et son lien avec l’utilisation excessive des antibiotiques est bien documenté [2]. Deuxièmement, les antibiotiques, initialement destinés à détruire les micro-organismes pathogènes, ont des conséquences imprévues pour l’écosystème microbien humain, assorties de modifications qui sont parfois difficilement réversibles [3]. Le corps humain abrite un réseau complexe de micro-organismes appelé « microbiote », lequel joue un rôle important sur la santé. Du fait de l’altération du microbiote et de la perte de ses fonctions, les communautés microbiennes des personnes vivant dans les sociétés industrialisées pourraient s’avérer sous-optimales pour la santé [3]. La communauté médicale doit être sensibilisée à ces enjeux et les responsables des politiques de santé doivent formuler des recommandations claires dans ce domaine.
D’après le rapport 2018 de l’OMS [2], la quantité totale d’antibiotiques consommée par l’homme dépasse largement les 6 500 tonnes par an (données de 65 pays, hors Chine et États-Unis). En moyenne, 18 personnes sur 1 000 prennent chaque jour une dose définie d’antibiotiques, ce qui correspond à 139 millions de doses par jour. Cette moyenne est plus faible dans les pays africains (12 ‰) qu’en Europe (17,8 ‰) ou en Amérique (18,2 ‰), alors que les infections sont responsables de 36,6 % des décès en Afrique, mais seulement de 2,7 % ou 4,5 % en Europe ou en Amérique respectivement. Les pays émergents affichent toujours des taux élevés de mortalité par maladies infectieuses, mais des taux faibles de recours aux antibiotiques. Dans ces pays, un accès limité et le recours à des médicaments inadaptés ou à de mauvais schémas thérapeutiques peuvent contribuer à l’émergence d’infections résistantes comme la tuberculose
Dans les pays développés, jusqu’à la moitié des prescriptions d’antibiotiques peuvent être considérées comme inutiles [2]. Ce phénomène accélère le développement des résistances, et des souches multirésistantes de Pseudomonas aeruginosa, Escherichia coli, Klebsiella pneumoniae, Staphylococcus aureus, etc., sont de plus en plus nombreuses [2].
Acclamés comme l'une des avancées majeures de la médecine du XXème siècle, les antibiotiques ont sauvé des millions de vies. Mais ils ont aussi un impact sur notre microbiote en provoquant des dysbioses. Examinons de plus près ce rôle ambivalent.
Même si la plupart des antibiothérapies n’entraînent pas d’effets indésirables immédiats évidents, les dommages collatéraux sur la composition du microbiote intestinal et ses fonctions sont préoccupants [3]. La diarrhée constitue la complication la plus fréquemment associée aux antibiotiques et touche 15 à 25 % des patients. La plupart des diarrhées induites par les antibiotiques sont d’intensité légère et spontanément résolutives, mais de plus en plus de patients développent des formes plus sévères, notamment dues à Clostridioides difficile. Les troubles induits par les antibiotiques favorisent la germination des spores de C. difficile dans l’intestin, la prolifération de formes végétatives et la production de toxines, ce qui entraîne des lésions épithéliales et une colite. Le tableau clinique peut varier de diarrhées spontanément résolutives à un mégacôlon toxique ou une colite fulminante, voire au décès du patient [4].
Dès la naissance et pendant les trois premières années de vie, la diversité du microbiote intestinal augmente rapidement avant de se stabiliser vers un état « adulte ». Ensuite, l’essentiel de sa composition est stable, mais l’abondance de différentes bactéries peut fluctuer en réponse à des facteurs extérieurs (alimentation, médicaments, voyages, etc.). Des études ont montré que les antibiotiques provoquent des variations très importantes des abondances relatives. Dans le microbiote de patients sous β-lactamines ou quinolones, le nombre de taxa partagés par la plupart des sujets est passé de 29 à 12, le nombre total de taxa observés a diminué de 25 % et le genre dominant n’était plus Faecalibacterium, mais Bacteroides [5].
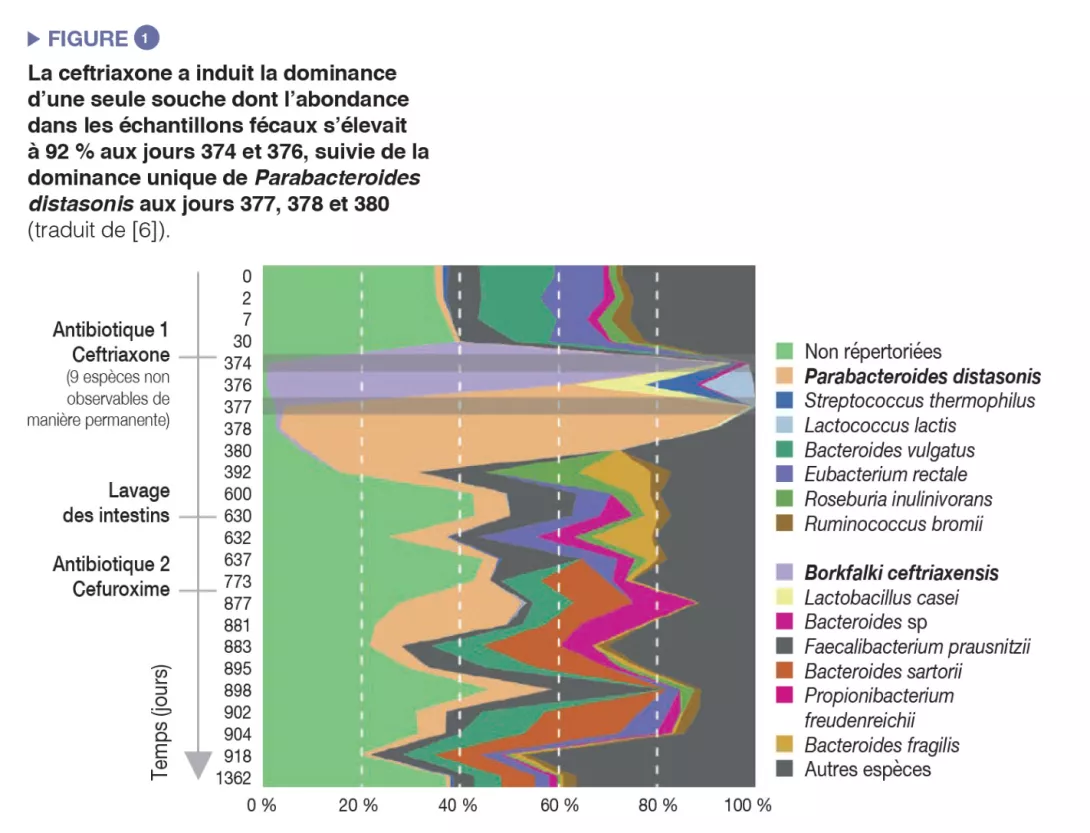
L’administration d’antibiotiques a induit une diminution de la diversité microbienne (perte de richesse de l’écosystème) et une prolifération d’espèces résistantes, ce qui a entraîné une augmentation globale de la charge microbienne (nombre de bactéries par gramme de selles) [5]. Des cas extrêmes de prolifération induite par les antibiotiques ont été rapportés : une souche unique peut devenir prédominante et atteindre une abondance relative de 92 % dans des échantillons fécaux après administration intraveineuse de ceftriaxone [6] (Figure 1).
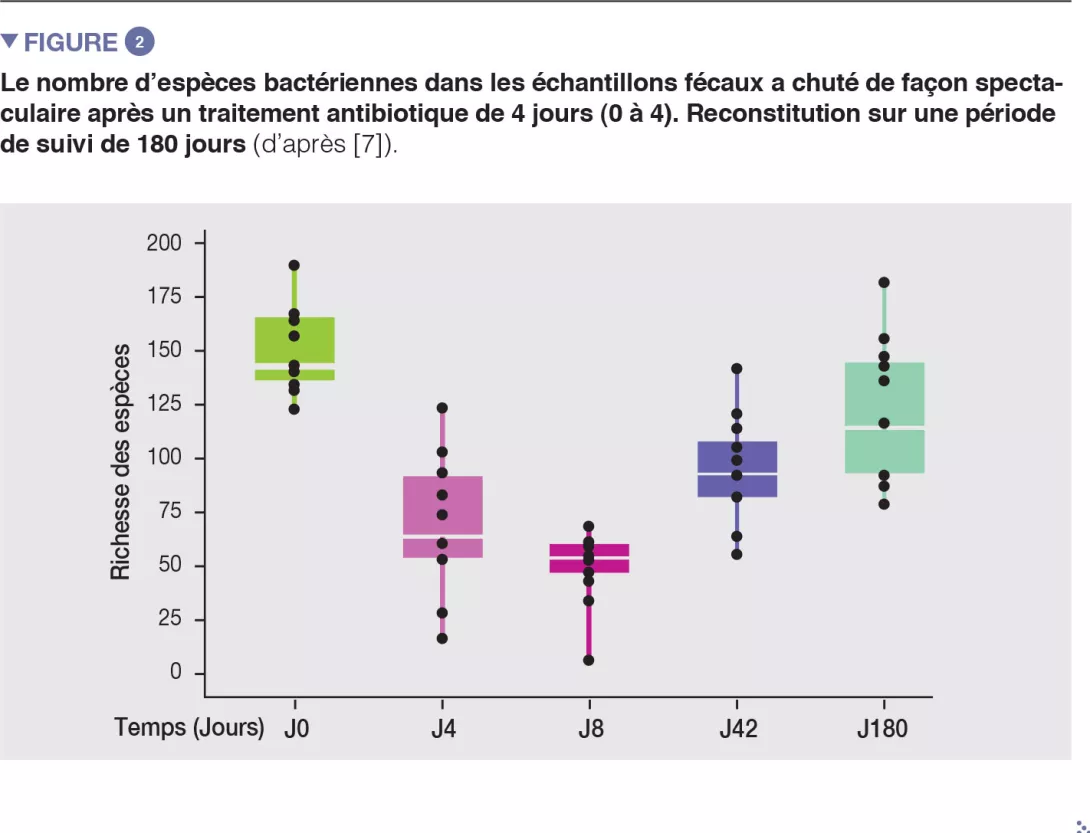
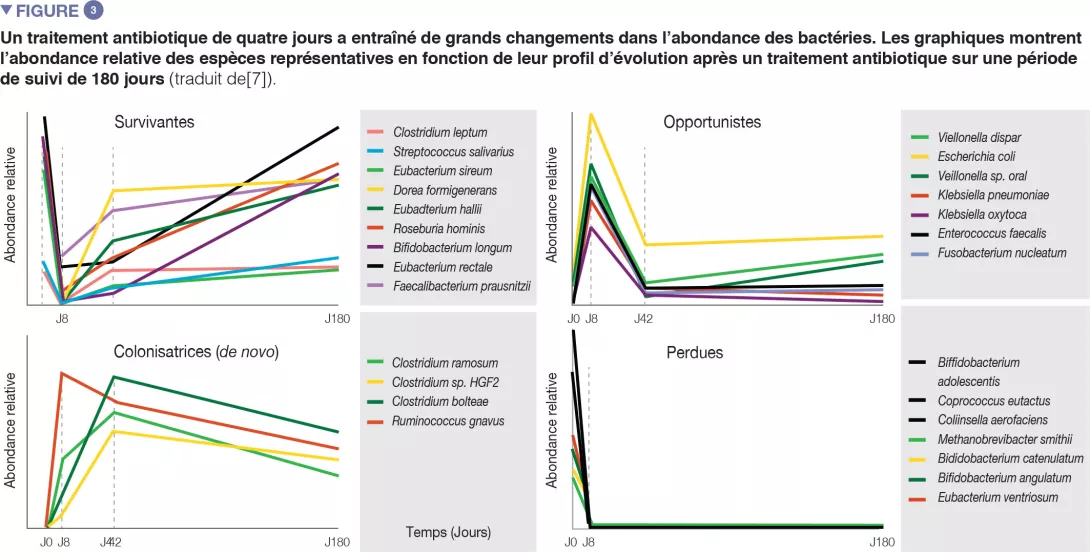
Chez des volontaires sains, une intervention antibiotique de 4 jours a entraîné la prolifération d’entérobactéries et d’autres pathobiontes, comme Enterococcus faecalis et Fusobacterium nucleatum, et la déplétion des bactéries du genre Bifidobacterium et des producteurs de butyrate [7]. Le microbiote intestinal des sujets étudiés est revenu à un état proche de l’état initial en 1,5 mois, bien que 9 espèces, qui étaient présentes chez tous les sujets avant le traitement, soient restées indétectables après 180 jours (Figures 2 et 3).
La dysbiose correspond à une altération de la composition et de la fonction du microbiote, qui entraîne des perturbations de l’écosystème microbien dépassant ses capacités de résistance et de résilience [8]. L’impact fonctionnel des antibiotiques sur les producteurs d’acides gras à chaîne courte, et en particulier de butyrate, pourrait avoir des conséquences à long terme en raison de la rupture de l’équilibre symbiotique entre le microbiote et l’hôte. L’incapacité à produire du butyrate augmente le flux d’oxygène en direction de la muqueuse et perturbe le micro-écosystème en favorisant la survie des bactéries résistantes à l’oxygène (entérobactéries) et en empêchant la récupération des producteurs de butyrate comme Faecalibacterium, qui sont des anaérobies stricts [9]. Ces modifications affectent gravement la capacité de résilience de l’écosystème et font évoluer le déséquilibre vers la chronicité.
Les effets indésirables des antibiotiques sur le microbiote intestinal et la perte d’attributs fonctionnels qui s’ensuit pourraient entraîner la rupture de l’équilibre symbiotique entre le microbiote et l’hôte.
Le résistome est l’ensemble des gènes bactériens qui contribuent directement ou indirectement à la résistance aux antibiotiques. Les gènes de résistance ne semblent pas avoir été sélectionnés en réponse à une exposition récente aux antibiotiques. Ces derniers, de même que la résistance, existent depuis des centaines de millions d’années, et le nombre de gènes composant le résistome reflète la co-évolution continue des micro-organismes producteurs d’antibiotiques et des micro-organismes cibles. La composition du résistome et la prévalence des gènes de résistance dans les bactéries associées à l’homme s’adaptent aux forces sélectives dérivées de l’action humaine.
Les espèces porteuses de gènes de résistance aux β-lactamines sont sélectionnées positivement pendant et après l’administration d’antibiotiques [7]. De même, le portage de gènes de résistance aux aminoglycosides augmente les probabilités de colonisation de novo. Le portage de gènes de résistance aux antibiotiques module le processus de récupération après l’administration d’antibiotiques [7].
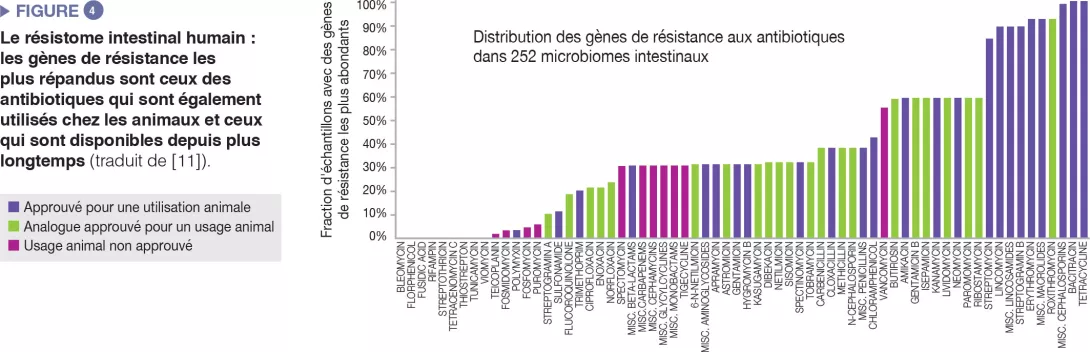
Le microbiome intestinal humain héberge un répertoire divers de gènes de résistance aux antibiotiques, que l’on peut analyser grâce à des technologies de séquençage moléculaire [10]. Une étude portant sur 252 échantillons fécaux humains provenant de différents pays a montré que les gènes de résistance les plus abondants dans le microbiome sont ceux correspondant aux antibiotiques également utilisés chez l’animal et à ceux disponibles depuis longtemps (Figure 4) [11]. Une correspondance a été établie entre les données sur le recours aux antibiotiques chez l’homme et l’animal, et les différences spécifiques à chaque pays ont été observées au niveau de la prévalence des gènes de résistance. Dans l’ensemble, ces données suggèrent l’existence d’une corrélation positive entre l’exposition aux antibiotiques et la prévalence des gènes d’antibiorésistance.
Certains gènes de résistance s’échangent facilement entre les bactéries par transfert horizontal. Des études ont montré qu’en cas de stress induit par les antibiotiques, des bactéries opportunistes disséminent les gènes de résistance au sein de la communauté microbienne. Une étude longitudinale du microbiome intestinal conduite chez des enfants finlandais a montré que le recours aux antibiotiques favorisait la dispersion des gènes d’antibiorésistance dans l’intestin en raison de la prolifération de bactéries porteuses de gènes de résistance et de la mobilisation accrue de ces gènes par les plasmides [12]. Les gènes de résistance aux antibiotiques présents sur les chromosomes microbiens ont montré un pic d’abondance après l’antibiothérapie, puis une forte baisse, alors que l’abondance des gènes de résistance présents sur les éléments mobiles persistait longtemps après la fin de l’antibiothérapie. Ce phénomène pourrait s’expliquer par le fait que les gènes épisomiques peuvent se diffuser largement entre différentes espèces par transfert horizontal.
En conditions de stress induit par les antibiotiques, des bactéries opportunistes disséminent les gènes de résistance au sein de la communauté microbienne intestinale. Le microbiote intestinal humain est un réservoir de gènes de résistance pour les micro-organismes pathogènes.
Le microbiote intestinal humain pourrait constituer le réservoir de gènes de résistance le plus accessible pour les microorganismes pathogènes. Un traitement antibiotique précoce est associé à une réduction de la diversité microbienne, mais également à un risque accru d’antibiorésistance.
Les probiotiques pourraient prévenir la prolifération d’espèces résistantes pendant l’antibiothérapie et minimiser l’expansion des gènes de résistance aux antibiotiques.
Des perturbations de l’écosystème microbien intestinal dans les premières années de vie et un terrain génétique favorable pourraient avoir un impact à long terme sur le système immunitaire, ce qui se traduit plus tard par des maladies ou une prédisposition aux maladies. Il a en effet été démontré que les maladies inflammatoires chroniques de l’intestin, les maladies métaboliques (diabète de type 2, obésité) et les maladies atopiques étaient associées à une altération de la composition du microbiote intestinal.
Le fait qu’une exposition répétée aux antibiotiques déclenche des altérations du microbiote intestinal ainsi qu’une inflammation est une hypothèse fréquemment avancée pour expliquer la pathogenèse des troubles inflammatoires. Une étude a montré que les nourrissons recevant des antibiotiques avant l’âge d’un an avaient 5,5 fois plus de risque de développer une MICI que les enfants non exposés [13]. De la même manière, une exposition aux antibiotiques au cours des deux premières années de vie, période de développement des populations adipocytaires de l’hôte, est associée à une obésité infantile [14]. La réduction de la richesse microbienne intestinale a été associée à une augmentation de l’adiposité, à une résistance à l’insuline et à la leptine, et à un phénotype inflammatoire plus prononcé.
Malgré la capacité de résilience du microbiote intestinal, la dispersion des gènes d’antibiorésistance constitue aujourd’hui une menace majeure pour la santé humaine, et l’utilisation excessive des antibiotiques semble en être la cause principale. En outre, de plus en plus d’éléments établissent un lien entre la fragilité du microbiote humain dans les pays industrialisés et l’expansion des maladies chroniques non transmissibles [3]. Là encore, l’exposition aux antibiotiques est une cause évidente. L’adoption de pratiques médicales plus durables constitue un défi majeur de santé publique au XXIe siècle. Une utilisation restreinte et rationnelle des antibiotiques représente incontestablement la meilleure manière de prévenir les déséquilibres néfastes du microbiote intestinal humain. Par ailleurs, l’utilisation d’un probiotique à l’efficacité prouvée pour la prévention des diarrhées induites par les antibiotiques a permis de limiter la prolifération d’espèces résistantes pendant l’antibiothérapie [15]. Cette stratégie pourrait également réduire la dispersion des gènes de résistance aux antibiotiques.
Depuis 2015, l'OMS organise chaque année la Semaine mondiale de sensibilisation à la résistance aux antimicrobiens (WAAW) dont l'objectif est de sensibiliser sur le phénomène mondial de la résistance aux antimicrobiens. Cette campagne, qui se tiendra du 18 au 24 novembre, encourage le grand public, les professionnels de santé et les décideurs à faire un bon usage des antimicrobiens afin d'éviter l'apparition de résistance.
 Les deux visages des Antibiotiques : Sauveurs de Vie et Perturbateurs de Microbiote
Les deux visages des Antibiotiques : Sauveurs de Vie et Perturbateurs de Microbiote
 Résistance aux antibiotiques : le microbiote au premier plan
Résistance aux antibiotiques : le microbiote au premier plan
Synthèse
Par le Pr. Gianluca Ianiro
Centre des maladies digestives, Fondazione Policlinico « A. Gemelli » IRCCS, Rome, Italie
Ces dernières années, l’infection à C. difficile (ICD) est devenue, au niveau international, un poids tout à la fois clinique et socioéconomique, du fait d’une augmentation de la morbidité, de la sévérité, de la mortalité et du risque de récidive de la maladie. Le microbiote intestinal joue un rôle considérable dans l’ICD, et ce pour de nombreuses raisons. Tout d’abord, la plupart des facteurs de risque de survenue d’une ICD, notamment le recours excessif aux antibiotiques à large spectre ou aux inhibiteurs de la pompe à protons, sont associés à un déséquilibre du microbiote intestinal. En outre, des modulateurs spécifiques du microbiote jouent un rôle dans la prévention (probiotiques spécifiques) ou le traitement (transplantation de microbiote fécal) de l’ICD. Dans cet article, nous allons examiner l’épidémiologie, les facteurs de risque et les traitements homologués de l’ICD, via une approche centrée sur le microbiote.
Clostridioides difficile (C. difficile, anciennement Clostridium difficile) est une bactérie anaérobie stricte à Gram positif sporulée. Les spores permettent à C. difficile de rester dans l’environnement et de se disséminer à partir des sujets infectés. Des conditions particulières (comme la dysbiose induite par les antibiotiques) favorisent la germination des spores dans le côlon, qui prennent alors une forme végétative dont le résultat est l’infection clinique (infection à Clostridium difficile [ICD]). Pendant la phase d’infection, C. difficile produit deux toxines : l’entérotoxine A et la cytotoxine B qui endommagent les colonocytes et déclenchent la réponse inflammatoire, ce qui conduit à différents tableaux cliniques allant d’une colite d’intensité légère à une colite pseudo-membraneuse et un mégacôlon toxique [1].
Ces dernières années, l’ICD est devenue un enjeu sanitaire et économique considérable dans la plupart des pays. Des études réalisées aux États-Unis rapportent une incidence de près de 453 000 cas et de près de 29 000 décès par ICD en 2011, et une incidence en Europe de 124 000 cas/an, avec près de 3 700 décès/ an. L’augmentation de la morbidité, de la durée d’hospitalisation et de la mortalité contribue à l’impact économique majeur de l’ICD, qui représentait près de 5 milliards de dollars aux États-Unis en 2011 et près de 3,7 milliards d’euros en Europe en 2013 [2, 3]. Ces chiffrent montrent que l’incidence des ICD a augmenté partout dans le monde, et ce pour différentes raisons. La première est le recours accru aux antibiotiques, qui sont un facteur de risque connu d’ICD. En outre, la diffusion de ribotypes spécifiques (principalement le ribotype virulent 027, mais également le 017 en Asie, le 018 en Italie, le 17 621 en Europe de l’Est et le 24 422 en Océanie) a permis le développement de cas groupés d’ICD. Le nombre de diagnostics a également augmenté du fait du développement de tests ultrasensibles (comme la PCR) et de la sensibilisation accrue auprès des professionnels de santé. La principale cause de l’augmentation globale de l’incidence des ICD semble être le taux accru de récidives. De 2001 à 2012, l’incidence annuelle des ICD récidivantes a augmenté de près de 189 %, alors que l’augmentation de l’incidence des ICD dans leur ensemble pendant la même période était de près de 43 % [2]. Dans la mesure où une infection récidivante a moins de chances d’être guérie par des antibiotiques qu’un premier épisode, elle est associée à une augmentation de la durée d’hospitalisation, de la morbidité et de la mortalité.
L’ICD est la première cause de diarrhée infectieuse associée aux soins de santé, mais des données récentes suggèrent une augmentation des cas communautaires. À ce jour, près de 25 à 35 % des cas d’ICD sont d’origine communautaire, probablement par le biais de différentes voies de transmission féco-orale (zoonose et alimentation par exemple).
Malgré cette augmentation des diagnostics d’ICD, un nombre considérable de patients atteints n’est toujours pas diagnostiqué, comme le montre l’étude EUCLID, ce qui augmente le risque de diffusion de la maladie.
Les ICD nosocomiales et communautaires semblent différer à plusieurs égards.
Tout d’abord, les patients atteints d’une infection nosocomiale ont davantage de risques de présenter un tableau clinique sévère, alors que ceux atteints d’une infection communautaire peuvent être des porteurs asymptomatiques, ce qui augmente le risque de diffusion de l’ICD.
D’autre part, on sait que l’ICD communautaire se diffuse également chez les patients sans facteur de risque standard.
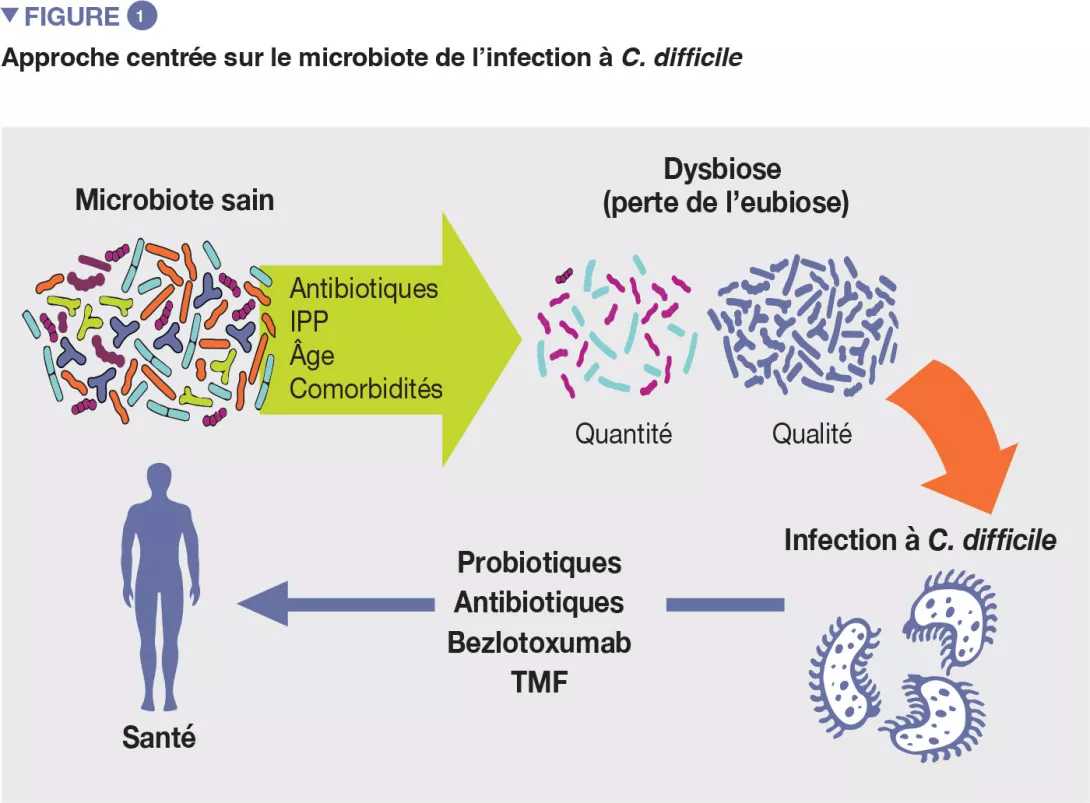
Bien que les mécanismes pathogènes exacts de l’ICD ne soient pas encore clairement établis, plusieurs facteurs de risque ont été identifiés [4]. Il est important de les connaître car le contrôle des facteurs de risque modifiables constitue une mesure de prévention contre les ICD. Les principaux facteurs de risque sont notamment l’âge, le recours aux antibiotiques et les inhibiteurs de la pompe à protons (Figure 1).
Si les antibiotiques restent aujourd’hui des molécules indispensables à l’arsenal thérapeutique, il convient également de prendre en compte leurs effets indésirables sur le microbiote intestinal ; un nombre important de données montre en effet une association entre leur utilisation et de nombreuses maladies associées à la dysbiose, notamment l’ICD [5].
Tout d’abord, les antibiotiques peuvent détruire les bactéries commensales capables d’exercer une action directe contre C. difficile (grâce à la sécrétion d’un certain nombre de bactériocines) et également entrer en compétition avec le pathogène pour les nutriments (comme l’acide sialique et le succinate). Les bactéries commensales jouent également un rôle protecteur indirect par la régulation des acides biliaires.
Clostridium scindens a récemment été associée à une résistance à la colonisation par C. difficile. Cette bactérie possède un opéron inductible par les acides biliaires qui est capable de coder pour des enzymes déshydroxylantes qui transforment les acides biliaires primaires en acides biliaires secondaires. Les acides biliaires primaires favorisent la germination des spores de C. difficile, alors que les acides biliaires secondaires ont la capacité d’inhiber ce processus [6].
Le recours aux antibiotiques systémiques est le facteur de risque modifiable le plus important des ICD. Le microbiote intestinal peut être un élément déterminant dans la réussite ou l’échec de la colonisation du côlon par C. difficile, via des voies directes et indirectes. En principe, le déséquilibre du microbiote intestinal induit par les antibiotiques à large spectre peut avoir plusieurs conséquences conduisant à une ICD.
En corollaire, les patients souffrant d’ICD récidivante ont un profil microbien déséquilibré, caractérisée par une abondance relative plus importante de familles bactériennes nocives telles que Enterobacteriaceae et Veillonellaceae, et une abondance relative plus faible de familles bénéfiques, notamment Ruminococcaceae, Bacteroidaceae et Lachnospiraceae.
Un certain nombre de revues systématiques, seules ou avec méta-analyse, ont évalué le rôle de différentes classes d’antibiotiques dans la survenue d’ICD. Dans la première méta-analyse (1998), le recours aux antibiotiques a été associé à une multiplication par 6 du risque de survenue d’une ICD, avec un risque maximal pour les fluoroquinolones, la clindamycine et les céphalosporines. De plus, le recours aux antibiotiques s’est avéré être un facteur prédictif indépendant de la récidive de l’ICD (risque relatif 1,76). L’un des facteurs-clés de la prévention des ICD est la promotion du bon usage des antibiotiques, et il est donc essentiel de connaître le risque d’ICD associé aux différentes classes d’antibiotiques (Tableau 1).
L’utilisation des antibiotiques suivants est associée à un doublement du risque d’ICD chez les patients hospitalisés : clindamycine, céphalosporines, carbapénèmes, fluoroquinolones, triméthoprime et sulfonamides. Dans la communauté, les antibiotiques sont associés à différents niveaux de risque de survenue ou de récidive de l’ICD, à savoir : clindamycine (risque multiplié par 8 à 20), céphalosporines et fluoroquinolones (multiplication par 3 à 5), macrolides (multiplication par 2 à 3) [5].
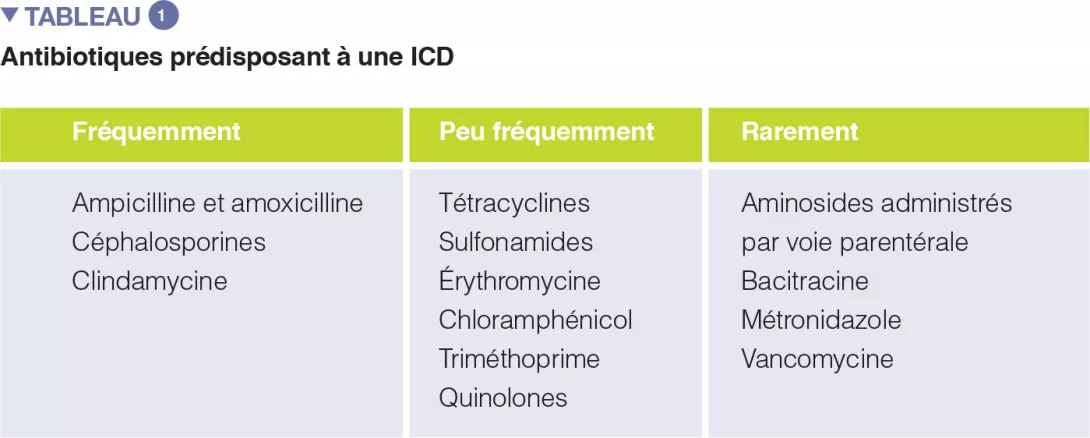
Les inhibiteurs de la pompe à protons (IPP) sont largement utilisés dans le monde entier pour le traitement de différents troubles gastro-intestinaux, notamment le reflux gastro-oesophagien, la hernie hiatale, la gastrite, l’infection à H. pylori (avec une antibiothérapie d’éradication) et l’ulcère gastroduodénal.
Dans l’ensemble, les IPP sont considérés comme des médicaments sûrs. Cependant, de nombreuses données montrent que le recours aux IPP est associé de façon significative à la survenue d’ICD.
En principe, les IPP peuvent majorer le risque de colonisation par C. difficile selon différents mécanismes, notamment la réduction de la production d’acide pouvant conduire à une pullulation bactérienne de l’intestin grêle et à une dysbiose et l’augmentation des sels biliaires, qui peut favoriser la germination des spores de C. difficile. Enfin, le fait qu’un pH gastrique plus élevé constitue un environnement plus sûr pour les spores n’est pas clairement établi [6].
Les preuves cliniques d’une association significative entre les IPP et l’ICD proviennent de plusieurs revues systématiques et méta- analyses, avec des odds ratios allant de 1,26 à 2,34, selon les études (de 3 à 67, en fonction des méta-analyses).
La plupart des données sont hétérogènes et proviennent de cohortes observationnelles, donc des facteurs de confusion tels que les autres médicaments utilisés, et les comorbidités pourraient altérer la qualité de cette observation. Cependant, l’association entre les IPP et l’ICD est restée significative même après stratification selon le recours aux antibiotiques, à la fois dans les études de cohorte et les études cas-témoins.
Le rôle délétère des IPP s’est avéré plus prononcé dans les ICD communautaires, ce qui suggère une utilisation excessive chronique dans la communauté plutôt qu’à l’hôpital.
Plus spécifiquement, les IPP ont été associés non seulement aux ICD dans leur ensemble, mais également aux ICD récidivantes, d’après plusieurs méta-analyses (comprenant de 3 à 16 études), avec des odds ratios allant de 1,52 à 2,51, même si les définitions de la récidive variaient significativement entre les études.
L’âge est l’un des facteurs de risque les plus connus de l’ICD primaire et récidivante.
Des preuves établies montrent que les taux d’ICD sont beaucoup plus élevés chez les adultes de plus de 65 ans que dans la population plus jeune. Dans une méta-analyse de 33 études, un âge supérieur à 65 ans a été identifié comme facteur prédictif indépendant de la récidive de l’ICD (risque relatif 1,63).
Toutefois, l’âge est un facteur de confusion important dans la mesure où l’utilisation de différents médicaments favorisant les ICD (antibiotiques, IPP) est plus fréquente chez les personnes âgées. De plus en plus d’éléments suggèrent que le microbiote des patients âgés est moins sain (du fait d’une réduction de la diversité microbienne et d’une augmentation des espèces opportunistes), ce qui confirme par ailleurs le rôle du déséquilibre du microbiote dans l’ICD [7].
L’association entre l’ICD et certaines comorbidités a été étudiée de manière systématique. Dans une revue systématique, un risque d’ICD significativement plus élevé a été observé pour les maladies inflammatoires chroniques de l’intestin (OR 3,72), l'insuffisance rénale (OR 2,64), les cancers hématologiques (OR 1,75) et le diabète (OR 1,15). Cette observation était particulièrement vraie pour les ICD communautaires [7].
Traditionnellement, le métronidazole et la vancomycine sont les options thérapeutiques les plus courantes dans l’ICD, les deux étant utilisées en première ligne ; mais seule la vancomycine a été recommandée selon un schéma à doses dégressives et intermittentes pour traiter les récidives [8].
Cependant ces dernières années, l’ICD est devenue plus difficile à traiter. Notamment, le métronidazole a montré des taux de guérison plus faibles que la vancomycine, de sorte que la vancomycine a été préférée au métronidazole pour les primo-infections également. Dans l’ensemble, la vancomycine perd également de son efficacité, et les taux de récidives augmentent. On assiste en outre à l’émergence de souches hypervirulentes de C. difficile, notamment le ribotype 027, qui répond moins bien aux antibiotiques standard et qui est associé à des tableaux cliniques plus sévères [8].
Ces dernières années, la fidaxomicine (un antibiotique à spectre étroit) s’est montrée supérieure à la vancomycine dans le traitement des récidives d’ICD. Cependant, son coût élevé et les récentes preuves de sa moindre efficacité par rapport à la transplantation de microbiote fécal (TMF) dans le traitement de l’ICD récidivante constituent des obstacles potentiels à son utilisation à grande échelle [9].
Les antibiotiques constituent une découverte scientifique extraordinaire qui permet de sauver des millions de vies, mais leur utilisation excessive et injustifiée suscite désormais de grandes inquiétudes pour la santé, notamment en raison de l'apparition de résistance aux antibiotiques et de dysbioses. Lisons la page consacrée à cette question.
Depuis 2015, l'OMS organise chaque année la Semaine mondiale de sensibilisation à la résistance aux antimicrobiens (WAAW) dont l'objectif est de sensibiliser sur le phénomène mondial de la résistance aux antimicrobiens. Cette campagne, qui se tiendra du 18 au 24 novembre, encourage le grand public, les professionnels de santé et les décideurs à faire un bon usage des antimicrobiens afin d'éviter l'apparition de résistance.
Les probiotiques sont généralement considérés comme une option fiable pour rétablir un microbiote intestinal sain après une dysbiose, induite par exemple par une antibiothérapie. Dans l’ensemble, certains probiotiques se sont révélés efficaces contre la diarrhée associée aux antibiotiques (DAA), qui est un effet indésirable fréquent de l’antibiothérapie [10-12]. Dans une méta-analyse de 21 essais randomisés, Saccharomyces boulardii a significativement réduit le risque de DAA (rapport de risques : 0,47) [11].
Dans la mesure où l’ICD peut être considérée comme un sous-groupe de DAA, l’efficacité des probiotiques dans la prévention des ICD a été évaluée. Une revue Cochrane a récemment montré, dans une méta-analyse de 23 essais, que les probiotiques sont à la fois sûrs et efficaces pour prévenir les ICD [13]. Cependant, seuls des probiotiques spécifiques, notamment Saccharomyces boulardii, Lactobacillus casei, un mélange de L. acidophilus et de Bifidobacterium bifidum, et un mélange de L. acidophilus, L. casei et L. rhamnosus, se sont révélés efficaces pour prévenir les ICD primaires après une antibiothérapie. En particulier, S. boulardii a montré son efficacité dans la prévention des ICD dans une cohorte de patients âgés hospitalisés, avec des économies potentielles à la clé. En effet, une étude canadienne a montré que l’utilisation de probiotiques préventifs avait permis d’économiser 518 $/patient par rapport à la prise en charge habituelle et de réduire le risque d’ICD [11]. Cependant, d’autres études à plus grande échelle sont nécessaires pour confirmer le rôle de probiotiques spécifiques dans la prévention des ICD.
Sur la base de ces résultats exceptionnels, les sociétés scientifiques ont inclus la TMF dans les options thérapeutiques pour les ICD récidivantes [14, 15]. On sait également que la TMF augmente la survie globale et réduit la durée d’hospitalisation chez les patients souffrant d’ICD récidivantes [16].
Bien que la TMF soit de plus en plus standardisée, elle reste encore sous-utilisée. Les approches qui garantiront à l’avenir une large diffusion de la TMF sont notamment la TMF par gélules et les médicaments à base de micro-organismes intestinaux.
La TMF consiste en l’introduction des selles d’un donneur sain dans le tube digestif d’un patient receveur afin de guérir les troubles liés à la dysbiose. À ce jour, plusieurs revues systématiques et méta-analyses ont montré que la TMF est extrêmement efficace pour guérir les ICD récidivantes (avec des taux de guérison pouvant atteindre 90 %).
L’ICD est une maladie lourde qui touche principalement les patients présentant plusieurs facteurs de risque, dont la plupart sont associés à un déséquilibre du microbiote intestinal, notamment le recours excessif aux antibiotiques, aux inhibiteurs de la pompe à protons et l’âge. Du point de vue microbiologique, le profil microbien des patients atteints d’ICD est caractérisé par une importante dysbiose du microbiote intestinal. Des modulateurs thérapeutiques du microbiote se sont révélés efficaces pour prévenir (probiotiques spécifiques, certaines souches de Lactobacillus et S. boulardii) ou guérir (TMF) les ICD récidivantes, ouvrant la voie à une approche basée sur le microbiote pour la prise en charge de cette maladie.
 La résistance aux antibiotiques est un inextricable enchevêtrement de problèmes
La résistance aux antibiotiques est un inextricable enchevêtrement de problèmes
 Résistance aux antibiotiques : le microbiote au premier plan
Résistance aux antibiotiques : le microbiote au premier plan
