El impacto de la gravedad en el síndrome del intestino irritable (SII)
Por la Dra. Maria Teresa Galiano
Servicio de Gastrología y Endoscopia, Servimed, Bogotá, Colombia

En un artículo reciente [1], se ha sugerido que el SII puede deberse a sistemas anatómicos, fisiológicos y neuropsicológicos ineficaces de gestión de la gravedad diseñados para optimizar la forma y la función gastrointestinales, proteger la integridad somática y visceral y maximizar la supervivencia en un mundo sometido a la gravedad.
¿Puede comentar esta hipótesis desde su punto de vista clínico?
Esta hipótesis es muy interesante. Creo que se debe tener en cuenta como una de las numerosas hipótesis que intenta explicar el síndrome del intestino irritable. Sin embargo, hay que demostrarla. Se deben realizar estudios para demostrar que alteraciones físicas debidas a cambios de gravedad afectan a la fisiología gastroenterológica. Creo que puede ser cierto que la gravedad afecta a la fisiología de un organismo y que normalmente estamos en equilibrio con esta fuerza permanente a la que estamos sometidos los seres humanos y todos los seres vivos de la Tierra. Las consecuencias que se producen cuando se altera este equilibrio pueden incluir el SII.
¿Está de acuerdo con la explicación del autor de que la gravedad puede provocar, como consecuencia, una alteración de la microbiota intestinal?
Estoy de acuerdo con el autor en que la gravedad puede alterar la microbiota intestinal y en que es posible que también altere su funcionamiento, incluida la fermentación. Creo que también es posible que altere el volumen de gas que actúa sobre la pared intestinal. Esto también debe comprobarse en los estudios correspondientes, pero estoy de acuerdo con la idea del autor de que la microbiota intestinal es susceptible a la gravedad.
¿Compartiría esta hipótesis con sus pacientes?
Compartiría esta hipótesis con los pacientes a los que creo que se podría aplicar en función de su fisiopatología y como una posible explicación de sus síntomas. De hecho, he observado cambios en mis pacientes cuando viajan a lugares que están al nivel del mar y regresan a Bogotá, donde vivo. Bogotá está a 2600 metros sobre el nivel del mar. Cuando estos pacientes regresan a Bogotá, presentan más síntomas debido a los cambios en la presión barométrica. Los cambios en la presión barométrica provocan cambios en la sensación, distensión y gases en los intestinos. En muchas ocasiones, les explico la sintomatología basándome en cambios en la fisiología que se producen según las distintas altitudes a las que se encuentran. Los cambios causados por las variaciones del equilibrio con la fuerza de la gravedad podrían servir para explicar los síntomas de estos pacientes.
Microbiota Vaginal #19
Por la Prof. Satu Pekkala
Becaria de investigación de la Academia de Finlandia, Facultad de Ciencias del Deporte y la Salud, Universidad de Jyväskylä, Finlandia
Cáncer de cuello uterino: el cerco se estrecha sobre Lactobacillus iners
El virus del papiloma humano de alto riesgo (VPH-AR) es la principal causa de cáncer de cuello uterino. La actividad sexual, el tabaquismo y la anticoncepción oral son algunos de los muchos factores que influyen en la infección inicial, en la resolución o en la aparición del VPH-AR, y su posible progresión a cáncer. En los últimos años, la microbiota vaginal se ha añadido a esta lista. Por ello, el valor de este estudio es evidente, ya que ha hecho un seguimiento de la microbiota cervicouterina de mujeres chinas infectadas con VPH-AR y que presentaban lesiones en su mayoría de grado leve, confirmadas histológicamente. Mediante un análisis del ARNr 16S de la microbiota de las 73 participantes en el estudio (con edades comprendidas entre los 24 y los 68 años) se observó que el VPH había desaparecido en 45 mujeres (61,6 %) al final del periodo de estudio de doce meses. La eliminación o no del virus no se debía a diferencias en la edad de las pacientes, a la fase de la enfermedad, al subtipo de VPH, al tipo de comunidad bacteriana vaginal o la diversidad de la microbiota vaginal. Pero sí que parecía que había ciertas especies bacterianas implicadas: las mujeres con una baja población de enterococo ASV_62 y con una población rica de Lactobacillus iners al inicio del estudio tenían menos probabilidades de haber eliminado el VPHHR al cabo de doce meses. La única excepción fueron 22 mujeres sometidas a tratamiento quirúrgico (conización) por lesiones graves, quizá porque el impacto inmediato de la resección de la lesión sobre la eliminación del VPH ocultó el impacto de la flora. Previamente, se había informado de una posible relación entre L. iners y el VPHAR, en un metaanálisis que sugería un riesgo dos e incluso tres veces mayor de VPH-AR persistente cuando la microbiota vaginal está dominada por L. iners. Parece ser que esta bacteria es flexible y adaptable, y domina la microbiota vaginal de ciertas mujeres durante la menstruación o los episodios de vaginosis bacteriana. Por otro lado, una microbiota vaginal dominada por L. iners (CST III) se suele considerar como uno de los tipos más comunes de comunidad bacteriana vaginal entre mujeres asiáticas en edad fértil. Por lo tanto, la bibliografía actual no deja claro todavía si esta cepa concreta de Lactobacillus debe considerarse beneficiosa, patógena o ambas cosas. Además, hay que seguir trabajando para aclarar los mecanismos por los que L. iners favorece la infección persistente por VPH o la progresión de las lesiones, sobre todo teniendo en cuenta que en el estudio actual participó un número reducido de pacientes.
Microbiota Intestinal #19
Por la Prof. Satu Pekkala
Becaria de investigación de la Academia de Finlandia, Facultad de Ciencias del Deporte y la Salud, Universidad de Jyväskylä, Finlandia

Metabolismo microbiano del 5-ASA y síndrome del intestino irritable
La enfermedad del intestino irritable (EII) se trata con ácido 5-aminosalicílico (5-ASA). Sin embargo, más de la mitad de los pacientes no responden favorablemente al tratamiento. Estudios anteriores sugieren que esto se debe, en parte, a que el 5-ASA puede ser metabolizado por las bacterias intestinales en ácido N-acetil-5-aminosalicílico, que no es eficaz clínicamente. En este sofisticado estudio, Mehta et al. se propusieron identificar las enzimas microbianas intestinales que generan el ácido N-acetil-5-aminosalicílico. Las muestras de heces humanas se analizaron mediante multiómica. Los análisis metabolómicos no dirigidos de las muestras antes y después de la administración de 5-ASA revelaron posibles mediadores microbianos de los efectos antiinflamatorios del 5-ASA. Entre ellos, se encontraba una disminución del 2-aminoadipato, un metabolito bacteriano que se ha relacionado con un mayor estrés oxidativo. Además, parecía que el 5-ASA alteraba el metabolismo del nicotinato, lo que posiblemente permitiría explicar algunos efectos antiinflamatorios. Además, los autores trataron de identificar las enzimas microbianas que metabolizan potencialmente el 5-ASA. Gracias a la combinación de la metatranscriptómica y de la metabolómica, identificaron tres Acetil-CoA C-cetiltransferasas (AcylCoA NAT) que se asociaban con los niveles de ácido N-acetil-5-aminosalicílico en las personas que habían recibido 5-ASA. Además, se identificaron algunas tiolasas como posibles enzimas candidatas. A continuación, las enzimas candidatas se expresaron heterólogamente en Escherichia coli, y se midió su actividad bioquímica. Firmicutes CAG:176 tiolasa y Faecalibacterium prausnitzii acyl-CoA NAT fueron capaces de acetilar 5-ASA utilizando acetil-CoA. Por último, un análisis metagenómico de muestras de heces reveló que las acetiltransferasas inactivadoras de 5-ASA de la microbiota intestinal se asociaban a un mayor riesgo de fracaso del tratamiento en pacientes que habían recibido 5-ASA. En conjunto, los hallazgos de este estudio pueden ayudar a avanzar en la posibilidad de un tratamiento personalizado de la EII basado en el microbioma.

Helicobacter pylori y cáncer colorrectal
La infección por Helicobacter pylori puede provocar cáncer gástrico y aumentar el riesgo de cáncer colorrectal (CCR). Sin embargo, faltan datos farmacodinámicos en este ámbito. El estudio de Ralser y colaboradores identifica, en un modelo de roedores, los mecanismos subyacentes de cómo la infección por H. pylori contribuye al CCR. Cuando los autores infectaron con H. pylori a ratones APC, que son excelentes modelos animales portadores de múltiples neoplasias intestinales, se observó un aumento de la carga tumoral en el intestino delgado y el colon. Se sabe que la respuesta inmunitaria de los linfocitos T del anfitrión contribuye a la carcinogénesis gástrica, lo que llevó a los autores a estudiar estas respuestas en el intestino. Descubrieron una reducción de los linfocitos T reguladores y de los linfocitos T proinflamatorios, así como un aumento de la IL-17A, que se ha demostrado que es uno de los principales agentes de la respuesta inmunitaria frente a H. pylori. Los ratones infectados se caracterizaban por una mayor abundancia de microbios intestinales conocidos por ser proinflamatorios y de bacterias que degradan la mucosidad, como Akkermansia. Gracias al estudio de los perfiles transcriptonómicos de las células epiteliales intestinales, descubrieron que H. pylori causaba la activación de las vías NF-κB y STAT3. La activación de estas vías también se ha observado anteriormente en pacientes con CCR. Curiosamente, en los ratones axénicos infectados con H. pylori apenas se observó activación de la señalización de STAT3, lo que sugiere que la carcinogénesis inducida por H. pylori en el intestino delgado depende, en parte, del microbioma intestinal. Por último, los autores han demostrado que la carcinogénesis colorrectal inducida por H. pylori se puede prevenir mediante la erradicación de la bacteria con antibióticos. Los autores llegan a la conclusión de que se debe tener en cuenta el estado de H. pylori en la implementación de medidas de prevención del CCR.
Diversidad vírica en el intestino del lactante sano
En la primera infancia, el microbioma intestinal contribuye a la maduración del sistema inmunitario para proteger frente a las enfermedades crónicas en las etapas posteriores de la vida. Aunque se sabe que los bacteriófagos (es decir, virus que infectan a bacterias) pueden controlar el crecimiento de las poblaciones bacterianas, los viromas intestinales no se han estudiado en profundidad. Mediante la secuenciación del metagenoma, este estudio analiza 647 viromas de una cohorte danesa de lactantes de un año. El primer hallazgo sorprendente fue que las unidades taxonómicas operativas virales del intestino del lactante estaban ausentes en gran medida de las bases de datos de virus intestinales. Esto sugiere que el intestino del lactante alberga virus especializados diferentes de los del intestino del adulto. Los subtipos víricos más predominantes de los lactantes no se habían descrito en gran medida. Sin embargo, los anelovirus (Anelloviridae) de ADN monocatenario que infectan a vertebrados y los microvirus (Petitvirales) bacterianos de ADN monocatenario se encontraban entre los más abundantes. Además, las familias virulentas de caudovirus Skunaviridae, Salasmaviridae, β-crassviridae y Flandersviridae también se encontraban bien representadas en los viromas de los lactantes. En general, los virus temperados eran menos prevalentes que los virulentos, a pesar de encontrarse en un mayor número de niños. La abundancia a escala de familia no se relacionó significativamente con el estilo de vida de los fagos, determinado por la integrasa como indicador de un estilo de vida temperado. Sin embargo, las familias de caudovirus temperados eran genéticamente más diversas que las familias virulentas. El análisis de predicción de los anfitriones bacterianos de los viromas mostró que Bacteroides, Faecalibacterium y Bifidobacterium eran los tres géneros de anfitriones más predominantes en el intestino de los lactantes. Entre ellos, las familias que infectaron a Bacteroides eran más a menudo virulentas y específicas del anfitrión. Aunque no se ha llegado a conclusiones claras, el estudio enriquece los conocimientos sobre la taxonomía de los fagos y contribuye al desarrollo de investigaciones futuras sobre el viroma intestinal del lactante.

55º encuentro anual de la Espghan
Por la Dra. Tania Mahler
Dolor crónico y trastornos digestivos funcionales, Médica adjunta en el Hospital Universitario Infantil Reina Fabiola, Bélgica

En el encuentro de la Espghan (Sociedad Europea de Gastroenterología, Hepatología y Nutrición Pediátrica) de 2023 celebrado en Viena, se reunieron 4300 participantes de todo el mundo. Tras el complicado período de la pandemia de Covid-19, este evento ha supuesto una bocanada de aire fresco, ya que los participantes pudieron asistir a presentaciones en directo y tener conversaciones cara a cara. Estas interacciones en persona han resultado mucho más agradables y enriquecedoras que las alternativas virtuales. Varios grupos de investigación centraron su trabajo en el microbioma intestinal y presentaron datos relevantes en el campo de la pediatría.
El impacto del microbioma en la salud y en la enfermedad está ampliamente reconocido, por lo que es lógico que tanto pediatras como investigadores en pediatría intenten entender mejor cómo podemos manipular el microbioma y cómo podemos detectar una enfermedad en su fase temprana utilizando la firma del microbioma. El objetivo de este resumen es arrojar luz sobre varios temas clave que se trataron profusamente.
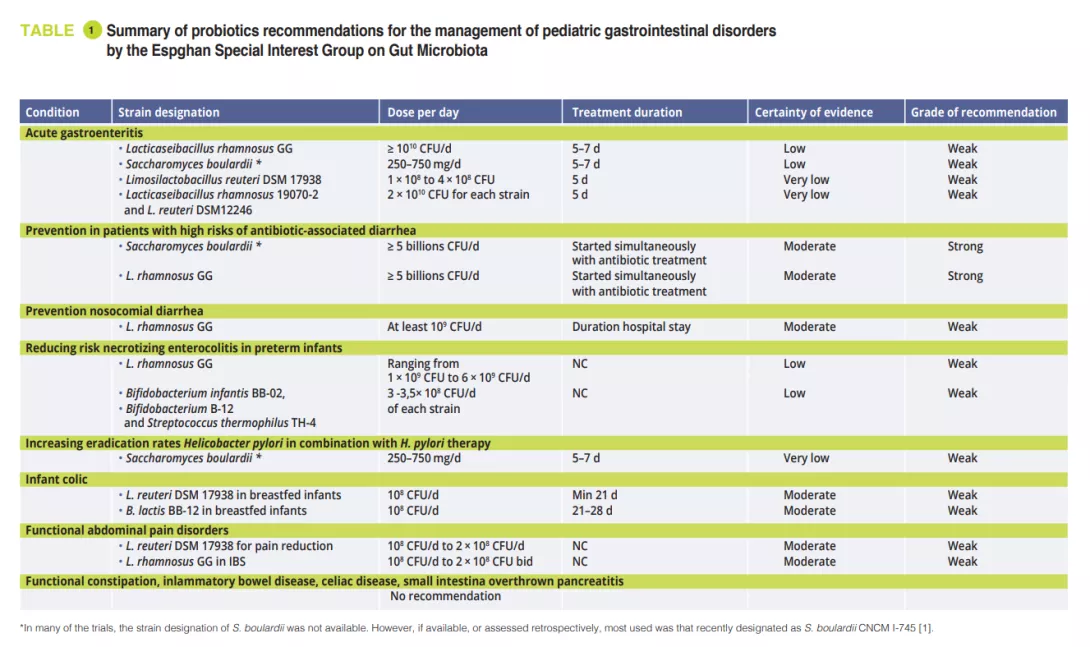
Recomendaciones para el uso de probióticos en trastornos digestivos pediátricos seleccionados
En febrero de 2023, el grupo de interés especial de la Espghan sobre la microbiota intestinal publicó unas recomendaciones para el uso de probióticos en el abordaje de trastornos digestivos pediátricos seleccionados, basadas en revisiones sistemáticas y/o metanálisis mediante el proceso Delphi modificado [1]. En la reunión del grupo de interés especial sobre microbiota intestinal y modificaciones, la profesora Szajewska nos mostró los resultados de este trabajo. Solo se ha demostrado la utilidad de algunas cepas probióticas específicas en determinadas condiciones. En el artículo original [1] se puede encontrar una descripción clara de las recomendaciones precisas.
TMF en adolescentes con SII resistente al tratamiento
La Dra. De Bruijn, del grupo de investigación de la Amsterdam UMC presentó, en la sesión plenaria sobre el resumen mejor puntuado, su estudio sobre la eficacia del TMF en adolescentes con SII resistente al tratamiento en un ensayo aleatorizado controlado con placebo y con doble ciego [2]. Su charla fue fascinante, pero también provocó muchas reacciones entre el público cuando proyectó una diapositiva de un paciente que recibía una inyección de materia fecal. Hasta donde sabemos, solo otro estudio pediátrico había evaluado el alivio de la distensión abdominal mediante TMF, otro signo de los trastornos en la interacción intestino-cerebro y que suele presentarse en el SII [3].
El dolor crónico en el SII puede tener un impacto enorme en la actividad de niños y adultos, y desembocar en absentismo escolar y laboral y en una mala calidad de vida. El origen de la enfermedad es multifactorial y se puede explicar mejor mediante el modelo biopsicosocial. Uno de los factores clave es la disbiosis de la microbiota intestinal. En adultos se han publicado diferentes estudios sobre el efecto positivo observado con el TMF [4].
En pediatría, tratamientos no farmacológicos como la información, la hipnosis y el mindfulness son más eficaces que los tratamientos farmacológicos [5]. Aun así, los síntomas se mantienen en aproximadamente el 25 % de los pacientes. Se han probado prebióticos, probióticos y simbióticos con distintos resultados para corregir la disbiosis en el SII. El cambio a una dieta FODMAP también puede influir en la flora gastrointestinal [5]. Pero en grupos específicos de pacientes, el TMF - siempre que sea seguro - podría ser el tratamiento definitivo para restablecer de manera eficaz un microbioma gastrointestinal sano. En el estudio de De Bruijn et al., se seleccionaron y aleatorizaron 32 pacientes con SII resistente al tratamiento de entre 16 y 21 años. Un grupo recibió instilaciones fecales alogénicas (donante sano) y el otro grupo autólogas (propias) mediante sonda nasogástrica al inicio y 6 semanas después. La eficacia clínica se definió como la proporción de pacientes con una reducción de más de 50 puntos en el Sistema de Puntuación de la Gravedad del SII (SPG-SII). Se evaluó a los pacientes 12 semanas y 6 meses después del TMF. Ambos grupos presentaron una puntuación similar en el SPG-SII al inicio. Después de la primera evaluación no hubo diferencias estadísticas, pero a los 6 meses del seguimiento hubo mejoría en el 60 % de los pacientes que recibieron un TMF alogénico frente al 25 % del grupo autólogo (p = 0,048). La calidad de vida relacionada con la salud (CDV) era uno de los beneficios secundarios. No hubo diferencias en la puntuación total de CDV entre ambos grupos al inicio, pero mejoró de manera significativa tras el TMF alogénico. No se registraron acontecimientos adversos. El TMF alogénico parece ser una manera interesante de tratar el SII resistente al tratamiento entre los jóvenes, pero es necesario hacer más estudios.
Microbiota y enfermedades inflamatorias intestinales
En la sesión de gastroenterología sobre la enfermedad del intestino irritable (EII), un grupo checo presentó un estudio cuyo objetivo era evaluar si los cambios en la microbiota en la enfermedad de Crohn activa (EC) se debían al tratamiento anti-TNFα o si eran el resultado de una menor actividad inflamatoria de la mucosa [6]. Para ello, compararon a niños bajo tratamiento anti-TNFα con EC o con artritis idiopática juvenil (AIJ). Los resultados mostraron que la cicatrización de la mucosa en la EC era fundamental para conseguir cambios en el bacterioma. El tratamiento anti-TNFα en la AIJ no tuvo ninguna repercusión en el bacterioma de este grupo de pacientes. Schwerd et al. hicieron un seguimiento de 20 pacientes pediátricos con EC recién diagnosticada que recibieron primero un tratamiento con nutrición enteral exclusiva con (NEE) con muestras de heces [7]. La enfermedad remitió en quince de veinte pacientes. Se observaron cambios claros temporales e individuales en la microbiota intestinal y los metabolitos, con una menor abundancia de Lachnospiraceae y un incremento de los ácidos grasos insaturados de cadena larga. La fermentación ex vivo con medios similares a la NEE y su posterior transferencia en modelos murinos gnotobióticos mostró un efecto protector en contraste con los medios ricos en fibra y con los colonizados directamente con la microbiota inicial del paciente. Basándose en esos resultados, llegaron a la conclusión de que los microbiomas de los pacientes modulados con NEE regulan la inflamación intestinal. Además, explicaron detalladamente la posibilidad de recurrir a una dieta baja en fibra para la remisión a largo plazo. Un estudio multicéntrico realizado en el Reino Unido (en niños y adultos) estudió la posibilidad de utilizar una dieta de alimentos sólidos para tratar la enfermedad de Crohn (CD-TREAT) con la finalidad de elaborar una dieta más sabrosa que pudiera influir en la inflamación intestinal modificando las bacterias intestinales [8]. La dieta se personalizó para cada paciente, pero excluyó componentes alimenticios concretos, como el gluten, la lactosa o el alcohol. El 55 % de los pacientes que siguieron este régimen presentaron una calprotectina fecal significativamente inferior, además de cambios microbianos y metabólicos en la misma línea que los pacientes que siguieron una NEE satisfactoria. Esto no se observó en los pacientes que no respetaron la dieta. A partir de estos hallazgos, podría ser interesante utilizar heces autólogas de pacientes tratados satisfactoriamente con NEE para el TMF. El grupo de Schwerd analizó esta posibilidad mediante cápsulas de TMF autólogo. Llegaron a la conclusión de que este planteamiento no era adecuado, ya que seguía habiendo una carga de patógenos demasiado elevada y una diversidad de la microbiota demasiado baja [9].
El grupo de Colonia presentó un poster muy interesante sobre el seguimiento de 2 casos de EII muy temprana resistente a los esteroides y al tratamiento antiTNFα. El primer paciente tiene colitis ulcerosa y actualmente lleva 3 años en remisión total con la administración semanal de un edema con una preparación de heces de donante. El segundo paciente, con EC, se encuentra solo en remisión parcial después de un año de seguimiento [10].
Este resumen ha dado un breve repaso de una selección de charlas, comunicaciones y pósteres de la Espghan. En el Journal of Pediatric Gastroenterology se recogen muchos otros datos interesantes en forma de resúmenes. Los esfuerzos conjuntos de los investigadores y de los médicos seguirán desentrañando el misterio de la microbiota intestinal, para acabar aportando nuevas maneras de tratar y prevenir enfermedades.
1. Szajewska H, Berni Canani R, Domellöf M, et al. Probiotics for the Management of Pediatric Gastrointestinal Disorders: Position Paper of the ESPGHAN Special Interest Group on Gut Microbiota and Modifications. J Pediatr Gastroenterol Nutr 2023; 76: 232-47
2. De Bruijn C, Zeevenhoven J, Vlieger A, et al. Efficacy of fecal microbiota transplantation in adolescents with refractory irritable bowel: a randomized, double-blind, placebo-controlled trial. J Pediatr Gastroenterol Nutr 2023; 76(S1 Suppl 1): 1-1407
3. Wang YZ, Xiao FF, Xiao YM, et al. Fecal microbiota transplantation relieves abdominal bloating in children with functional gastrointestinal disorders via modulating the gut microbiome and metabolome. J Dig Dis 2022; 23: 482-92
4. El-Salhy M, Winkel R, Casen C, et al. Efficacy of Fecal Microbiota Transplantation for Patients with Irritable Bowel Syndrome at 3 Years After Transplantation. Gastroenterology 2022; 163: 982-94.e14
5. Mahler T, Hoffman I, Smets F, et al. The Belgian consensus on irritable bowel syndrome: the paediatric gastroenterologist view. Acta Gastroenterol Belg 2022; 85: 384-6
6. Hurych J, Mascellani Bergo A, Lerchova T, et al. The faecal microbiome and metabolome changes in Crohn’s disease are associated with decreased mucosal inflammatory activity. J Pediatr Gastroenterol Nutr 2023; 76 (S1 Suppl 1): 1-1407
7. Schwerd S, Häcker D, Siebert K, et al. Exclusive enteral nutrition initiates protective functions in the gut microbiota and metabolome to induce remission in pediatric Crohn’s disease. J Pediatr Gastroenterol Nutr 2023; 76 (S1 Suppl 1): 1-1407
8. Macdonald J, Wilson D, Henderson P, Din S, e Chantges in faecal microbiome and metabolome are more pronounced in Crohn’s disease patients who adhered to the CD-TREAT diet and responded by calprotectin. J Pediatr Gastroenterol Nutr 2023; 76 (S1 Suppl 1): 1-1407
9. Hölz H, Heetmeyer J, Tsakmaklis A, et al . Autologous fecal microbiota transfer in pediatric Crohn ́s disease patients under treatment with exclusive enteral nutrition harbors major challenges - a feasibility test. J Pediatr Gastroenterol Nutr 2023; 76 (S1 Suppl 1): 1-1407
10. Fritz T, Huenseler C, Broekaert I. Safety and efficacy of long-term faecal microbiota transfer in very early onset inflammatory bowel disease. J Pediatr Gastroenterol Nutr 2023; 76 (S1 Suppl 1): 1-1407
Microbiota: fundamental para la salud, pero con una escasa concienciación global
¿Qué saben hoy nuestros conciudadanos sobre el papel de la microbiota? ¿Qué saben sobre cómo cuidar a su microbiota? ¿Qué papel desempeñan los profesionales sanitarios a la hora de informar a los pacientes? Para responder a estas preguntas, el Biocodex Microbiota Institute encargó a Ipsos una gran encuesta internacional sobre el tema: el Observatorio Internacional de la Microbiota. Para realizar esta encuesta, Ipsos interrogó a 6500 personas de 7 países. La encuesta se realizó del 21 de marzo al 7 de abril de 2023. Los resultados se presentaron el 27 de junio, en el Día Mundial del Microbioma.
Muy poco conocimiento de la palabra ”microbiota”
En general, el conocimiento de la microbiota es bastante bajo: solo 1 de cada 5 personas afirma que conoce el significado exacto del término microbiota (21 %), mientras que el resto admite que conoce el término solo de oídas (43 %). Más de 1 de cada 3 personas afirman que ni siquiera han oído la palabra (36 %). Además, al profundizar en su nivel de conocimiento, las nociones son superficiales. Mientras que una pequeña minoría afirma que sabe lo que es la microbiota intestinal (el 53 %, pero solo el 24 % sabe exactamente en qué consiste), muchos otros tipos de microbiota son menos conocidos: ya sea la microbiota vaginal (el 45 % de los entrevistados conoce el término, pero solo el 18 % sabe exactamente qué es), la microbiota bucal (el 43 % lo conoce de nombre, pero solo el 17 % sabe en qué consiste) o la microbiota cutánea (el 40 % conoce el término, pero solo el 15 % sabe en qué consiste). Otros términos son incluso menos conocidos, como la microbiota urinaria (solo el 14 % sabe exactamente en qué consiste), la microbiota pulmonar (el 13 % sabe exactamente en qué consiste), y la microbiota ORL (el 11 % sabe exactamente en qué consiste).
Y un conocimiento relativamente escaso sobre la función y la importancia de la microbiota
Alrededor de 3 de cada 4 entrevistados son conscientes de que un riesgo de desequilibrio en la microbiota puede tener consecuencias graves para la salud (75 %), de que nuestra alimentación tiene consecuencias importantes para el equilibrio de nuestra microbiota (74 %) y de que nuestra microbiota desempeña una función real en los mecanismos de defensa inmunitaria (72 %). Por lo demás, los conocimientos siguen siendo muy moderados. Más de 1 de cada 3 personas desconoce que los antibióticos afectan a nuestra microbiota (34 %). Casi 1 de cada 2 personas desconoce que la microbiota está conformada por bacterias, hongos y virus (46 %), y que permite al intestino transmitir información fundamental sobre la salud al cerebro (47 %). 1 de cada 2 personas cree que cuando la microbiota está desequilibrada o no funciona bien, no hay mucho que se pueda hacer al respecto (47%). Por último, la gran mayoría de las personas entrevistadas desconocía que muchas enfermedades como el Parkinson, el Alzheimer y el autismo pueden estar relacionadas con la microbiota (75 %).
1 de cada 5 personas afirma que conoce el significado exacto del término microbiota (21 %)
24% sabe exactamente en qué consiste
1 de cada 3 personas fue informado que la toma de antibióticos podría tener consecuencias negativas sobre el equilibrio de su microbiota
¿El principio de la toma de conciencia?
En la actualidad, más de 1 de cada 2 personas afirman haber adoptado comportamientos en sus vidas diarias para mantener el equilibrio de la microbiota (57 %). Hay que aplaudir esta toma de conciencia, pero también hay que ponerla en perspectiva. En primer lugar, porque solo 1 de cada 7 personas afirma que lo hace “mucho” (15 %), mientras que la mayoría del resto de personas afirma que lo hace solo “un poco” (42 %). En segundo lugar, el 43 % de las personas encuestadas afirma no haber adoptado ningún comportamiento específico. Los resultados del Observatorio Internacional de la Microbiota demuestran que aún queda mucho por hacer en este ámbito.
Información proporcionada por profesionales sanitarios: ¡un vector de información revolucionario!
Menos de 1 de cada 2 pacientes dice que su médico le ha explicado alguna vez cómo mantener una microbiota equilibrada (44 %, pero solo se lo han explicado más de una vez al 19 %), o le ha recetado probióticos o prebióticos (46 %, pero solo el 21 % afirma haberlo hecho varias veces). Solo una minoría de los entrevistados afirma que su médico le ha informado sobre la importancia de tener una microbiota equilibrada (42 %). Por último, solo 1 de cada 3 dice que su médico le ha enseñado alguna vez qué es la microbiota y para qué sirve (37 %, y solo al 15 % se lo habían explicado varias veces).
La información que proporcionan los médicos al recetar antibióticos ilustra hasta qué punto sigue siendo inadecuada la concienciación de los pacientes sobre los riesgos del tratamiento en términos de desequilibrio de la microbiota. Al prescribir antibióticos se debería de proporcionar información fundamental sobre la microbiota, pero en muchos casos no es así. Cuando se usan antibióticos se pone en peligro la microbiota del paciente. Cuando se recetan antibióticos, menos de 1 de cada 2 pacientes afirma que su médico le ha informado sobre el riesgo de problemas digestivos relacionados con los antibióticos (41 %). Solo 1 de cada 3 recibió consejos sobre cómo minimizar las consecuencias negativas de la toma de antibióticos para la microbiota (34 %) o se le informó de que tomar antibióticos podía tener consecuencias negativas para el equilibrio de la microbiota (33 %).
Lo que demuestra la encuesta es que, una vez que el paciente ha recibido toda la información sobre el tema y de forma repetitiva, su relación con la microbiota cambia significativamente, y se diferencia de la media. Más de 9 de cada 10 personas (95%) a las que su médico les ha informado en repetidas ocasiones, han adoptado comportamientos para mantener la microbiota equilibrada, frente al 57 % de todos los encuestados. Esto demuestra que la información repetida por parte de un profesional sanitario tiene una repercusión muy importante en los niveles de conocimiento y en los comportamientos.
Esta encuesta exclusiva revela, por un lado, una falta general de conocimiento sobre cómo afecta la microbiota a nuestra salud y, por otro, la importancia del papel que desempeñan los profesionales sanitarios a la hora de divulgar conocimientos.
1 de cada 2 pacientes dice que su médico le ha explicado alguna vez cómo mantener una microbiota equilibrada
95% a los que su médico les ha informado en repetidas ocasiones, han adoptado comportamientos para mantener la microbiota equilibrada
vs 57% de todos los encuestados
La colestasis altera el desarrollo de la microbiota intestinal y la actividad de la hidrolasa de los ácidos biliares en recién nacidos prematuros
ARTÍCULO COMENTADO - Niño
Por el Prof. Emmanuel Mas
Gastroenterología y nutrición, Hospital de Niños, Toulouse, Francia

Comentario sobre el artículo original de Lynch LE et al. (Gut Microbes) [1]
La colestasis es un trastorno del flujo de la bilis del hígado al intestino. En neonatos, la colestasis provoca una deficiencia en el crecimiento y puede evolucionar a una insuficiencia hepática y la muerte. El flujo biliar normal necesita un eje hígado-intestino-microbiota intacto, en el que los ácidos biliares primarios derivados del hígado se transforman en ácidos biliares secundarios. Las enzimas microbianas (hidrolasa de los ácidos biliares [BSH]) son responsables de la primera etapa, que consiste en desconjugar los ácidos biliares primarios conjugados con glicina y taurina. En los neonatos, la colestasis suele tratarse con ácido ursodesoxicólico (UDCA), un potente ácido biliar colerético, a pesar de que no se comprenden bien las interacciones entre el UDCA, las bacterias intestinales y los otros ácidos biliares. Este estudio, basado en 124 muestras de heces procedentes de 24 recién nacidos, revela nuevas asociaciones que relacionan los ácidos biliares isoméricos y la actividad de la BSH con las trayectorias del crecimiento neonatal. Estos datos ponen de relieve el hecho de que la desconjugación de los ácidos biliares es una función microbiana fundamental, adquirida al principio del desarrollo neonatal y alterada por la colestasis.
¿Qué se sabe ya sobre este tema?
Los neonatos prematuros, nacidos antes de las 37 semanas de amenorrea (SA), presentan un mayor riesgo de padecer colestasis. La colestasis, que se define por una disminución del flujo biliar, se ve favorecida por distintos factores de riesgo como la prematuridad, un bajo peso al nacer y la nutrición parenteral.
A falta de una causa, se habla de colestasis neonatal transitoria. Para mejorar la colestasis, se suele iniciar un tratamiento con ácido ursodesoxicólico (UDCA).
Los ácidos biliares son necesarios para la absorción de lípidos y vitaminas liposolubles. Durante una colestasis, se produce una disminución de la cantidad de ácidos biliares en el intestino, pero también una modificación de las proporciones de los distintos ácidos biliares.
Los ácidos biliares primarios se producen a partir del colesterol y se conjugan en el hígado. Sus interacciones con la microbiota intestinal son importantes, y dan lugar a la formación de ácidos biliares secundarios, mediante la interacción de una enzima microbiana, la hidrolasa de ácidos biliares (BSH).
Los ácidos biliares y la microbiota intestinal repercuten en el crecimiento y el desarrollo de los bebés prematuros. Los autores han querido estudiar el impacto de la colestasis en el desarrollo de la microbiota intestinal en bebés extremadamente prematuros y en la desconjugación de los ácidos biliares.
¿Cuáles son los principales resultados aportados por este estudio?
Los autores incluyeron a 24 recién nacidos prematuros, 12 con colestasis y 12 testigos, nacidos en la 27,2 ± 1,8 SA, con un peso medio al nacer de 946 ± 249,6 g. El pico medio de bilirrubina conjugada fue de 7,0 mg/dl. No había diferencias entre los dos grupos en cuanto al entorno intrauterino, el tipo de parto y el uso de antibióticos a lo largo del tiempo.
Se recogieron muestras de heces desde el nacimiento hasta las seis semanas de vida. La secuenciación de las muestras (secuenciación indiscriminada o shotgun) mostró que, en los testigos, la diversidad alfa aumentaba durante el primer mes de vida. En cuanto a los filos, Proteobacteria y Firmicutes fueron los más abundantes; en cuanto a los géneros, Staphylococcus fue el más abundante al nacer para luego disminuir, mientras que la abundancia de Klebsiella aumentaba progresivamente (figura 1). Clostridium perfringens fue la especie cuya abundancia relativa aumentaba más con el paso del tiempo, que se definió por la edad postmenstrual (PMA, suma de la edad al término del parto (SA) y de la edad postnatal) (p = 0,01). El análisis metagenómico demostró que la vía metabólica más enriquecida en las heces maduras (32-40 semanas PMA) en comparación con las menos maduras (25- 28 semanas PMA) era la de la biosíntesis de ácidos biliares secundarios.
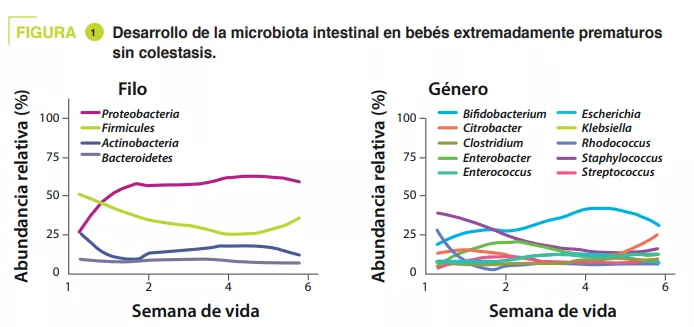
En los testigos, el análisis de componentes principales demostró que el principal factor que influía en la composición de la microbiota intestinal es la PMA, mientras que no afectaba a los prematuros con colestasis. La vía de biosíntesis de ácidos biliares secundarios era la más enriquecida en las heces de los testigos frente a las de los colestásicos a las 32-40 PMA (p = 0,04). Del mismo modo, los autores observaron una disminución del 55 % de la abundancia relativa del gen BSH (p = 0,04) y de Clostridium perfringens (p = 0,0008) en los neonatos con colestasis (figura 2).
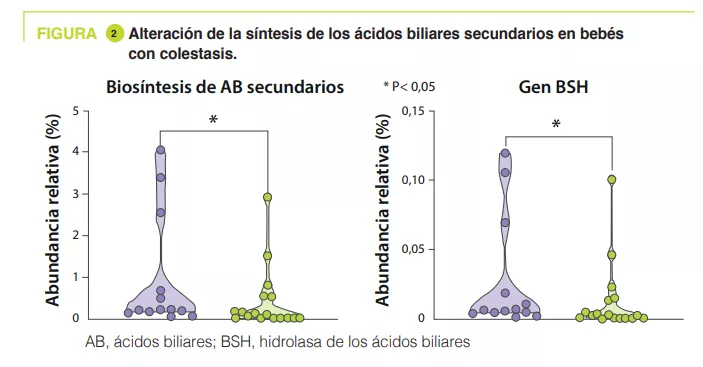
El perfil de los ácidos biliares fecales medido por espectrometría de masas mostró que la proporción de ácidos biliares no conjugados aumentaba de un 4 % a las 25-28 semanas PMA hasta un 98 % a las 32-40 semanas PMA en los testigos, pero solo hasta 46 % en los neonatos con colestasis. Cabe señalar que ciertos isómeros podrían tener un valor predictivo, porque aumentaban antes del inicio de la colestasis. El UDCA, utilizado en cinco de los 12 neonatos prematuros, se encuentra en sus heces con una concentración 522 veces superior a los otros 7 no tratados.
El UDCA modificó la microbiota intestinal con un aumento relativo de Firmicutes y una disminución de Proteobacteria (p <0,05), y en cuanto a las especies, con un enriquecimiento de Clostridium perfringens.
Por último, los bebés prematuros con una abundancia fecal del gen BSH > 0,005 % a las 32-40 semanas PMA presentaron velocidades de crecimiento estatural y ponderal incrementadas en un factor de 1,2 en comparación con los que presentaban una abundancia 30 % se relacionó con un aumento medio de las velocidades de crecimiento de la altura (14 %), del peso (18 %) y del perímetro cefálico (15,8 %) (p < 0,05).
¿Cuáles son las consecuencias en la práctica?
Este estudio permite comprender mejor los mecanismos fisiopatológicos implicados en la desregulación del eje hígado-intestino-microbiota intestinal durante la colestasis. Es posible que esto permita plantear una corrección del ciclo entero-hepático con ayuda de ciertos probióticos u otros medicamentos (basados en la actividad de la BSH) u otros medicamentos.
CONCLUSIÓN
En los recién nacidos extremadamente prematuros, la colestasis altera el desarrollo de la microbiota intestinal, reduciendo la adquisición de Clostridium perfringens y la capacidad de sintetizar ácidos biliares secundarios. Por el contrario, un aumento de ciertos ácidos biliares, relacionado con la actividad de la BSH, se asocia a un mejor crecimiento neonatal.
El ácido indol-3-acético (3-IAA) derivado de la microbiota intestinal influye en la eficacia de la quimioterapia en el cáncer de páncreas
ARTÍCULO COMENTADO
Por el Prof. Harry Sokol
Gastroenterología y nutrición, Hospital Saint-Antoine, París, Francia
Comentario del artículo de Tintelnot et al. Nature 2023 [1]
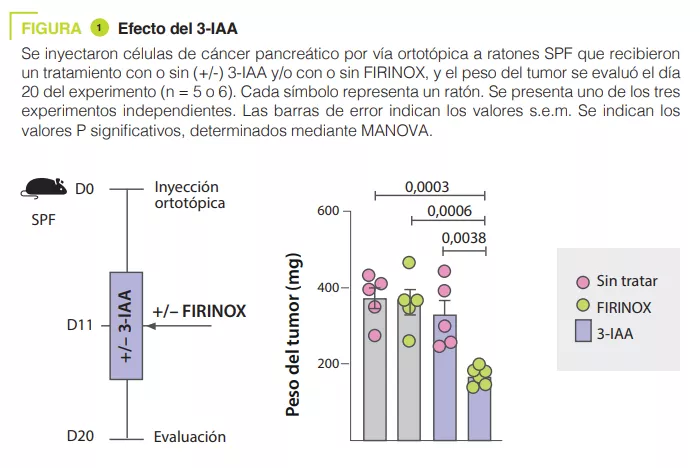
Se prevé que el adenocarcinoma ductal de páncreas (PDAC) sea el segundo cáncer con mayor mortalidad de aquí a 2040, debido a la alta incidencia de la enfermedad metastática y a las respuestas limitadas al tratamiento. Menos de la mitad de los pacientes responden favorablemente a la quimioterapia, el tratamiento principal del PDAC, y las variaciones genéticas entre los pacientes no son suficientes para explicar las diferencias en las respuestas. Es posible que la alimentación sea uno de los factores ambientales que repercuten en la respuesta a los tratamientos, sin embargo, su papel en el PDAC no está claro. Aquí, mediante la secuenciación metagenómica y un cribado metabolómico, los autores han demostrado que el ácido indol-3-acético (3-IAA), un metabolito del triptófano derivado de la microbiota, se encuentra enriquecido en los pacientes que responden favorablemente al tratamiento. El trasplante de microbiota fecal, las intervenciones nutricionales con triptófano y la administración oral de 3-IAA, aumentan la eficacia de la quimioterapia en modelos murinos humanizados de PDAC. Mediante una combinación de experimentos de pérdida y ganancia de función, los autores han demostrado que la eficacia del 3-IAA y de la quimioterapia requiere la presencia de la mieloperoxidasa (MPO) procedente de los neutrófilos. La MPO oxida el 3-IAA que, en combinación con la quimioterapia, provoca una regulación a la baja de enzimas que degradan las especies reactivas del oxígeno (ROS) como la glutatión peroxidasa 3 y la glutatión peroxidasa 7. Todo ello conduce a una acumulación de ROS y a una regulación a la baja de la autofagia en las células cancerosas, lo que repercute en su capacidad metabólica y, en última instancia, en su proliferación. En humanos, los autores han observado una correlación significativa entre los niveles de 3-IAA y la eficacia del tratamiento en dos cohortes independientes de PDAC. En resumen, los autores han identificado un metabolito derivado de la microbiota que tiene implicaciones clínicas en el tratamiento del PDAC, y han dado una justificación para plantear intervenciones nutricionales durante el tratamiento de los pacientes con cáncer.
¿Qué se sabe ya sobre este tema?
La poliquimioterapia, bien con 5-fluorouracilo (5-FU), irinotecán y oxaliplatino en combinación con ácido folínico (FOLFIRINOX), bien con gemcitabina y nabpaclitaxel (Abraxane), se considera el tratamiento estándar para pacientes con PDAC metastásico (mPDAC). Sin embargo, menos de la mitad de los pacientes responden favorablemente al tratamiento, y los pacientes que no lo hacen (pacientes que no responden, NR) tienen dolor y mueren a las pocas semanas. Las alteraciones genéticas del PDAC no bastan para explicar las diferencias entre los pacientes que responden favorablemente al tratamiento (pacientes que responden, R) y los pacientes NR, lo que convierte a los factores ambientales, incluida la microbiota intestinal, en posibles mediadores de la eficacia de la quimioterapia. Por lo tanto, urge identificar los factores ambientales que podrían explicar las diferencias entre pacientes R y NR, para desarrollar nuevas ideas para futuros tratamientos. Se ha demostrado que la microbiota intestinal interviene en la respuesta a la inmunoterapia en pacientes con melanoma y muchos otros tipos de cáncer, y que puede modularse mediante la alimentación 2, 3. En los poco frecuentes pacientes con PDAC localizado y supervivientes a largo plazo, las bacterias pueden pasar del intestino al tumor y controlar la activación inmunitaria antitumoral. Sin embargo, la mayoría de pacientes con PDAC agresivo y resistente a la inmunoterapia reciben un tratamiento de poliquimioterapia, y aún se desconoce si la microbiota o los hábitos alimenticios influyen en su eficacia y de qué manera 4 .
- La microbiota intestinal es diferente en pacientes con PDAC que responden, o no, a la quimioterapia
- El metabolito del triptófano 3-IAA, producido por la microbiota, está enriquecido en pacientes que responden al tratamiento
- El 3-IAA aumenta la eficacia de la quimioterapia aumentando la producción de ROS mediante los neutrófilos y disminuyendo la autofagia en las células cancerosas
¿Cuáles son los principales resultados aportados por este estudio?
El análisis de la microbiota de 30 pacientes con mPDAC reveló diferencias entre los pacientes R y los NR. La transferencia de microbiota de los R a ratones con tumores pancreáticos permitió reducir el tamaño de los tumores tras la quimioterapia. El metabolito del triptófano 3-IAA se enriqueció en los pacientes R y en los ratones con una microbiota R, posiblemente contribuyendo a la respuesta a la quimioterapia. La administración de 3-IAA mejoró la eficacia de la quimioterapia en los ratones (figura 1). El análisis de las células inmunitarias en los ratones mostró un aumento de linfocitos T CD8+ y una disminución de neutrófilos en ratones con una microbiota asociada a una buena respuesta a la quimioterapia. El 3-IAA afectó a la MPO de los neutrófilos, lo que redujo su supervivencia. La combinación del 3-IAA y de la quimioterapia redujo el número de neutrófilos e inhibió el crecimiento del tumor, en lo que la MPO desempeñó un papel fundamental. Se ha sugerido que la MPO induce la producción de ROS, lo que conduce a la muerte celular durante la quimioterapia. Los experimentos in vitro han demostrado que el 3-IAA aumenta los niveles de ROS. Este efecto se confirmó in vivo, y se observó que la inhibición de las ROS mediante la N-acetilcisteína eliminaba la eficacia de FIRINOX en ratones que presentaban niveles elevados de 3-IAA. A continuación, los autores demostraron que el efecto del 3-IAA estaba relacionado con una regulación a la baja de la autofagia. Por último, las concentraciones séricas elevadas de 3-IAA se correlacionaban con un descenso del recuento de neutrófilos y una mejora de la supervivencia en dos cohortes de pacientes humanos.
¿Cuáles son las consecuencias en la práctica?
La microbiota intestinal repercute en la respuesta a la quimioterapia. Los mecanismos implicados en este estudio demuestran el papel de los metabolitos microbianos y, en particular, de los metabolitos del triptófano. Entre ellos, el 3-IAA no solo es un marcador predictivo de la respuesta a la quimioterapia en el PDAC, sino que también podría representar una molécula terapéutica adyuvante.
CONCLUSIÓN
La microbiota intestinal repercute en la respuesta a la quimioterapia en el PDAC. Uno de sus metabolitos, el 3-IAA, es predictor de una buena respuesta a la quimioterapia y aumenta su efecto al provocar una acumulación de ROS y una reducción de la autofagia de las células cancerosas.
1.Tintelnot J, Xu Y, Lesker TR, et al. Microbiota-derived 3-IAA influences chemotherapy efficacy in pancreatic cancer. Nature 2023 ; 615 : 168-74.
2. Baruch EN, Youngster I, Ben-Betzalel G, et al. Fecal microbiota transplant promotes response in immunotherapy-refractory melanoma patients. Science 2021 ; 371 : 602-9.
3. Spencer CN, McQuade JL, Gopalakrishnan V, et al. Dietary fiber and probiotics influence the gut microbiome and melanoma immunotherapy response. Science 2021 ; 374 : 1632-40.
4. Thomas RM, Jobin, C. Microbiota in pancreatic health and disease: the next frontier in microbiome research. Nat Rev Gastroenterol Hepatol 2020 ; 17 : 53-64.
Microbioma intestinal y resistencia a los antimicrobianos
Por el Dr. Joseph Nesme
Profesor adjunto, Sección de Microbiología, Departamento de Biología, Universidad de Copenhague, Dinamarca
La resistencia a los antibióticos es antigua y está extendida, y el resistoma ambiental es un reservorio de genes de resistencia que agrava la pandemia silenciosa de resistencia a los antimicrobianos. La transferencia genética horizontal, la dinámica de poblaciones y la coselección aceleran la aparición y la diseminación del resistoma, de modo que para el 2050, más de 10 millones de muertes al año estarán causadas por la resistencia a los antibióticos. La colonización temprana del microbioma intestinal puede conducir a disbiosis que desemboquen en una mayor abundancia y diversidad del resistoma intestinal de los lactantes. Adoptar el enfoque “Una sola salud”, es decir, tener en cuenta la interconexión de la salud humana, la salud animal y el medioambiente es fundamental para hacer frente a los complejos retos que plantea la resistencia a los antibióticos.
La resistencia a los antibióticos en el medioambiente: Un reto preexistente
La resistencia a los antibióticos (RA) es un fenómeno antiguo y extendido en el medioambiente, que existe desde mucho antes de la introducción de moléculas antibióticas como tratamiento. El medioambiente es un amplio reservorio de genes de resistencia a los antibióticos con comunidades microbianas diversas que albergan mecanismos de resistencia. Se ha observado RA en numerosos entornos ambientales, como el suelo, el agua, las plantas, los animales e incluso en el permafrost ártico de 30 000 años de antigüedad 1, 2 . La función ecológica de las moléculas antibióticas y la resistencia asociada en entornos no clínicos sigue sin estar clara, pero pone de relieve el hecho de que un conjunto de genes fácilmente disponible es anterior al uso clínico de los antibióticos, y explica la rápida aparición de patógenos. La actual crisis de los antibióticos es un fenómeno evolutivo, y las estrategias de mitigación deben tener en cuenta la ecología microbiana. Lo problemático es la rápida adquisición de resistencia por parte de patógenos que antes eran sensibles a los antibióticos, lo que lleva a los fracasos terapéuticos, sobre todo cuando está previsto que solo se comercialicen unos pocos antimicrobianos nuevos en el futuro 3 .
Mecanismos que fomentan la aparición y la diseminación del resistoma
El resistoma se refiere a la totalidad de genes que codifican las proteínas relacionadas con la resistencia a los antibióticos (RA) o las proteínas que tienen la capacidad de convertirse en agentes potentes de RA 4 (figura 1). Esto incluye genes de RA reconocidos en bacterias patogénicas (las problemáticas), genes de RA de organismos que producen antibióticos, como Streptomyces spp. que produce el antibiótico estreptomicina y el gen de resistencia asociado 5 , genes de RA crípticos (es decir, genes que podrían ofrecer resistencia en un entorno genético diferente, por ejemplo, bombas de eflujo reguladas al alza o porinas reguladas a la baja), y genes precursores de la RA que codifican proteínas con un nivel mínimo de afinidad o resistencia a los compuestos antibióticos.
Hay que destacar que una parte considerable de estos subconjuntos de genes se solapa debido a la homología de las secuencias, lo que indica antecedentes evolutivos comunes.
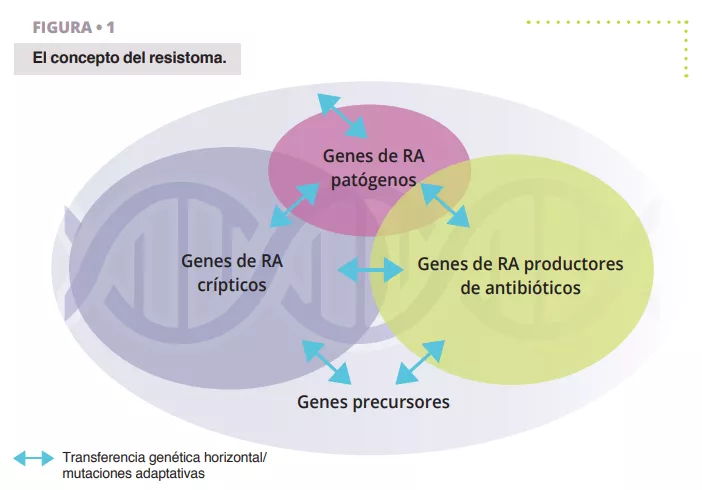
Es importante señalar que la noción de resistoma es diferente de una “RA funcional y clínicamente relevante”. Los genes del resistoma pueden pasar por los distintos estados descritos más arriba mediante transferencia genética horizontal (TGH), mutaciones puntuales o recombinación, lo que da lugar a nuevos anfitriones o contextos genéticos en los que puede expresarse el fenotipo de RA clínicamente relevante. Por lo tanto, un gen de resistencia no es problemático en sí, ya que depende del anfitrión y del contexto genético, pero todos los genes del resistoma representan una amenaza potencial con distintos riesgos asociados para la salud pública. El descubrimiento de un gen de resistencia a una molécula clínicamente relevante, localizado en un elemento móvil y alojado en un patógeno humano es un riesgo crítico, pero el mismo gen, o sus homólogos cercanos, encontrados en una bacteria del suelo no patógena y no asociado a un elemento genético móvil es un gen de RA de muy bajo riesgo. Por lo tanto, es fundamental clasificar el riesgo de resistencia a los antibióticos en los estudios sobre el resistoma.
La transferencia genética horizontal (TGH) es un mecanismo clave responsable de la rápida propagación de genes de RA entre las bacterias, incluso entre especies lejanas. Por ejemplo, Bacteroides spp., un grupo predominante en la microbiota intestinal, posee un gen de resistencia a los macrólidos ermB idéntico a los que se encuentran en numerosos Clostridium perfringens, Streptococcus pneumoniae y Enterococcus faecalis aislados de distintos orígenes, que indican la conexión genética entre Bacteroides y algunas bacterias grampositivas que no son predominantes en el intestino humano 6. Los elementos genéticos, como los plásmidos, facilitan la transferencia de genes de resistencia entre distintas especies microbianas 7. La TGH posibilita la diseminación de genes a través de distintos entornos y poblaciones bacterianas, lo que contribuye a la prevalencia y a la diversidad general de la RA. La coselección es otro factor importante en la propagación de la RA. El uso de compuestos no antibióticos, como metales pesados y biocidas, puede coseleccionar genes de RA ejerciendo presiones selectivas sobre las poblaciones microbianas, ya sea por corresistencia (distintos factores determinantes de resistencia presentes en el mismo elemento genético) o resistencia cruzada (el mismo factor determinante genético responsable de la resistencia a los antibióticos y a los metales) 8. La exposición a compuestos antimicrobianos de origen natural, como los producidos por microorganismos competidores o cualquier compuesto coselectivo, puede impulsar la selección de cepas resistentes 9. La presencia de antibióticos en el medioambiente, ya sean procedentes de fuentes naturales o de la actividad humana, contribuye aún más a la presión de selección de las resistencias. Además, el uso de antibióticos en la agricultura y en las prácticas veterinarias pueden conducir a la contaminación del medioambiente, lo que, con el tiempo, favorece la aparición y la propagación de genes de resistencia ambiental a los antibióticos 10.
Comprender las barreras y las tendencias de las transferencias de vectores genéticos que albergan genes de resistencia es fundamental para diseñar estrategias de mitigación ecoevolutivas e informadas para limitar la diseminación de la RA en los primeros años de vida y, en general, en contextos clínicos.
Disbiosis y resistoma intestinal en lactantes: Un equilibrio delicado
La diversidad del reservorio ambiental de genes de RA y su potencial para ser transferidos representa una amenaza para el microbioma intestinal humano en los primeros años de vida. Estrategias como un mejor tratamiento de las aguas residuales, un uso responsable de los antibióticos en la agricultura y en la medicina veterinaria, y una reducción de la contaminación ambiental con residuos antibióticos y bacterias resistentes a los antibióticos pueden ayudar a mitigar la propagación de la resistencia 11. Además, el seguimiento y la vigilancia de los reservorios ambientales puede proporcionar información valiosa sobre la aparición y la persistencia de la resistencia a los antibióticos para adaptar las intervenciones de salud pública.
Adoptar el enfoque “Una sola salud”, tener en cuenta la interconexión de la salud humana, la salud animal y el medioambiente es fundamental para hacer frente a los complejos retos que plantea la RA.
Después de nacer, el intestino es colonizado rápidamente por microorganismos adquiridos de la madre de su entorno. Durante los primeros años de vida es cuando se producen cambios drásticos, caracterizados por una baja resistencia, en comparación con el microbioma intestinal más estable de un adulto sano. Por lo tanto, los recién nacidos y los lactantes son más propensos a alteraciones en las comunidades microbianas, lo que se conoce como disbiosis. Durante ese período, hay numerosos factores que pueden influir y perturbar la maduración intestinal y, potencialmente, tener consecuencias a largo plazo sobre la salud 12. Estudios con ratones han demostrado que, durante este intervalo decisivo para el desarrollo, en lugar de un efecto directo de las moléculas antibióticas, es la alteración de la composición de la microbiota intestinal la que desencadena consecuencias metabólicas, como la obesidad 13 .
Descifrar el resistoma antibiótico en el intestino de lactantes: Conclusiones de un extenso estudio de cohortes
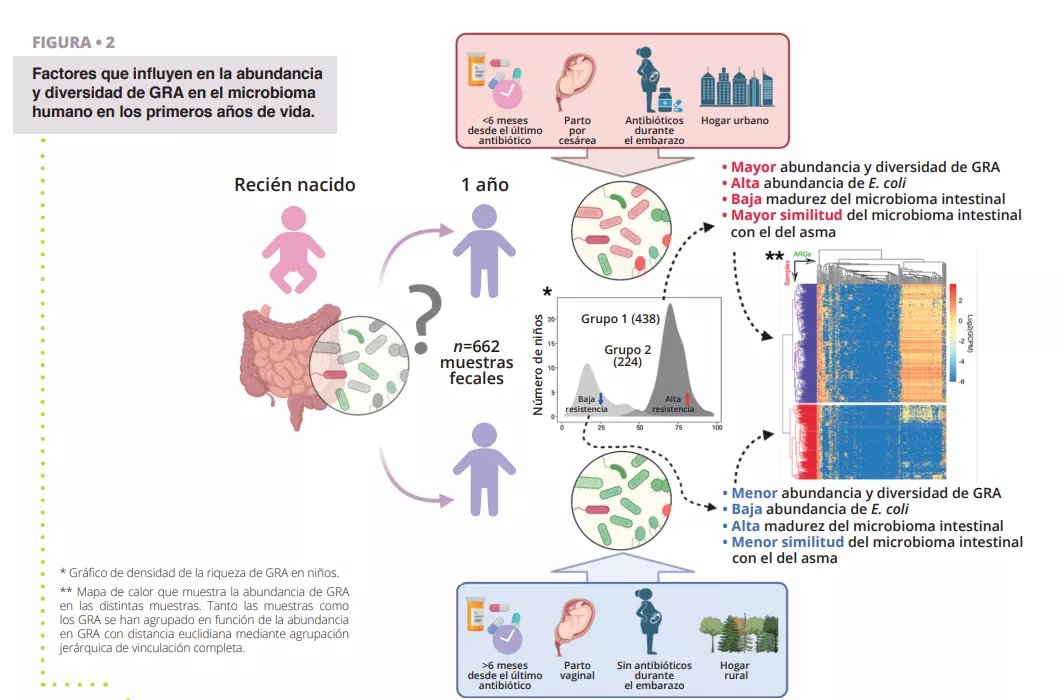
Aunque la resistencia a los antibióticos es problemática a cualquier edad, el establecimiento del microbioma intestinal en una edad temprana representa un intervalo de oportunidad para limitar el desarrollo de un reservorio de genes de RA en el intestino. Por lo tanto, es importante identificar los distintos factores que aumentan o reducen la abundancia de genes de RA que podrían propagarse a patógenos infecciosos y provocar el fracaso de tratamientos con antibióticos a lo largo de la vida. Para estudiar el resistoma global del intestino humano, los investigadores se sirven de planteamientos holísticos que interrogan tanto la presencia de especies como el potencial funcional de los genomas, incluido el resistoma bacteriano. Los investigadores utilizan la extracción de ADN ambiental a partir de muestras indirectas (como muestras de heces para el intestino) seguida de una secuenciación masiva no dirigida (metagenómica indiscriminada). Aproximadamente el 80 % de las especies de bacterias del intestino humano detectadas con herramientas moleculares no se pueden cultivar, en concreto, los anaerobios especializados que habitan en el intestino. Es probable que haya una gran cantidad de microorganismos organizados en agregados celulares de múltiples especies con codependencias metabólicas cuyo aislamiento puro de cepa sea difícil, si no imposible. Sin embargo, con métodos computacionales, es posible reconstruir genomas casi completos a partir de metagenomas y asociar los genes codificados a especies o incluso cepas concretas de bacterias. En un estudio reciente, los investigadores analizaron muestras fecales de 662 lactantes de una cohorte que hizo un seguimiento de los niños desde el nacimiento hasta los 7 años 14. Los objetivos del estudio eran establecer una visión general, a escala de cohorte, del resistoma al año e identificar los factores perinatales y ambientales asociados con la abundancia y la diversidad de GRA. Los investigadores realizaron una secuenciación metagenómica indiscriminada de muestras obtenidas al año de los 662 niños para identificar los GRA y los taxones bacterianos presentes en las muestras (figura 2). Aprovechando el extenso conjunto de datos, los autores pudieron reconstruir los genomas asociados a los metagenomas (GAM), lo que les permitió anotar con seguridad la taxonomía de los genomas recuperados y su contenido en genes de RA.
Uno de los primeros resultados observados fue que todos los niños tenían como mínimo un tipo de GRA a múltiples fármacos en el intestino, lo que indica que, incluso sin haber recibido un tratamiento antibiótico, existe un resistoma residente asociado al microbioma intestinal. De hecho, muchos genes de resistencia a múltiples fármacos se identificaron como bombas de eflujo. Estas proteínas son componentes normales de cualquier célula bacteriana, pero algunas pueden conferir resistencia a los antibióticos y son coseleccionadas con mucha facilidad por resistencia cruzada, por ejemplo, hacia metales pesados o biocidas, lo que posiblemente explica su gran abundancia en el intestino, pero también en todos los ambientes [8, 15]. Otro resultado sorprendente fue la clara división de la cohorte en dos grupos, en función del perfil de su resistoma. El primer grupo se caracterizó por una mayor diversidad de GRA y una abundancia relativa, con Escherichia coli como el mayor contribuidor de GRA, como se muestra en la figura 2. Esto concuerda con observaciones anteriores según las cuales Enterobacteriaceae son abundantes en los primeros años de vida, pero deberían disminuir rápidamente cuando la población de Bacteroidetes empieza a colonizar el intestino. En algunos niños, la alteración de esta maduración se puede asociar a la conjunción de varios factores, como el consumo de antibióticos, el tipo de parto, haberse criado en un entorno rural o urbano, factores que pueden retrasar la reducción de la población de Escherichia coli y conducir a un aumento del resistoma al año. Esto también se confirma por la observación de que una mayor abundancia de GRA se asocia con una menor puntuación de madurez del microbioma intestinal, basada en ratios de taxones específicos relacionados con la edad 16 .
Obviamente, el tratamiento con antibióticos durante el embarazo y el primer año de vida tuvo un impacto significativo en la abundancia del resistoma intestinal del lactante. Se pudo observar una clara tendencia temporal entre el tiempo (más de 6 meses o menos) desde la última toma de antibiótico de un niño y la abundancia de GRA en el intestino.
Esto indica una capacidad de recuperación a una edad temprana, que quizá se podría mejorar con una intervención dirigida, por ejemplo, con probióticos o prebióticos, y que aún se debe probar. Sin embargo, a escala de cohorte, las mayores diferencias en la abundancia de GRA se encontraron en genes de resistencia a antibióticos que no se recetaron a los niños, lo que indica que factores perinatales y ambientales, además del tratamiento con antibióticos, también influyen en el resistoma intestinal. Otra observación que se desprende de este estudio que conecta el entorno circundante y su resistoma asociado, y el resistoma intestinal, fue que los niños cuyos hogares estaban en zonas urbanas tenían una carga significativamente mayor de GRA que los niños que vivían en zonas rurales. Hay un sinfín de posibles factores de confusión que podrían explicar esto, pero refuerza el hecho de que la contribución del entorno al desarrollo del microbioma en los primeros años de vida es extremadamente importante.
Se puede plantear la hipótesis de que la vida urbana se asocia a un menor contacto con el exterior y a una menor diversidad del microbioma, o que el tipo de vivienda de los entornos rurales (casas) o urbanos (apartamentos) repercute en el microbioma interior, como se ha observado en el polvo de las camas.
Conclusión
La resistencia a los antibióticos es antigua y está extendida, y el resistoma ambiental es un reservorio de posibles genes de resistencia clínica. Los mecanismos como la transferencia genética horizontal, la compleja dinámica de poblaciones y los efectos de la coselección se han identificado como aceleradores en la evolución y propagación del resistoma antibiótico, lo que ha hecho que las cepas de patógenos que antes eran sensibles al tratamiento antibiótico ahora sean resistentes. El microbioma intestinal humano y su fase de desarrollo inicial son propensos a la disbiosis y a la posible invasión, selección y coselección de bacterias que albergan genes de resistencia, lo que puede tener consecuencias para la salud a largo plazo. No está claro hasta qué punto estarán relacionados con el fracaso de futuros tratamientos con antibióticos. Se necesitan estudios multiómicos a largo plazo y de alta resolución temporal para identificar, en primer lugar, los patrones longitudinales de desarrollo del microbioma intestinal y, en segundo lugar, las vías más probables de transferencia de genes de resistencia a los antibióticos mediante TGH. El conjunto de estos resultados ofrecerá información para diseñar estrategias de prevención que permitan minimizar el desarrollo de la RA desde las primeras etapas de la vida y limitar la transferencia a patógenos problemáticos. Este esfuerzo es de la máxima importancia para reducir las consecuencias de la pandemia mundial de resistencia a los antimicrobianos y mejorar la salud pública mundial, así como para mantener la eficacia de los pocos antibióticos disponibles para luchar contra las enfermedades infecciosas.
1. Nesme J, Cécillon S, Delmont TO, et al. Large-scale metagenomic-based study of antibiotic resistance in the environment. Curr Biol 2014; 24: 1096-100.
2. D’Costa VM, King CE, Kalan L, et al. Antibiotic resistance is ancient. Nature 2011; 477: 457-61.
3. Renwick MJ, Brogan DM, Mossialos E. A systematic review and critical assessment of incentive strategies for discovery and development of novel antibiotics. J Antibiot 2016; 69: 73-88.
4. Wright GD. The antibiotic resistome: the nexus of chemical and genetic diversity. Nat Rev Microbiol 2007; 5: 175-86.
5. Benveniste R, Davies J. Aminoglycoside antibiotic-inactivating enzymes in actinomycetes similar to those present in clinical isolates of antibiotic-resistant bacteria. Proc Natl Acad Sci USA 1973; 70: 2276-80.
6. Shoemaker NB, Vlamakis H, Hayes K, et al. Evidence for extensive resistance gene transfer among Bacteroides spp. and among Bacteroides and other genera in the human colon. Appl Environ Microbiol 2001; 67: 561-8.
7. Martínez JL, Coque TM, Baquero F. Prioritizing risks of antibiotic resistance genes in all metagenomes. Nat Rev Microbiol 2015; 13: 396.
8. Baker-Austin C, Wright MS, Stepanauskas R. Co-selection of antibiotic and metal resistance. Trends Microbiol 2006; 14: 176-82.
9. Wencewicz TA. Crossroads of Antibiotic Resistance and Biosynthesis. J Mol Biol 2019; 431: 3370-99.
10. Knapp CW, Dolfing J, Ehlert PA, et al. Evidence of increasing antibiotic resistance gene abundances in archived soils since 1940. Environ Sci Technol 2010; 44: 580-7.
11. Wolters B, Hauschild K, Blau K, et al. Biosolids for safe land application: does wastewater treatment plant size matters when considering antibiotics, pollutants, microbiome, mobile genetic elements and associated resistance genes? Environ Microbiol 2022; 24: 1573-89.
12. Dogra S, Sakwinska O, Soh SE, et al. Rate of establishing the gut microbiota in infancy has consequences for future health. Gut Microbes 2015; 6: 321-5.
13. Cox LM, Yamanishi S, Sohn J, et al. Altering the intestinal microbiota during a critical developmental window has lasting metabolic consequences. Cell 2014; 158: 705-21.
14. Li X, Stokholm J, Brejnrod A, et al. The infant gut resistome associates with E. coli, environmental exposures, gut microbiome maturity, and asthma-associated bacterial composition. Cell Host Microbe 2021; 29: 975-87.e4.
15. Mulder I, Siemens J, Sentek V, et al. Quaternary ammonium compounds in soil: implications for antibiotic resistance development. Rev Environ Sci Bio/Technology 2018; 17: 159-85.
16. Subramanian S, Huq S, Yatsunenko T, et al. Persistent gut microbiota immaturity in malnourished Bangladeshi children. Nature 2014; 510: 417-21.
¿Una bacteria para prevenir la diabetes?
Por el momento, este sueño solo se ha hecho realidad en tubos de ensayo y en ratones. Falta concretarlo en el ser humano. La bacteria A. indistinctus podría mejorar la resistencia a la insulina de las personas prediabéticas.
La microbiota intestinal Diabetes de tipo 2
(sidenote: Diabetes Enfermedad crónica que surge cuando el páncreas no produce suficiente insulina o cuando el cuerpo no es capaz de utilizar eficazmente la insulina que produce. Con el paso del tiempo, la diabetes puede causar lesiones vasculares en el corazón, los ojos, los riñones y los nervios. ) is all about sugar, and more specifically, blood sugar levels ( (sidenote: Glucemia Nivel de azúcar en la sangre. ) ). The aim is to avoid peaks (i.e., hyperglycemia) following meals. In healthy individuals, (sidenote: Insulina Hormona producida por el páncreas que se encarga de reducir el nivel de azúcar hasta aproximadamente 1 g/l. ) regulates blood sugar levels at around 1 g/L. In diabetic patients, the body fails in this regard, either because the pancreas does not produce enough insulin (type I diabetes mellitus), or because the body resists insulin’s commands (type II diabetes mellitus). The gut microbiota appears to be involved in the insulin resistance observed in type II diabetes, but the mechanisms involved remain unclear. Or rather “remained” unclear, since a study published in the prestigious journal Nature has shed some light on the subject.
Los miniazúcares favorecen la resistencia a la insulina
Los investigadores observaron que cuanto más abundantes ciertas moléculas en las heces, más elevado el nivel de insulinorresistencia. Las heces de los voluntarios resistentes a la insulina contenían mucho más (sidenote: Carbohidratos Familia de macronutrientes que incluye los azúcares (o «carbohidratos simples») que generalmente tienen un sabor dulce (glucosa, fructosa, galactosa, maltosa, lactosa, sacarosa) y los almidones (o «carbohidratos complejos»). ) , especialmente (sidenote: Miniazúcares Fructosa, galactosa, manosa y xilosa. ) producidos por bacterias. Estos miniazúcares, lejos de ser inofensivos, favorecen la acumulación de grasas, estimulan el sistema inmunitario a tal punto que llegan a provocar inflamación (leve, pero prolongada y nociva) y finalmente conducen a la resistencia a la insulina.
2 millones de muertes relacionadas con la diabetes (1,5 millones) y con enfermedades renales causadas por la diabetes (0,5 millones) en 2019.
422 millones 108 millones de diabéticos en 1980 frente a 422 millones en 2014.
+3% Entre 2000 y 2019, la tasa de mortalidad debida a la diabetes según la edad aumentó un 3%.

Bacterias implicadas
Dos tipos de bacterias parecen estar implicadas:
- bacterias de la familia Lachnospiraceae que se asocian con una mayor producción de estos miniazúcares y resistencia a la insulina.
- y bacterias de tipo Bacteroidales (Bacteroides, Alistipes y Flavonifractor) que reducen estos azúcares y la resistencia a la insulina.
De hecho, se ha observado un efecto directo de las bacterias «beneficiosas» de la familia Bacteroidales dado que, en tubos de ensayo, estas bacterias devoran los miniazúcares. ¿Cuál es la más voraz? La especie Alistipes indistinctus es la que consume la variedad más amplia de miniazúcares. Y esto también es cierto en ratones obesos: una pizca de A. indistinctus reduce la cantidad de miniazúcares en las heces y la glucemia de los animales, y mejora su sensibilidad a la insulina.
Estos resultados tienen dos consecuencias, aunque por supuesto es imprescindible confirmarlos mediante estudios en el ser humano antes de emitir cualquier recomendación:
• Lachnospiraceae podría ser un biomarcador de la prediabetes;
• y el probiótico A. indistinctus podría mejorar la sensibilidad a la insulina en las personas prediabéticas.
La microbiota intestinal
 Diagnóstico más temprano de la diabetes gestacional gracias a la microbiota
Diagnóstico más temprano de la diabetes gestacional gracias a la microbiota







