O impacto da gravidade na síndrome do intestino irritável
Pela Dra. Maria Teresa Galiano
Serviço de Gastrologia e Endoscopia, Servimed, Bogotá, Colômbia

Num artigo recente [1], foi sugerido que a SII pode resultar de sistemas anatómicos, fisiológicos e neuropsicológicos ineficazes de gestão da gravidade, concebidos para otimizar a forma e a função gastrointestinal, proteger a integridade somática e visceral e maximizar a sobrevivência num mundo ligado à gravidade.
Pode comentar esta hipótese com o seu ponto de vista clínico?
Esta hipótese é muito interessante. Penso que pode ser considerada entre as múltiplas hipóteses que procuram explicar a síndrome do cólon irritável. No entanto, tem de ser testada. Devem ser realizados estudos que comprovem que as alterações físicas devidas às mudanças de gravidade afetam a fisiologia gastroenterológica. Penso que pode ser verdade que a gravidade afeta a fisiologia de um organismo e que, normalmente, estamos em equilíbrio com esta força permanente a que o homem e todos os seres vivos da Terra estão sujeitos. As consequências que surgem quando este equilíbrio é alterado podem incluir a SII.
Concorda com a explicação do autor de que as consequências da gravidade resultam numa alteração da microbiota intestinal?
Concordo com o autor que a gravidade pode alterar a microbiota intestinal e que pode também alterar o seu funcionamento, incluindo a fermentação. Penso que também pode alterar o volume de gás que atua sobre a parede do intestino. Isto também tem de ser testado nos estudos correspondentes, mas concordo com o ponto de vista do autor relativamente à suscetibilidade da microbiota intestinal à gravidade.
Partilharia esta hipótese com os seus doentes?
Partilho esta hipótese com os pacientes a quem acredito que ela se pode aplicar com base na sua fisiopatologia e como uma possível explicação para os seus sintomas. De facto, observo alterações nos meus doentes quando viajam para locais que estão ao nível do mar e regressam a Bogotá, onde vivo. Bogotá está situada a 2.600 metros acima do nível do mar. Quando estes doentes regressam a Bogotá, apresentam mais sintomas devido às alterações da pressão barométrica. As mudanças na pressão barométrica causam mudanças na sensação, distensão e gases nos intestinos. Em muitas ocasiões, explico a sua sintomatologia com base nas alterações da sua fisiologia resultantes das diferentes altitudes que experimentam. As alterações que ocorrem devido a variações no equilíbrio com a força da gravidade podem ser utilizadas para explicar os sintomas destes doentes.
A Microbiota vaginal #19
Pela Prof. Satu Pekkala
Investigador na Academia da Finlândia, Faculdade de Ciências do Desporto e da Saúde, Universidade de Jyväskylä, Finlândia
Cancro do colo do útero: os muros estão a fechar-se para Lactobacillus iners
O papilomavírus humano de alto risco (HR-HPV) é a principal causa de cancro do colo do útero. A atividade sexual, o tabagismo e a contraceção oral estão entre os muitos fatores que influenciam a infeção inicial, a resolução ou o aparecimento do HR-HPV e a potencial progressão para cancro. Nos últimos anos, a microbiota vaginal foi acrescentada a esta lista. O valor deste estudo é, portanto, claro, uma vez que monitorizou a microbiota cervical de mulheres chinesas infetadas com HR-HPV que apresentavam lesões cervicais histologicamente confirmadas e, na sua maioria, de baixo grau. Uma análise do rRNA 16S da microbiota das 73 participantes no estudo (com idades compreendidas entre os 24 e os 68 anos) mostrou que o HPV tinha desaparecido em 45 das mulheres (61,6%) no final do período de estudo de doze meses. A eliminação ou não do vírus não se deveu a diferenças na idade das pacientes, na fase da doença, no subtipo de HPV, no tipo de comunidade bacteriana vaginal ou na diversidade da microbiota vaginal. Em vez disso, certas espécies bacterianas parecem estar envolvidas: as mulheres empobrecidas em enterococo ASV_62 e enriquecidas em Lactobacillus iners na linha de base tinham menos probabilidades de ter uma eliminação do HPV-HR no mês doze. A única exceção foram 22 mulheres que foram submetidas a tratamento cirúrgico (conização) para lesões de alto grau, talvez porque o impacto imediato da ressecção da lesão na eliminação do HPV tenha mascarado o impacto da flora. Uma possível ligação entre a L. iners e o HR-HPV tinha sido anteriormente referida por uma meta-análise que sugeria um risco duas ou mesmo três vezes superior de HR-HPV persistente quando a microbiota vaginal é dominada pela L. iners. Esta bactéria parece ser flexível e adaptável, dominando a microbiota vaginal de certas mulheres durante a menstruação ou episódios de vaginose bacteriana. Por outro lado, uma microbiota vaginal dominada por L. iners (CST III) é frequentemente relatada como um dos tipos mais comuns de comunidade bacteriana vaginal entre mulheres asiáticas em idade fértil. Por conseguinte, a literatura atual ainda não esclarece se esta estirpe específica de Lactobacillus deve ser considerada benéfica, patogénica ou ambas. Também é necessário mais trabalho para esclarecer os mecanismos pelos quais o L. iners promove a infeção persistente pelo HPV ou a progressão da lesão, especialmente porque o presente estudo envolveu um pequeno número de pacientes.
A Microbiota Intestinal #19
Pela Prof. Satu Pekkala
Investigador na Academia da Finlândia, Faculdade de Ciências do Desporto e da Saúde, Universidade de Jyväskylä, Finlândia

Metabolismo microbiano do 5-ASA e doença do inflamatória intestinal
A doença inflamatória intestinal (DII) é tratada com ácido 5-aminosalicílico (5-ASA). No entanto, mais de metade dos doentes não responde ao tratamento. Estudos anteriores sugeriram que isto se deve em parte ao facto de o 5-ASA poder ser metabolizado pelas bactérias intestinais em N-acetil 5-ASA, que é clinicamente ineficaz. Neste elegante estudo, Mehta et al. tinham como objetivo identificar as enzimas microbianas intestinais que geram N-acetil 5-ASA. As amostras de fezes humanas foram analisadas com recurso à multi-ómica. As análises metabolómicas não direcionadas das amostras antes e depois da administração do 5-ASA revelaram potenciais mediadores microbianos dos efeitos anti-inflamatórios do 5-ASA. Estes incluíram uma diminuição do 2-aminoadipato, um metabolito bacteriano que tem sido associado a um maior stress oxidativo. Além disso, o 5-ASA parece alterar o metabolismo do nicotinato, o que também pode explicar alguns efeitos anti-inflamatórios. Os autores procuraram ainda identificar as enzimas microbianas que potencialmente metabolizam o 5-ASA. Combinando metatranscriptómica e metabolómica, identificaram três acetil-CoA C-acetiltransferases (Acil-CoA NAT) que se associaram aos níveis de N-acetil 5-ASA em utilizadores de 5-ASA. Além disso, foram identificadas algumas tiolases como potenciais enzimas candidatas. As enzimas candidatas foram então expressas heterologamente em Escherichia coli e a sua atividade bioquímica foi medida. A Firmicutes CAG:176 tiolase e a Faecalibacterium prausnitzii acilCoA NAT foram capazes de acetilar o 5-ASA utilizando acetil-CoA. Por fim, uma análise metagenómica das amostras de fezes revelou que as acetiltransferases inativadoras de 5-ASA do microbioma intestinal estavam associadas a um maior risco de insucesso do tratamento nos utilizadores de 5-ASA. No seu conjunto, os resultados deste estudo podem ajudar a avançar a possibilidade de um tratamento personalizado da DII baseado no microbioma.

Helicobacter pylori e cancro colorretal
A infeção por Helicobacter pylori pode levar ao cancro gástrico e aumentar o risco de cancro colorretal (CCR). No entanto, faltam dados mecanicistas sobre este último. Neste artigo, Ralser e colaboradores identificam, num modelo de roedor, os mecanismos subjacentes à forma como a infeção por H. pylori contribui para o CCR. Quando os autores infetaram com H. pylori ratinhos Apc, que são excelentes modelos animais portadores de neoplasias intestinais múltiplas, observou-se um aumento da carga tumoral no intestino delgado e no cólon. Sabe-se que a resposta imunitária das células T do hospedeiro contribui para a carcinogénese gástrica, pelo que os autores estudaram estas respostas no intestino. Verificaram uma redução das células T reguladoras e das células T pró-inflamatórias, bem como um aumento da IL-17A, que se revelou um dos principais intervenientes na resposta imunitária à H. pylori. Os ratinhos infetados caracterizavam-se por uma maior abundância dos chamados micróbios intestinais pró-inflamatórios e de bactérias que degradam o muco, como a Akkermansia. Ao estudar os perfis transcriptómicos das células epiteliais intestinais, descobriram que a H. pylori induziu a ativação das vias NF-κB e STAT3. A ativação destas vias foi anteriormente demonstrada também em doentes com CRC. Curiosamente, os ratinhos sem germes infetados com H. pylori quase não apresentaram ativação da sinalização STAT3, o que sugere que a carcinogénese induzida por H. pylori no intestino delgado depende em parte do microbioma intestinal. Em última análise, os autores mostram que a carcinogénese colorretal induzida por H. pylori pode ser prevenida através da erradicação da bactéria com antibióticos. Os autores concluem que a implementação do estado da H. pylori nas medidas preventivas do CCR deve ser considerada.
Diversidade viral no intestino de um bebé saudável
Na infância, o microbioma intestinal contribui para a maturação do sistema imunitário, protegendo-o contra doenças crónicas mais tarde na vida. Embora se saiba que os bacteriófagos (ou seja, vírus que infetam bactérias) podem controlar o crescimento das populações bacterianas, os viromas intestinais não foram extensivamente estudados. Utilizando a sequenciação do metagenoma, este estudo analisou 647 viromas de uma coorte dinamarquesa de bebés de 1 ano de idade. A primeira descoberta surpreendente foi que os vOTUs do intestino infantil estavam largamente ausentes das bases de dados de vírus do intestino. Isto sugere que o intestino do bebé alberga vírus especializados distintos do intestino do adulto. Os clados virais mais predominantes dos bebés não estavam, em grande parte, descritos. No entanto, os anellovírus ssDNA infetantes de vertebrados (Anelloviridae) e os microvírus ssDNA bacterianos (Petitvirales) encontravam-se entre os mais abundantes. Além disso, as famílias caudovirais virulentas Skunaviridae, Salasmaviridae, β-crassviridae e Flandersviridae também estavam bem representadas nos viromas infantis. Em geral, os vírus temperados eram menos prevalentes do que os virulentos, apesar de serem encontrados em mais crianças. A abundância ao nível da família não foi significativamente associada ao estilo de vida do fago, determinado pela integrase como um indicador de um estilo de vida temperado. No entanto, as famílias caudovirais temperadas eram geneticamente mais diversas do que as famílias virulentas. A análise de previsão dos hospedeiros bacterianos dos viromas mostrou que Bacteroides, Faecalibacterium e Bifidobacterium eram os três géneros hospedeiros mais proeminentes no intestino dos bebés. Destes, as famílias que infetam Bacteroides eram mais frequentemente virulentas e específicas do hospedeiro. Embora não tenham sido apresentadas conclusões claras, o estudo aumenta o conhecimento sobre a taxonomia dos fagos e ajuda no desenvolvimento de futuras investigações sobre o viroma intestinal infantil.

Destaques do 55º ESPGHAN
Pela Dra. Tania Mahler
Médico, Gastroenterologia Pediátrica e Nutrição, Clínico adjunto no Hospital Universitário Infantil Rainha Fabíola, Bélgica

Na reunião ESPGHAN (Sociedade Europeia de Gastroenterologia, Hepatologia e Nutrição Pediátrica) 2023, realizada em Viena, reuniram-se 4300 participantes de todo o mundo. Após o período desafiante da pandemia de Covid-19, este evento proporcionou uma experiência revitalizante, uma vez que os participantes puderam participar em apresentações ao vivo e ter discussões presenciais. Esta interação presencial provou ser significativamente mais agradável e enriquecedora do que as alternativas virtuais. Vários grupos de investigação centraram o seu trabalho no microbioma intestinal e apresentaram dados convincentes no domínio da pediatria.
O impacto do microbioma na saúde e na doença é amplamente reconhecido, pelo que é evidente que também os clínicos e investigadores pediátricos tentam compreender melhor como podemos manipular o microbioma e como, utilizando a assinatura do microbioma, podemos detetar a doença numa fase precoce. Esta revisão tem como objetivo lançar luz sobre vários tópicos-chave que têm sido amplamente discutidos
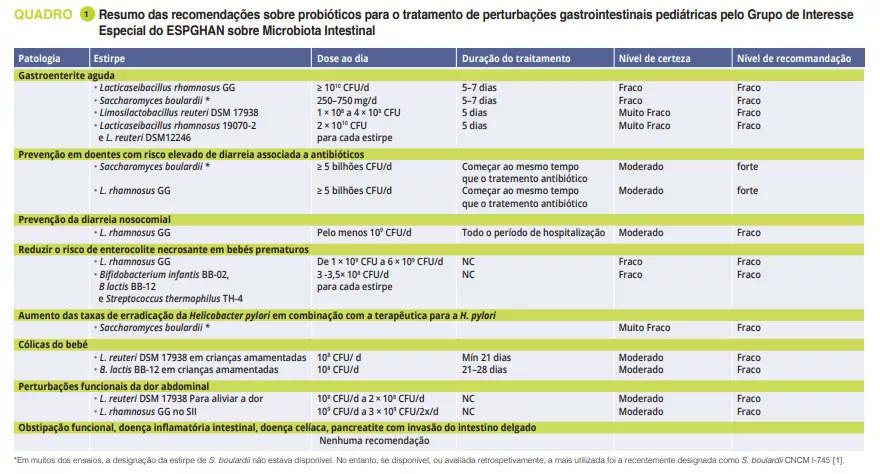
Recomendações para a utilização de probióticos em doenças gastrointestinais pediátricas selecionadas
Em fevereiro de 2023, o grupo de interesse especial da ESPGHAN sobre a microbiota intestinal publicou recomendações para a utilização de probióticos na gestão de perturbações gastrointestinais pediátricas selecionadas, com base em revisões sistemáticas e/ou meta-análises utilizando o processo Delphi- modificado [1]. Na reunião do grupo de interesse especial sobre microbiota intestinal e modificações, o Prof. Szajewska mostrou-nos os resultados deste trabalho. Apenas algumas estirpes probióticas específicas demonstram alguma utilidade em determinadas condições. No artigo original1 pode encontrar uma visão geral clara das recomendações atuais.
Transplante de microbiota fecal (FMT) em adolescentes que sofrem de síndrome do intestino irritável (SII) refratária
A Dra. De Bruijn, do grupo do UMC de Amesterdão, apresentou na sessão plenária do resumo com maior pontuação o seu estudo sobre a eficácia do FMT em adolescentes com SII refratária, num ensaio aleatório, em dupla ocultação e controlado por placebo [2]. A sua intervenção foi fascinante, mas também suscitou muitas reações na audiência quando mostrou um diapositivo de um doente a receber seringas com material fecal. Tanto quanto sabemos, apenas um outro estudo pediátrico avaliou o FMT para aliviar o inchaço abdominal, outra caraterística dos distúrbios da interação intestino-cérebro e que está frequentemente presente na SII [3].
A dor crónica na SII pode ter um enorme impacto no funcionamento de crianças e adultos, levando ao absentismo na escola e no trabalho e a uma má qualidade de vida. A origem da doença é multifatorial e pode ser melhor explicada pelo modelo biopsicossocial. Um dos principais fatores é a disbiose da microbiota intestinal. Em adultos, foram publicados diferentes estudos sobre o efeito positivo observado com o FMT [4].
Em pediatria, os tratamentos não farmacológicos como a educação, a hipnose e a atenção plena são mais eficazes do que a terapia farmacológica [5]. No entanto, em cerca de 25% dos doentes, os sintomas persistem. Os pré, pró e simbióticos são testados com vários resultados para corrigir a disbiose na SII. A mudança para uma dieta FODMAP também pode influenciar a flora gastrointestinal [5]. Mas em grupos específicos de pacientes, o FMT pode ser, se for segura, o tratamento definitivo para restaurar efetivamente um microbioma gastrointestinal saudável. No estudo de De Bruijn et al., 32 pacientes com SII refratária entre 16 e 21 anos de idade foram recrutados e randomizados. Um grupo recebeu infusões fecais alogénicas (de um dador saudável) e o outro grupo recebeu infusões fecais autólogas (do próprio) por sonda nasogástrica no início e 6 semanas depois. A eficácia clínica foi definida como a proporção de doentes com uma redução de mais de 50 pontos no IBS Severity-Scoring-System (IBS-SSS). Os pacientes foram avaliados 12 semanas e 6 meses após o TMF. Ambos os grupos tinham um IBS-SSS semelhante no início do tratamento. Após a primeira avaliação não houve diferença estatística, mas aos 6 meses de seguimento houve uma melhoria em 60% dos pacientes que receberam TMF alogénico versus 25% no grupo autólogo (p = 0,048). O resultado secundário incluiu a qualidade de vida relacionada com a saúde (QdV). A pontuação total da QdV na linha de base não diferiu entre os grupos, mas melhorou significativamente após o TMF alogénico. Não foram registados quaisquer eventos adversos. O TMF alogénico parece ser uma forma interessante de tratar a SII refratária em jovens, mas são necessários mais estudos.
Microbiota e doenças inflamatórias intestinais (IBD)
Na sessão de gastroenterologia sobre DII, um grupo checo apresentou um estudo que tinha como objetivo avaliar se as alterações na microbiota na DC se deviam ao tratamento anti-TNFα ou se eram o resultado de uma diminuição da atividade inflamatória da mucosa [6]? Assim, compararam crianças em tratamento anti-TNFα com doença de Crohn (DC) ativa e artrite idiopática juvenil (AIJ). Os seus resultados mostraram que a cicatrização da mucosa na DC era essencial para obter alterações no bacterioma. O tratamento anti-TNFα na AIJ não teve qualquer impacto no bacterioma deste grupo de doentes. Schwerd et al. acompanharam 20 pacientes pediátricos recém-diagnosticados com DC tratados primeiramente com nutrição enteral exclusiva (NEE) com amostragem de fezes [7]. Quinze dos vinte pacientes entraram em remissão. Eles demonstraram claras mudanças temporais e individuais no microbioma intestinal e nos metabólitos com abundância reduzida de Lachnospiraceae e enriquecimento de ácidos graxos insaturados de cadeia longa. A fermentação ex vivo com um meio semelhante ao EEN e a subsequente transferência em modelos de ratos gnotobióticos mostrou um efeito protetor em contraste com os meios ricos em fibras e com os colonizados diretamente com a microbiota de base do doente. Com base nestes resultados, concluíram que os microbiomas dos doentes modulados por EEN estão a regular a inflamação intestinal. Também se debruçaram sobre a possibilidade de utilizar uma dieta pobre em fibras para uma remissão a longo prazo. Um estudo multicêntrico realizado no Reino Unido (crianças e adultos) estudou a possibilidade de utilizar uma dieta de alimentos sólidos para a Doença de Crohn TReatment-with-EATing (CD-TREAT) para criar uma dieta mais palatável que poderia influenciar a inflamação intestinal através da alteração das bactérias intestinais [8]. A dieta é personalizada para cada doente, mas exclui componentes alimentares específicos como o glúten, a lactose e o álcool. Os 55% dos doentes que aderiram a este regime apresentaram uma calprotectina fecal significativamente mais baixa e tiveram alterações microbianas e metabólicas na mesma linha que os doentes submetidos a uma EEN bem-sucedida. Este facto não foi observado nos doentes que não respeitaram a dieta. Com base nesses achados, poderia ser interessante usar fezes autólogas de pacientes com DC tratados com sucesso com EEN para FMT. O grupo de Schwerd analisou essa possibilidade usando FMT de cápsulas autólogas. Concluíram que esta abordagem não era adequada, uma vez que ainda existia uma carga patogénica demasiado elevada e uma diversidade microbiota demasiado baixa [9]. O grupo de Colónia apresentou um poster muito interessante sobre o acompanhamento de 2 casos de DII muito precoce refratária ao tratamento com esteroides e anti-TNFα. O primeiro doente tem colite ulcerosa e está agora há 3 anos em remissão total com a administração semanal de um enema de preparação de fezes de um dador. O segundo doente com DC está apenas em remissão parcial após um ano de seguimento [10].
Este resumo apresenta apenas uma panorâmica breve e selecionada das palestras, comunicações e posters do ESPGHAN. Muitos outros dados interessantes são apresentados sob a forma de resumos no Journal of Pediatric Gastroenterology. Os esforços combinados de investigadores científicos e clínicos continuarão a desvendar o mistério da microbiota intestinal e acabarão por trazer novas formas de tratar e prevenir doenças.
1. Szajewska H, Berni Canani R, Domellöf M, et al. Probiotics for the Management of Pediatric Gastrointestinal Disorders: Position Paper of the ESPGHAN Special Interest Group on Gut Microbiota and Modifications. J Pediatr Gastroenterol Nutr 2023; 76: 232-47
2. De Bruijn C, Zeevenhoven J, Vlieger A, et al. Efficacy of fecal microbiota transplantation in adolescents with refractory irritable bowel: a randomized, double-blind, placebo-controlled trial. J Pediatr Gastroenterol Nutr 2023; 76(S1 Suppl 1): 1-1407
3. Wang YZ, Xiao FF, Xiao YM, et al. Fecal microbiota transplantation relieves abdominal bloating in children with functional gastrointestinal disorders via modulating the gut microbiome and metabolome. J Dig Dis 2022; 23: 482-92
4. El-Salhy M, Winkel R, Casen C, et al. Efficacy of Fecal Microbiota Transplantation for Patients with Irritable Bowel Syndrome at 3 Years After Transplantation. Gastroenterology 2022; 163: 982-94.e14
5. Mahler T, Hoffman I, Smets F, et al. The Belgian consensus on irritable bowel syndrome: the paediatric gastroenterologist view. Acta Gastroenterol Belg 2022; 85: 384-6
6. Hurych J, Mascellani Bergo A, Lerchova T, et al. The faecal microbiome and metabolome changes in Crohn’s disease are associated with decreased mucosal inflammatory activity. J Pediatr Gastroenterol Nutr 2023; 76 (S1 Suppl 1): 1-1407
7. Schwerd S, Häcker D, Siebert K, et al. Exclusive enteral nutrition initiates protective functions in the gut microbiota and metabolome to induce remission in pediatric Crohn’s disease. J Pediatr Gastroenterol Nutr 2023; 76 (S1 Suppl 1): 1-1407
8. Macdonald J, Wilson D, Henderson P, Din S, e Chantges in faecal microbiome and metabolome are more pronounced in Crohn’s disease patients who adhered to the CD-TREAT diet and responded by calprotectin. J Pediatr Gastroenterol Nutr 2023; 76 (S1 Suppl 1): 1-1407
9. Hölz H, Heetmeyer J, Tsakmaklis A, et al . Autologous fecal microbiota transfer in pediatric Crohn ́s disease patients under treatment with exclusive enteral nutrition harbors major challenges - a feasibility test. J Pediatr Gastroenterol Nutr 2023; 76 (S1 Suppl 1): 1-1407
10. Fritz T, Huenseler C, Broekaert I. Safety and efficacy of long-term faecal microbiota transfer in very early onset inflammatory bowel disease. J Pediatr Gastroenterol Nutr 2023; 76 (S1 Suppl 1): 1-1407
A colestase altera o desenvolvimento da microbiota intestinal e a atividade da hidrolase dos ácidos biliares em recém-nascidos prematuros
ARTIGO COMENTADO - Rubrica pediátrica
Pelo Prof. Emmanuel Mas
Gastroenterologia e Nutrição, Hospital Saint-Antoine, Paris, França

Comentário ao artigo original de Lynch LE et al (Gut Microbes) [1]
A colestase é uma perturbação do fluxo de bílis do fígado para o intestino. Nos recém-nascidos, a colestase leva a um crescimento deficiente e pode progredir para insuficiência hepática e morte. O fluxo normal de bílis requer um eixo fígado-intestino-microbiota intacto, no qual os ácidos biliares primários derivados do fígado são transformados em ácidos biliares secundários. As enzimas microbianas (hidrolase dos ácidos biliares [BSH]) são responsáveis pelo primeiro passo, que consiste na desconjugação dos ácidos biliares primários conjugados com glicina e taurina. Os recém-nascidos colestáticos são frequentemente tratados com ácido ursodesoxicólico (UDCA), um potente ácido biliar colerético, embora as interações entre o UDCA, as bactérias intestinais e outros ácidos biliares sejam pouco conhecidas. Este estudo, baseado em 124 amostras de fezes de 24 recém-nascidos, revelou novas associações que ligam os ácidos biliares isoméricos e a atividade da BSH às trajetórias de crescimento neonatal. Estes dados realçam o facto de a desconjugação dos ácidos biliares ser uma função microbiana essencial, adquirida precocemente no desenvolvimento neonatal e prejudicada pela colestase.
O que é que já sabemos sobre isto?
Os recém-nascidos prematuros, nascidos antes das 37 semanas de amenorreia (SA), correm um maior risco de desenvolver colestase. A colestase, definida como uma redução do fluxo biliar, é favorecida por vários fatores de risco, como a prematuridade, o baixo peso à nascença e a nutrição parentérica. Na ausência de uma etiologia, a condição é conhecida como colestase neonatal transitória. Para melhorar a colestase, é frequentemente efetuado um tratamento com ácido ursodeoxicólico (UDCA). Os ácidos biliares são necessários para a absorção dos lípidos e das vitaminas lipossolúveis. Durante a colestase, verifica-se uma redução da quantidade de ácidos biliares no intestino e uma alteração das proporções dos diferentes ácidos biliares. Os ácidos biliares primários são produzidos a partir do colesterol e conjugados no fígado. Interagem extensivamente com a microbiota intestinal, levando à formação de ácidos biliares secundários através de uma enzima microbiana, a hidrolase dos ácidos biliares (BSH).
Os ácidos biliares e a microbiota intestinal têm um impacto no crescimento e desenvolvimento dos bebés prematuros. Os autores quiseram estudar o impacto da colestase no estabelecimento da microbiota intestinal em bebés muito prematuros e na desconjugação dos ácidos biliares.
Quais são as principais conclusões deste estudo?
Os autores incluíram 24 recém-nascidos pré-termo, 12 colestáticos e 12 controlos, nascidos com 27,2± 1,8 SA, com um peso médio à nascença de 946± 249,6 g. O pico médio de bilirrubina conjugada foi de 7,0 mg/dL. Não se registaram diferenças entre os dois grupos em termos de ambiente intrauterino, modo de parto ou utilização de antibióticos ao longo do tempo.
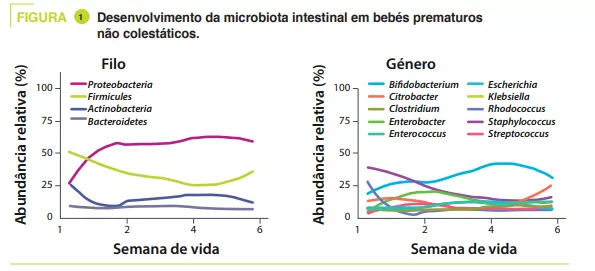
As fezes foram recolhidas desde o nascimento até às seis semanas de idade. A sequenciação (método shotgun) mostrou que a diversidade alfa aumentou nos controlos durante o primeiro mês de vida. Em termos de filos, Proteobacteria e Firmicutes foram os mais abundantes; em termos de géneros, Staphylococcus foi o mais abundante à nascença e depois diminuiu, enquanto a abundância de Klebsiella aumentou gradualmente (figura 1). Clostridium perfringens foi a espécie cuja abundância relativa mais aumentou ao longo do tempo, o que foi definido pela idade pós-menstrual (PMA, soma da idade a termo (SA) e idade pós-natal) (p = 0,01). A análise metagenómica mostrou que a via metabólica mais enriquecida nas fezes maduras (32-40 semanas PMA) em comparação com as fezes menos maduras (25-28 semanas PMA) foi a da biossíntese de ácidos biliares secundários.
Nos controlos, a análise de componentes principais mostrou que o principal fator que influenciava a composição da microbiota intestinal era a PMA, ao passo que não tinha qualquer efeito nos bebés prematuros colestáticos. A via biossintética secundária dos ácidos biliares foi mais enriquecida nas fezes dos controlos do que nos prematuros colestáticos às 32-40 semanas de PMA (p = 0,04). Da mesma forma, os autores encontraram uma diminuição de 55% na abundância relativa do gene BSH (p = 0,04) e Clostridium perfringens (p = 0,0008) em recém-nascidos colestáticos (figura 2).
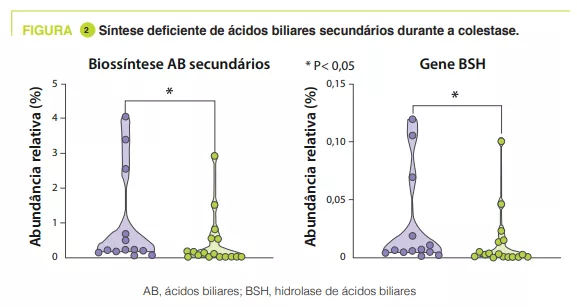
O perfil de ácidos biliares fecais medido por espetrometria de massa mostrou que a proporção de ácidos biliares não conjugados aumentou de 4% às 25-28 semanas PMA para 98% às 32-40 semanas PMA nos controlos, mas para apenas 46% nos doentes colestáticos. É de notar que certos isómeros podem ter um valor preditivo porque aumentam antes do início da colestase.
O UDCA, utilizado em cinco dos 12 bebés prematuros, foi encontrado nas suas fezes numa concentração 522 vezes superior à dos outros sete bebés que não foram tratados. O UDCA modificou a microbiota intestinal com um aumento relativo das Firmicutes e uma diminuição das Proteobacteria (p < 0,05), e ao nível das espécies com um enriquecimento do Clostridium perfringens.
Finalmente, os bebés pré-termo com uma abundância fecal do gene BSH > 0,005% às 32-40 semanas PMA tiveram um aumento de 1,2 vezes nas taxas de crescimento da estatura e de peso em comparação com aqueles com uma abundância < 0,005%. Da mesma forma, uma quantidade de ácido cólico fecal > 30% mostrou um aumento médio nas taxas de crescimento em altura (14%), peso (18%) e perímetro cefálico (15,8%) (p < 0,05).
Quais são as consequências práticas ?
Este estudo permite uma melhor compreensão dos mecanismos fisiopatológicos envolvidos na colestase na desregulação do eixo fígado-intestino-microbiota intestinal. Isto poderia levar à possibilidade de corrigir o ciclo entero-hepático utilizando certos probióticos ou outros medicamentos (com base na atividade do BSH) ou outros medicamentos.
CONCLUSÃO
Nos recém-nascidos extremamente prematuros, a colestase altera o desenvolvimento da microbiota intestinal, reduzindo a aquisição de Clostridium perfringens e a capacidade de síntese de ácidos biliares secundários. Inversamente, um aumento de certos ácidos biliares, ligado à atividade da BSH, está associado a um melhor crescimento neonatal.
Microbiota: essencial para a saúde, mas com pouca sensibilização a nível mundial
O que é que os nossos concidadãos sabem atualmente sobre o papel do microbiota? O que é que eles sabem sobre como cuidar do seu microbiota? Que papel desempenham os profissionais de saúde na informação aos pacientes? Para responder a estas questões, o Instituto Biocodex do Microbiota encomendou à Ipsos um grande inquérito internacional sobre este tema: o Observatório Internacional do Microbiota. Para levar a cabo este inquérito, a Ipsos questionou 6.500 pessoas em 7 países1 . O inquérito foi realizado de 21 de março a 7 de abril de 2023. Os resultados foram apresentados a 27 de junho, Dia Mundial do Microbioma.
Muito pouco conhecimento da palavra “microbiota”
Regra geral, o conhecimento do microbiota é bastante reduzido: apenas 1 em cada 5 pessoas afirma saber o significado exato do termo microbiota (21%), enquanto as restantes admitem conhecer o termo apenas de nome (43%). Mais de 1 em cada 3 pessoas diz nunca ter ouvido falar da palavra (36%). Além disso, quando aprofundamos o seu nível de conhecimento, as noções são superficiais. Enquanto uma pequena maioria afirma conhecer a microbiota intestinal (53%, mas apenas 24% sabem exatamente o que é), outros tipos de microbiota são muito menos conhecidos: quer se trate da microbiota vaginal (45% dos entrevistados conhecem o termo, mas apenas 18% sabem exatamente o que é), da microbiota oral (43% conhecem o nome, mas 17% sabem o que é) ou da microbiota da pele (40% conhecem o termo, mas apenas 15% sabem o que é). Outros são ainda menos conhecidos, como o microbiota urinário (apenas 14% sabem exatamente o que é), o microbiota pulmonar (13% sabem exatamente o que é) e o microbiota otorrinolaringológico (11% sabem exatamente o que é).
E um conhecimento relativamente fraco do papel e da importância do microbiota
Cerca de 3 em cada 4 entrevistados estão conscientes de que um risco de desequilíbrio do microbiota pode ter consequências importantes para a saúde (75%), que a nossa alimentação tem consequências importantes no equilíbrio do nosso microbiota (74%) e que o nosso microbiota desempenha um papel real nos mecanismos de defesa imunitária (72%). Quanto ao resto, o conhecimento continua a ser muito moderado. Mais de 1 em cada 3 pessoas não sabe que os antibióticos têm um impacto no nosso microbiota (34%). Quase 1 em cada 2 pessoas não sabe que o microbiota é constituído por bactérias, fungos e vírus (46%) e que permite ao intestino transmitir informações essenciais sobre a saúde ao cérebro (47%). 1 em cada 2 pessoas pensa que, quando o nosso microbiota está desequilibrado ou não funciona corretamente, não há muito que possamos fazer (47%). Por último, a grande maioria dos inquiridos não sabia que muitas doenças, como Parkinson, Alzheimer e autismo, podiam estar ligadas ao microbiota (75%).
1 em cada 5 pessoas afirma saber exatamente o que significa a palavra microbiota (21%)
24% sabem exatamente o que é
1 em cada 3 pessoas foi informada de que a toma de antibióticos poderia ter um impacto negativo no equilíbrio do seu microbiota
O início da consciência?
Atualmente, mais de 1 em cada 2 pessoas afirma ter adotado comportamentos na sua vida quotidiana para manter o equilíbrio do seu microbiota (57%). Esta nova consciência é de aplaudir, mas também deve ser relativizada. Em primeiro lugar, porque apenas 1 em cada 7 pessoas diz que o faz “muito” (15%), enquanto a maioria das outras diz que o faz apenas “um pouco” (42%). Em segundo lugar, 43% dos inquiridos afirmaram não ter adotado qualquer comportamento específico. Os resultados do Observatório Internacional do Microbiota mostram que ainda há muito a fazer neste domínio.
Informações fornecidas pelos profissionais de saúde: um vetor de informação revolucionário! qui change la donne !
Menos de 1 em cada 2 doentes afirma que o seu médico lhes explicou alguma vez como manter um microbiota equilibrado (44%, mas apenas 19% o fizeram mais do que uma vez), ou prescreveu probióticos ou prebióticos (46%, mas apenas 21% afirmam tê-lo feito várias vezes). Apenas uma minoria dos inquiridos afirma ter sido sensibilizada pelo seu médico para a importância de ter um microbiota equilibrado (42%). Por último, apenas 1 em cada 3 afirma que o seu médico lhe ensinou o que era o microbiota e para que servia (37%, e apenas 15% afirmam ter sido informados várias vezes).
A informação fornecida pelos médicos aquando da prescrição de antibióticos ilustra como ainda é insuficiente a sensibilização dos doentes para os riscos do tratamento em termos de desequilíbrio do microbiota. A prescrição de antibióticos deveria ser uma oportunidade para fornecer informações essenciais sobre o microbiota, mas em muitos casos não o é. Quando se prescrevem antibióticos, por exemplo, o microbiota do doente está em risco. Quando são prescritos antibióticos, menos de 1 em cada 2 doentes afirma que o seu médico os informou do risco de problemas digestivos associados aos antibióticos (41%). Apenas 1 em cada 3 recebeu conselhos sobre como minimizar as consequências negativas da toma de antibióticos no seu microbiota (34%) ou foi informado de que a toma de antibióticos poderia ter consequências negativas no equilíbrio do seu microbiota (33%).
O que o inquérito mostra é que, depois de um doente ter recebido toda a informação sobre o assunto, e repetidamente, a sua relação com a microbiota muda significativamente e destaca-se da média. Mais de 9 em cada 10 pessoas (95%) que receberam informações repetidas do seu profissional de saúde adoptaram comportamentos para manter o seu microbiota equilibrado, contra 57% do conjunto dos inquiridos. A informação repetida de um profissional de saúde tem, portanto, um impacto muito forte nos níveis de conhecimento e nos comportamentos.
Este inquérito exclusivo revela tanto uma falta de compreensão geral sobre a forma como o microbiota afecta a nossa saúde como o papel essencial dos profissionais de saúde na transmissão de conhecimentos.
1 em cada 2 doentes afirma que o seu médico já lhe explicou como manter uma microbiota equilibrada
95% das pessoas que receberam informações repetidas do seu profissional de saúde adoptaram comportamentos conducentes à manutenção de um microbiota equilibrado
contra 57% de todos das pessoas inquiridas
O ácido indole-3-acético (3-IAA) derivado da microbiota intestinal influencia a eficácia da quimioterapia no cancro do pâncreas
ARTIGO COMENTADO - Fase adulta
Pelo Prof. Harry Sokol
Gastroenterologia e Nutrição, Hospital Saint-Antoine, Paris, França
Comentário ao artigo de Tintelnot et al. Nature 2023 [1]
Prevê-se que o adenocarcinoma ductal pancreático (PDAC) seja o segundo cancro mais mortífero até 2040, devido à elevada incidência de doença metastática e à resposta limitada ao tratamento. Menos de metade dos doentes responde à quimioterapia, o principal tratamento para o PDAC, e as variações genéticas dos doentes não são suficientes para explicar estas diferenças de resposta. A dieta é um dos fatores ambientais que pode influenciar a resposta à terapia, mas o seu papel no PDAC não é claro. Aqui, utilizando a sequenciação metagenómica e o rastreio metabolómico, os autores demonstraram que o ácido indol-3-acético (3-IAA), um metabolito do triptofano derivado da microbiota, é enriquecido nos doentes que respondem ao tratamento. O transplante de microbiota fecal, as intervenções dietéticas com triptofano e a administração oral de 3-IAA aumentam a eficácia da quimioterapia em modelos de ratinhos humanizados de PDAC. Utilizando uma combinação de experiências de perda de função e de ganho de função, os autores mostraram que a eficácia do 3-IAA e da quimioterapia requer a presença de mieloperoxidase (MPO) derivada de neutrófilos. A MPO oxida o 3-IAA que, em combinação com a quimioterapia, regula em baixa as enzimas que degradam as espécies reativas de oxigénio (ROS), a glutationa peroxidase 3 e a glutationa peroxidase 7. Tudo isto leva a uma acumulação de ROS e a uma desregulação da autofagia nas células cancerígenas, comprometendo a sua capacidade metabólica e, em última análise, a sua proliferação. Em humanos, os autores observaram uma correlação significativa entre os níveis de 3-IAA e a eficácia da terapia em duas coortes independentes de PDAC. Em resumo, os autores identificaram um metabolito derivado da microbiota que tem implicações clínicas no tratamento do PDAC, e fornecem motivação para considerar intervenções nutricionais durante o tratamento de pacientes com cancro.
O que é que já sabemos sobre isto?
A terapêutica com múltiplos fármacos, quer com 5-fluorouracilo (5-FU), irinotecano e oxaliplatina em combinação com ácido folínico (FOLFIRINOX), quer com gemcitabina e nabpaclitaxel (GnP), é considerada o padrão de tratamento para doentes com PDAC metastático (mPDAC). No entanto, menos de metade dos doentes respondem à terapêutica e os doentes que não respondem (não respondedores, NRs) morrem em poucas semanas em sofrimento doloroso. As alterações genéticas no PDAC pouco explicam as diferenças entre os doentes que respondem ao tratamento (doentes respondedores (R)) e os doentes NR, deixando os fatores ambientais, incluindo a microbiota intestinal, como potenciais mediadores da eficácia da quimioterapia. Por conseguinte, é urgente identificar os fatores ambientais que poderiam explicar as diferenças entre os doentes R e NR, a fim de desenvolver novos conceitos para futuras terapias. Foi demonstrado que a microbiota intestinal está envolvida na resposta à imunoterapia em doentes com melanoma e muitos outros cancros e pode ser modulado pela dieta [2, 3]. Em doentes raros com PDAC localizado que sobrevivem a longo prazo, as bactérias podem passar do intestino para o tumor e controlar a ativação imunitária antitumoral. No entanto, a maior parte dos doentes com PDAC agressivo e resistente à imunoterapia são tratados com poliquimioterapia e ainda não é claro se e como a microbiota ou os hábitos alimentares influenciam a sua eficácia [4].
- A microbiota intestinal é diferente em doentes com PDAC que respondem e não respondem à quimioterapia
- O metabolito triptofano 3-IAA, produzido pela microbiota, é enriquecido em doentes que responderam
- O 3-IAA aumenta a eficácia da quimioterapia através do aumento da produção de ROS pelos neutrófilos e da diminuição da autofagia nas células cancerígenas
Quais são as principais conclusões deste estudo?
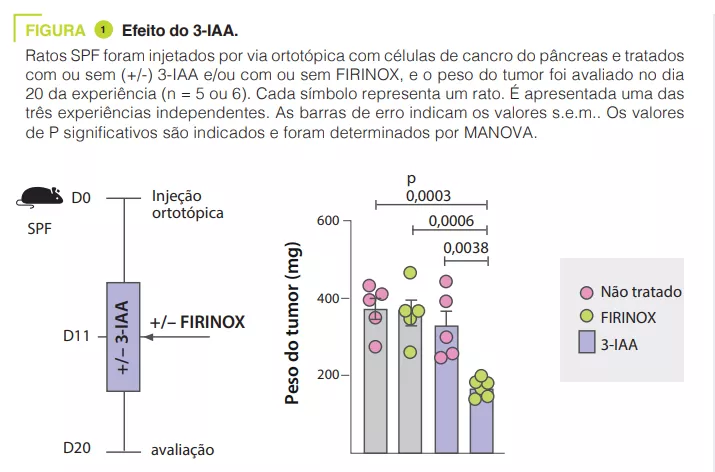
A análise da microbiota de 30 doentes com mPDAC revelou diferenças entre os doentes R e NR. A transferência da microbiota dos doentes R para ratinhos com tumores pancreáticos reduziu o tamanho dos tumores após a quimioterapia. O metabolito triptofano 3-IAA foi enriquecido tanto nos doentes R como nos ratinhos com microbiota R, contribuindo potencialmente para a resposta à quimioterapia. A administração de 3-IAA melhorou a eficácia da quimioterapia em ratinhos (figura 1). A análise das células imunitárias nos ratinhos revelou um aumento das células T CD8+ e uma diminuição dos neutrófilos nos ratinhos com um microbiota associado a uma boa resposta à quimioterapia. O 3-IAA afetou a MPO dos neutrófilos, reduzindo assim a sua sobrevivência. A combinação de 3-IAA e quimioterapia reduziu o número de neutrófilos e inibiu o crescimento do tumor, com a MPO a desempenhar um papel crucial. Foi sugerido que a MPO induz a produção de ROS, levando à morte celular durante a quimioterapia. Experiências in vitro mostraram que o 3-IAA aumentou os níveis de ROS (espécies reativas de oxigénio). Este efeito foi confirmado in vivo e a inibição das ROS pela N-acetilcisteína eliminou a eficácia do FIRINOX em ratinhos com níveis elevados de 3-IAA. Os autores mostraram então que o efeito do 3-IAA estava ligado a uma regulação negativa da autofagia. Por fim, concentrações séricas elevadas de 3-IAA foram correlacionadas com uma redução da contagem de neutrófilos e uma melhoria da sobrevivência em duas coortes de doentes humanos.
Quais são as consequências práticas?
A microbiota intestinal tem um efeito sobre a resposta à quimioterapia. Entre os mecanismos envolvidos, este estudo demonstra o papel dos metabolitos microbianos, particularmente os metabolitos do triptofano. Entre estes, o 3-IAA não só é um marcador preditivo da resposta à quimioterapia no PDAC, como também pode representar uma molécula terapêutica adjuvante.
CONCLUSÃO
A microbiota intestinal tem um efeito sobre a resposta à quimioterapia no PDAC. Um dos seus metabolitos, o 3-IAA, é preditivo de uma boa resposta à quimioterapia e aumenta o seu efeito induzindo a acumulação de ROS e reduzindo a autofagia das células cancerígenas.
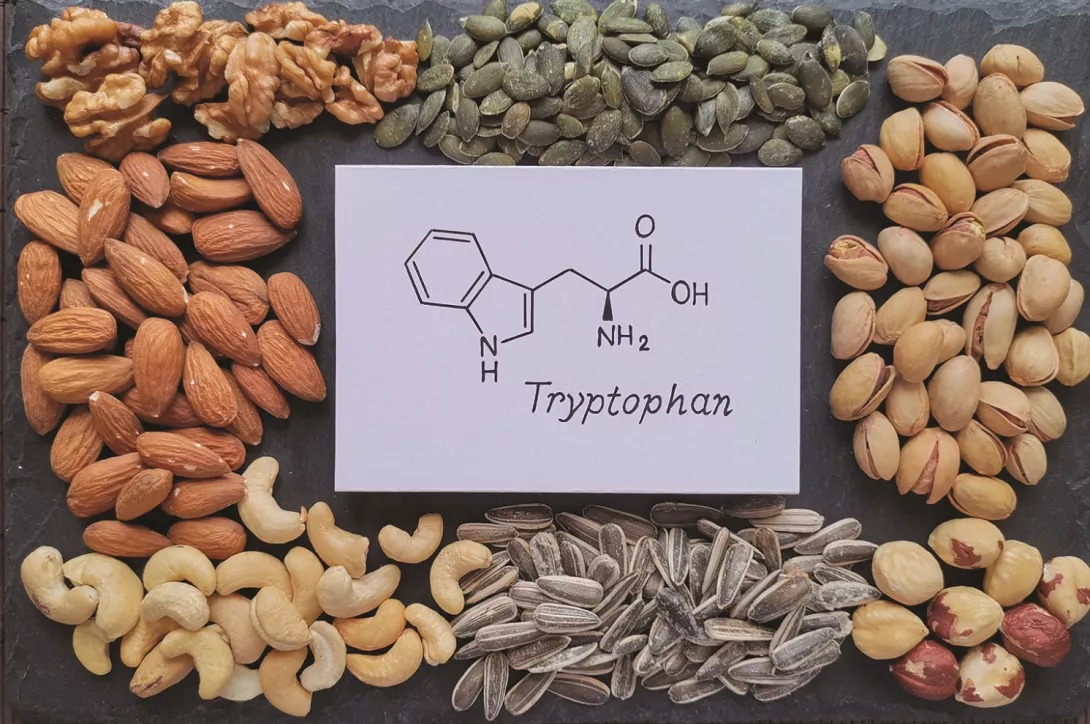
1.Tintelnot J, Xu Y, Lesker TR, et al. Microbiota-derived 3-IAA influences chemotherapy efficacy in pancreatic cancer. Nature 2023 ; 615 : 168-74.
2. Baruch EN, Youngster I, Ben-Betzalel G, et al. Fecal microbiota transplant promotes response in immunotherapy-refractory melanoma patients. Science 2021 ; 371 : 602-9.
3. Spencer CN, McQuade JL, Gopalakrishnan V, et al. Dietary fiber and probiotics influence the gut microbiome and melanoma immunotherapy response. Science 2021 ; 374 : 1632-40.
4. Thomas RM, Jobin, C. Microbiota in pancreatic health and disease: the next frontier in microbiome research. Nat Rev Gastroenterol Hepatol 2020 ; 17 : 53-64.
Microbioma intestinal e resistência antimicrobiana
Pelo Dr. Joseph Nesme
Professor Assistente, Microbiologia, Departamento de Biologia,Universidade de Copenhaga, Dinamarca
A resistência aos antibióticos é antiga e prevalecente e o resistoma ambiental é um reservatório de genes de resistência que alimenta a pandemia silenciosa de resistência antimicrobiana (RA). A transferência horizontal de genes, a dinâmica populacional e a co-seleção aceleram o aparecimento e a disseminação do resistoma e, em 2050, mais de 10 milhões de mortes por ano dever-se-ão à resistência aos antibióticos. A colonização precoce do microbioma intestinal é propensa a disbioses que podem levar a um aumento da abundância e diversidade do resistoma intestinal dos bebés. A adoção de uma abordagem “Uma Só Saúde”, que considere a interligação entre a saúde humana, a saúde animal e o ambiente, é essencial para enfrentar os desafios complexos colocados pela RA.
Resistência aos antibióticos no ambiente: Um desafio pré-existente
A resistência aos antibióticos (RA) é um fenómeno antigo e prevalecente no ambiente, existente muito antes da introdução de moléculas de antibióticos como terapêutica. O ambiente funciona como um vasto reservatório de genes de resistência aos antibióticos, com diversas comunidades microbianas que albergam mecanismos de resistência. A RA foi encontrada em vários contextos ambientais, incluindo o solo, a água, as plantas, os animais e até mesmo no permafrost ártico com 30 000 anos [1, 2]. O papel ecológico das moléculas antibióticas e da resistência associada em contextos não clínicos permanece pouco claro, mas realça o facto de um conjunto de genes prontamente disponíveis ser anterior à utilização clínica de antibióticos e explicar a rápida emergência de agentes patogénicos. A atual crise dos antibióticos é um fenómeno evolutivo e as estratégias de atenuação devem ter em conta a ecologia microbiana. O que é problemático é a rápida aquisição de resistência por parte de agentes patogénicos anteriormente sensíveis, que conduz a falhas terapêuticas, especialmente quando se espera que muito poucos novos antimicrobianos cheguem ao mercado [3].
Mecanismos que determinam o aparecimento e a disseminação do resistoma
O resistoma refere-se ao conjunto completo de genes que codificam as proteínas relacionadas com a resistência aos antibióticos (RA) ou as proteínas que podem potencialmente evoluir para poderosos agentes de RA [4] (figura 1). Isto inclui genes de RA reconhecidos em bactérias patogénicas (as problemáticas), genes de RA de organismos produtores de antibióticos, como Streptomyces spp. que produzem o antibiótico estreptomicina e o gene de resistência associado [5], genes de RA crípticos (ou seja, genes que poderiam proporcionar resistência num contexto genético diferente; por exemplo, bombas de efluxo reguladas positivamente ou porinas reguladas negativamente) e genes de RA precursores que codificam proteínas com um nível mínimo de afinidade ou resistência a compostos antibióticos.
É de salientar que uma parte considerável destes subconjuntos de genes se sobrepõe devido à homologia das sequências, indicando que provavelmente têm um passado evolutivo partilhado.
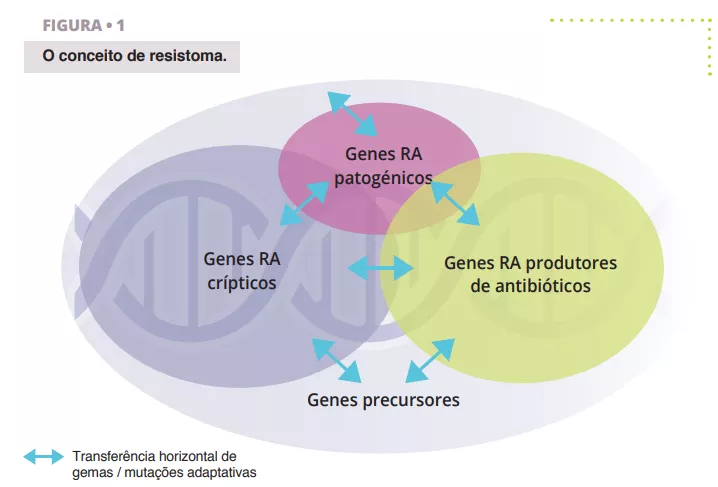
A noção de resistoma é distinta de “RA funcional e clinicamente relevante”. De facto, os genes do resistoma podem transitar entre os diferentes estados descritos acima por transferência horizontal de genes (HGT), mutações pontuais ou recombinação que conduzem a novos hospedeiros ou contextos genéticos onde o fenótipo de RA clinicamente relevante pode ser expresso. Por conseguinte, um gene de resistência não é problemático por si só, uma vez que depende do hospedeiro e do contexto genético, mas todos os genes do resistoma são uma ameaça potencial com diferentes resultados de risco para a saúde pública associados. A descoberta de um gene de resistência a uma molécula clinicamente relevante, localizado num elemento móvel e alojado num agente patogénico humano, constitui um risco crítico, mas o mesmo gene, ou homólogos próximos, encontrado numa bactéria não patogénica do solo e não associado a um elemento genético móvel, constitui um ARG (Gene de Resistência Antimicrobiana) de risco muito baixo. A classificação do risco de resistência aos antibióticos em estudos de resistoma é, por conseguinte, de importância primordial.
A transferência horizontal de genes (HGT) é um mecanismo fundamental responsável pela rápida disseminação de genes AR entre bactérias, mesmo entre linhagens distantes. Por exemplo, Bacteroides spp., um grupo predominante na microbiota intestinal humana, possui um gene de resistência aos macrólidos ermB idêntico ao encontrado em vários isolados de Clostridium perfringens, Streptococcus pneumoniae e Enterococcus faecalis de várias origens geográficas, indicando uma ligação genética entre Bacteroides e algumas bactérias Gram-positivas que não são predominantes no intestino humano [6]. Os elementos genéticos, como os plasmídeos, facilitam a transferência de genes de resistência entre diferentes espécies microbianas [7]. A HGT permite a disseminação de genes em diversos ambientes e populações bacterianas, contribuindo para a prevalência e diversidade globais da RA. A co-seleção é outro fator significativo na propagação da RA. A utilização de compostos não antibióticos, como os metais pesados e os biocidas, pode co-selecionar genes de RA, exercendo pressões seletivas sobre as populações microbianas, quer por co-resistência (diferentes determinantes de resistência presentes no mesmo elemento genético), quer por resistência cruzada (o mesmo determinante genético responsável pela resistência aos antibióticos e aos metais) [8]. A exposição a compostos antimicrobianos naturais, tais como os produzidos por microrganismos concorrentes ou quaisquer compostos co-seletivos, pode conduzir à seleção de estirpes resistentes [9]. A presença de antibióticos no ambiente, quer de fontes naturais quer de atividades humanas, contribui ainda mais para a pressão de seleção da resistência. Além disso, a utilização de antibióticos na agricultura e nas práticas veterinárias pode levar à contaminação do ambiente, promovendo o aparecimento e a disseminação de genes de resistência ambiental aos antibióticos ao longo do tempo [10].
A compreensão das barreiras e tendências à transferência de vetores genéticos que albergam genes de resistência é crucial para conceber estratégias de mitigação eco-evolutivas informadas para limitar a disseminação da RA no início da vida e, em geral, em contextos clínicos.
Disbiose e o resistoma intestinal nos bebés: Um equilíbrio delicado
A diversidade do reservatório ambiental de genes AR e o seu potencial de transferência representam uma ameaça para o microbioma intestinal humano no início da vida. Estratégias como a melhoria do tratamento de águas residuais, a utilização responsável de antibióticos na agricultura e na medicina veterinária e a redução da contaminação ambiental com resíduos de antibióticos e bactérias resistentes a antibióticos podem ajudar a atenuar a propagação da resistência [11]. Além disso, a monitorização e a vigilância dos reservatórios ambientais podem fornecer informações valiosas sobre a emergência e a persistência da RA e informar as intervenções de saúde pública.
A adoção de uma abordagem “Uma Só Saúde”, que considere a interligação entre a saúde humana, a saúde animal e o ambiente, é essencial para enfrentar os desafios complexos colocados pela RA.
O nosso intestino é rapidamente colonizado após o nascimento por microrganismos adquiridos das suas mães e do ambiente que as rodeia. É durante os primeiros anos de vida que as mudanças são drásticas e caracterizadas por uma baixa resiliência em comparação com o microbioma intestinal do adulto saudável, que é mais estável. Os recém-nascidos e os bebés são, portanto, mais propensos a perturbações nas comunidades microbianas, conhecidas como disbiose. Durante esse período, muitos fatores podem influenciar e perturbar a maturação intestinal e, potencialmente, ter consequências a longo prazo para a saúde [12]. Estudos em ratos mostraram que, durante esta janela crítica de desenvolvimento, mais do que um efeito direto das moléculas de antibióticos, é a alteração da composição da microbiota intestinal que desencadeia consequências metabólicas, como a obesidade [13].
Desvendar o resistoma dos antibióticos no intestino dos bebés: Informações de um grande estudo de coorte
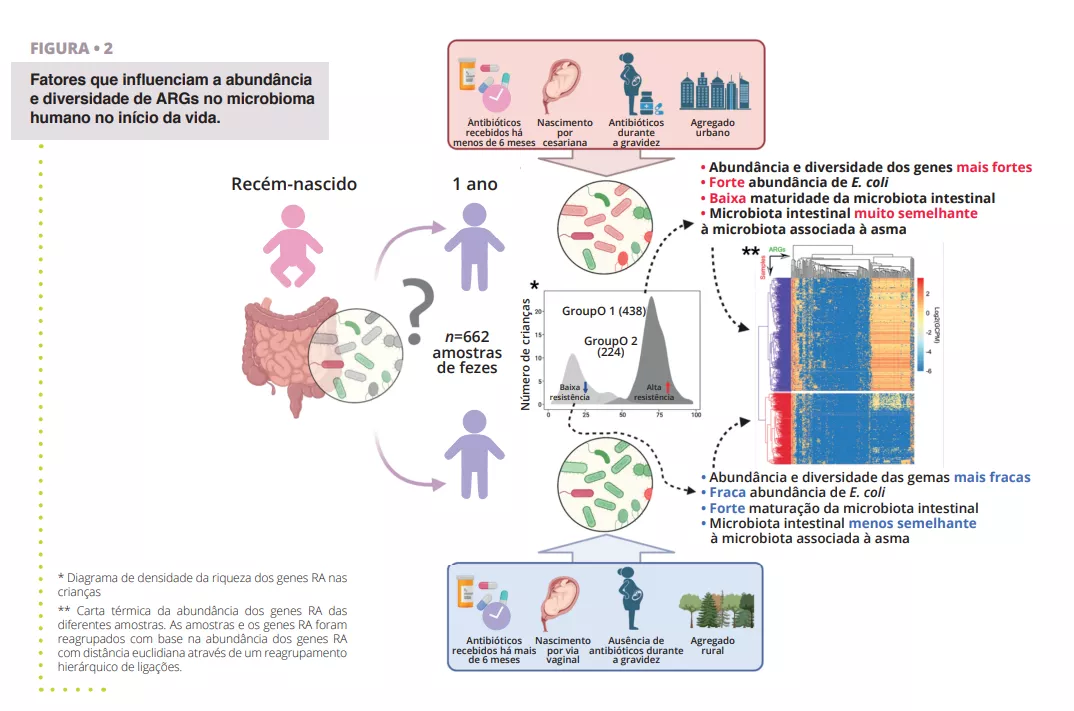
Embora a resistência aos antibióticos seja problemática em todas as idades da vida, o estabelecimento do microbioma intestinal em idades precoces representa uma janela de oportunidade para limitar a acumulação de um reservatório de genes AR no intestino. Por conseguinte, é importante identificar os vários fatores que aumentam ou reduzem a abundância de genes AR que podem propagar-se a agentes patogénicos infeciosos e causar falhas na terapia antibiótica ao longo da vida. Para estudar o resistoma global do intestino humano, os investigadores baseiam-se em abordagens holísticas que interrogam tanto a presença de espécies como o potencial funcional dos genomas, incluindo o resistoma aos antibióticos. Os investigadores baseiam-se na extração de ADN ambiental a partir de amostras de substituição (por exemplo, amostras de fezes para o intestino), seguida de sequenciação de alto rendimento não orientada (metagenómica shotgun). Cerca de 80% das espécies bacterianas do intestino humano detetadas por ferramentas moleculares não são cultiváveis, especialmente os anaeróbios especializados que habitam o intestino. É provável que muitos microrganismos estejam organizados em agregados celulares multi-espécies com co-dependências metabólicas que tornam o isolamento de estirpes puras delicado, se não impossível. No entanto, utilizando métodos computacionais, é possível reconstruir genomas quase completos a partir de metagenomas e associar genes codificados a espécies específicas ou mesmo a estirpes de bactérias. Num estudo recente, os investigadores analisaram amostras fecais de 662 bebés de uma coorte que acompanhou crianças desde o nascimento até aos 7 anos de idade [14]. Os objetivos do estudo eram estabelecer uma visão geral à escala da coorte do resistoma com 1 ano de idade e identificar fatores perinatais e ambientais associados à abundância e diversidade de ARG. Os investigadores utilizaram a sequenciação metagenómica shotgun de amostras obtidas com 1 ano de idade das 662 crianças para identificar ARGs e taxa bacterianos presentes nas amostras (figura 2).
Utilizando o seu grande conjunto de dados, os autores conseguiram reconstruir Genomas Associados a Metagenomas (MAGs), permitindo-lhes anotar com confiança a taxonomia dos genomas recuperados e o conteúdo dos seus genes AR. Um primeiro resultado observado foi que todas as crianças tinham pelo menos um tipo de ARG de múltiplos fármacos no seu intestino, indicando que, mesmo na ausência de tratamento com antibióticos, existe um resistoma residente associado ao microbioma intestinal. De facto, muitos genes de resistência a múltiplos fármacos foram identificados como bombas de efluxo. Estas proteínas são componentes normais de todas as células bacterianas, mas algumas podem conferir RA e são muito facilmente co-selecionadas por resistência cruzada, por exemplo, em relação a metais pesados ou biocidas, explicando potencialmente a sua elevada abundância no intestino, mas também em todos os ambientes [8, 15]. Outro resultado surpreendente foi a separação clara da coorte em dois grupos com base no seu perfil de resistoma. O primeiro grupo foi caracterizado por uma maior diversidade e abundância relativa de ARGs, com Escherichia coli como o principal contribuinte de ARGs, conforme mostrado na figura 2. Este facto está de acordo com observações anteriores de que as Enterobacteriaceae são abundantes no início da vida, mas devem diminuir rapidamente quando a população de Bacteroidetes começa a colonizar o intestino. A alteração desta maturação em algumas crianças pode estar associada a uma combinação de vários fatores, tais como a utilização de antibióticos, o modo de parto, o agregado familiar rural ou urbano, que parece atrasar a redução da população de Escherichia coli e levar a um aumento do resistoma com 1 ano de idade. Isto também é confirmado pela observação de que a maior abundância de ARGs está associada a uma menorpontuação de maturidade do microbioma intestinal, com base em rácios de taxa específicos relacionados com a idade [16].
O tratamento com antibióticos durante a gravidez e o primeiro ano de vida teve um impacto significativo na abundância do resistoma intestinal do bebé. Foi possível observar uma tendência temporal clara entre o tempo (mais de 6 meses ou menos) decorrido desde o último tratamento com antibióticos efetuado por uma criança e a abundância de ARGs no seu intestino.
Isto indica um certo nível de resiliência numa idade precoce que poderia ser potencialmente melhorado com uma intervenção direcionada, por exemplo, com pró- ou prebióticos, e que continua por testar. À escala da coorte, as maiores diferenças na abundância de ARG foram, no entanto, encontradas para genes de resistência a antibióticos que não foram prescritos às crianças, o que indica que os fatores perinatais e ambientais, para além da terapia antibiótica, também estão a determinar o resistoma intestinal. Outra observação desse estudo que liga o ambiente circundante, o resistoma associado e o resistoma intestinal foi o facto de as crianças cujos agregados familiares se situavam em zonas urbanas terem uma carga significativamente mais elevada de ARGs do que as crianças que viviam em zonas rurais. Há uma miríade de potenciais fatores de confusão que poderiam explicar este facto, mas reforça a ideia de que a contribuição do ambiente para o desenvolvimento do microbioma no início da vida é extremamente importante.
Pode colocar-se a hipótese de que a vida urbana está associada a um menor contacto com o exterior e a uma menor diversidade do microbioma, ou que o tipo de alojamento encontrado em ambiente rural (casa) ou urbano (apartamento) está associado a consequências no microbioma interior, tal como se observa no pó da cama.
Conclusão
A resistência aos antibióticos é antiga e prevalecente e o resistoma ambiental é um reservatório de potenciais genes de resistência clínica. Mecanismos como a transferência horizontal de genes, a intrincada dinâmica populacional e os efeitos de co-seleção foram identificados como aceleradores significativos da evolução e disseminação do resistoma aos antibióticos, tornando estirpes de agentes patogénicos anteriormente sensíveis agora resistentes à terapia antibiótica. O microbioma intestinal humano e a sua fase inicial de desenvolvimento são propensos à disbiose e à potencial invasão, seleção e co-seleção de bactérias portadoras de genes de resistência, o que pode ter consequências a longo prazo para a saúde. Não é claro até que ponto estas estão relacionadas com futuras terapias antibióticas falhadas. São necessários estudos multiómicos de longa duração e de alta resolução temporal para identificar, em primeiro lugar, padrões longitudinais de desenvolvimento do microbioma intestinal e, em segundo lugar, as vias mais prováveis de transferência de genes AR por HGT. Em conjunto, estes resultados fornecerão informações que permitirão conceber estratégias preventivas para minimizar o desenvolvimento de RA logo no início da vida e limitar a transferência para agentes patogénicos problemáticos. Este esforço é da maior importância para reduzir o peso da pandemia mundial de RAM e melhorar a saúde pública global, preservando a eficácia dos poucos antibióticos disponíveis para combater as doenças infeciosas.
1. Nesme J, Cécillon S, Delmont TO, et al. Large-scale metagenomic-based study of antibiotic resistance in the environment. Curr Biol 2014; 24: 1096-100.
2. D’Costa VM, King CE, Kalan L, et al. Antibiotic resistance is ancient. Nature 2011; 477: 457-61.
3. Renwick MJ, Brogan DM, Mossialos E. A systematic review and critical assessment of incentive strategies for discovery and development of novel antibiotics. J Antibiot 2016; 69: 73-88.
4. Wright GD. The antibiotic resistome: the nexus of chemical and genetic diversity. Nat Rev Microbiol 2007; 5: 175-86.
5. Benveniste R, Davies J. Aminoglycoside antibiotic-inactivating enzymes in actinomycetes similar to those present in clinical isolates of antibiotic-resistant bacteria. Proc Natl Acad Sci USA 1973; 70: 2276-80.
6. Shoemaker NB, Vlamakis H, Hayes K, et al. Evidence for extensive resistance gene transfer among Bacteroides spp. and among Bacteroides and other genera in the human colon. Appl Environ Microbiol 2001; 67: 561-8.
7. Martínez JL, Coque TM, Baquero F. Prioritizing risks of antibiotic resistance genes in all metagenomes. Nat Rev Microbiol 2015; 13: 396.
8. Baker-Austin C, Wright MS, Stepanauskas R. Co-selection of antibiotic and metal resistance. Trends Microbiol 2006; 14: 176-82.
9. Wencewicz TA. Crossroads of Antibiotic Resistance and Biosynthesis. J Mol Biol 2019; 431: 3370-99.
10. Knapp CW, Dolfing J, Ehlert PA, et al. Evidence of increasing antibiotic resistance gene abundances in archived soils since 1940. Environ Sci Technol 2010; 44: 580-7.
11. Wolters B, Hauschild K, Blau K, et al. Biosolids for safe land application: does wastewater treatment plant size matters when considering antibiotics, pollutants, microbiome, mobile genetic elements and associated resistance genes? Environ Microbiol 2022; 24: 1573-89.
12. Dogra S, Sakwinska O, Soh SE, et al. Rate of establishing the gut microbiota in infancy has consequences for future health. Gut Microbes 2015; 6: 321-5.
13. Cox LM, Yamanishi S, Sohn J, et al. Altering the intestinal microbiota during a critical developmental window has lasting metabolic consequences. Cell 2014; 158: 705-21.
14. Li X, Stokholm J, Brejnrod A, et al. The infant gut resistome associates with E. coli, environmental exposures, gut microbiome maturity, and asthma-associated bacterial composition. Cell Host Microbe 2021; 29: 975-87.e4.
15. Mulder I, Siemens J, Sentek V, et al. Quaternary ammonium compounds in soil: implications for antibiotic resistance development. Rev Environ Sci Bio/Technology 2018; 17: 159-85.
16. Subramanian S, Huq S, Yatsunenko T, et al. Persistent gut microbiota immaturity in malnourished Bangladeshi children. Nature 2014; 510: 417-21.






